Nat Methods:新泛基因组研究工具包PGR-TK,可在多种尺度上分析重复/临床相关复杂基因变异
时间:2023-07-12 17:34:30 热度:37.1℃ 作者:网络
导读
基因组学是了解物种生物特性、进化以及人类遗传疾病的基础。自人类基因组计划(Human Genome project)启动以来,有数百万人类基因组已被测序,短读长DNA测序的成本也大幅降低,但基因组中仍有一些基本信息有待挖掘。
泛基因组是一个物种内所有基因组信息的总和,与单个参考基因组相比涵盖了更多的遗传多样性,有助于群体研究。近年来,随着人类泛基因组的从头组装和CHM13端粒到端粒组装的结果发表,人们越发意识到泛基因组的重要性,构建泛基因组似乎已成为大多数动植物基因组研究的目标。但由于泛基因组的复杂性和多样性,当前需要更先进的工具来进行分析。
近日,美国基因检测公司GeneDx领衔的科研团队在Nature Methods发表了题为“Multiscale analysis of pangenomes enables improved representation of genomic diversity for repetitive and clinically relevant genes”的文章。研究团队开发了“PanGenome Research Tool Kit(PGR-TK)”软件包,可在多种尺度上分析复杂的泛基因组结构和单倍型变异。通过将PGR-TK中的图分解方法应用于二型主要组织相容性复合体(MHC-II)中,证明了人类泛基因组对分析复杂基因区域的重要性。综上,PGR-TK能够解析和可视化人类基因组中最复杂的区域。

文章发表在Nature Methods
主要研究内容
PGR-TK整体框架
PGR-TK的设计流程如图1所示,其由几个不同的组件组成,以促进快速的泛基因组分析。研究团队将前期为快速基因组组装而开发的计算技术和数据结构,应用于PGR-TK的泛基因组分析任务中,无需一次性建立一个完整的基因组图谱。PGR-TK还提供了构建索引序列数据库的工具,可从数据库中获取和查询目标序列,从而创建相应的泛基因组。
PGR-TK使用极小锚点在不同尺度上生成泛基因组图谱,不用进行较多的计算密集型序列比对,泛基因组图谱的生成步骤能够等效地考虑所有输入序列。此外,研究团队还开发了一种算法,将盘结的泛基因组图谱分解为更易于管理的units(主丛,Principal bundle),用户可以很容易地将线性基因组序列映射到units上。通过揭示单倍型之间重复和重排变异的对比,PGR-TK可以提供更直观的可视化。

图1. PGR-TK的结构和最小锚定图的构造。来源:Nature Methods
主丛分解
泛基因组图谱是分析种群重复结构变异的基础,PGR-TK提供了从一组同源序列生成局部泛基因组图谱(MAP-graph)的功能,其构建效率较高。传统的双序列比对方法很难比对具有复杂重复结构的多序列,但通过PGR-TK可轻松解决上述难题。为评估PGR-TK分析性能,研究团队使用其研究了AMY1A基因位点的重复结构,AMY1A具有不同数量的拷贝,由与基因周围重复相关的大规模结构变异引起。
为进行比较,研究团队从HPRC组装的结果(47个样本)中生成了两种不同尺度的AMY1A MAP-graph。结果显示,在不到3分钟的时间内,PRG-TK便可从索引序列数据中生成上述数据。除MAP-graph之外,研究团队还提供了分析MAP-graph的工具,其设计了一种算法可将图谱“线性化”为一组“主丛”。
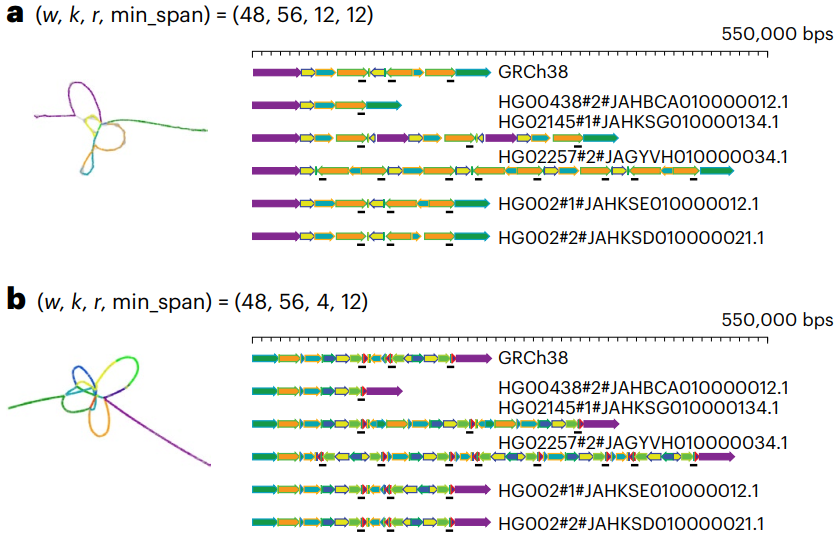
图2. AMY1A两种不同尺度的可视化。来源:Nature Methods
MHC II类基因座的泛基因组分析
人类基因组中的MHC区域具有高度多态性。MHC的基因组序列是了解人类适应性免疫系统和自身免疫性疾病的基础。
为展示PGR-TK在分析复杂的人类单倍型结构和序列方面的有效性,研究团队将其应用于HLA II类基因位点。结果显示,生成了一个由105个全长序列组成的集合,范围从650 kbp到800 kbp。图3a为该序列集合的主丛分解,图3b为105个序列的MAP-graph。PGR-TK可通过生成MHC II类分子的MAP-graph和主丛分解,来揭示单倍型变异的组合性质。
研究团队在主丛分解的基础上构建了一个层次树状图,以研究人类MHC区域高度多态性单倍型之间的关系。PGR-TK提供了一个命令行工具,用于计算两个序列之间派生的距离度量,并依据所有成对距离生成一个树状图。PGR-TK使用主丛来分类完整序列,而不是仅仅依赖基因片段,这种分类可能改进复杂地区更大人口数据中的基因分型或单倍型分型。在遗传变异和疾病易感性背景下,该研究结果为单倍型序列和基因组合之间的关系提供了有价值的见解。
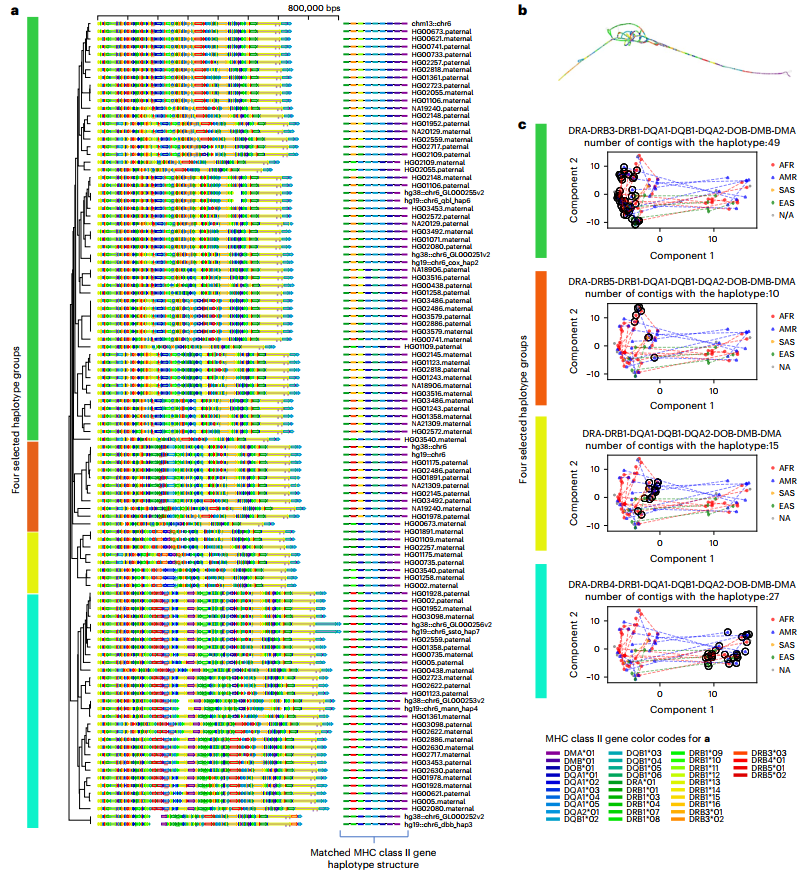
图3. 主丛分解揭示了不同的单倍型。来源:Nature Methods
分析医学相关的扩增子基因
已有研究对医学相关的扩增基因OPN1LW/OPN1MW/OPN1MW2/OPN1MW3和DAZ1/DAZ2/DAZ3/DAZ4进行了深入研究,但由于这些基因的变异过于复杂,目前的方法无法生成可与现有的基准测试工具兼容的变异调用。
OPN1MW和OPN1MW2基因相对于GRCh38有一个74 kb的缺失,因此HG002只包含GRCh38中4个该阵列拷贝中的2个。研究团队通过PGR-TK对上述基因进行了分析。结果显示,PGR-TK的可视化图清晰显示了每个单倍型中该阵列的基因数量变化。
另一个重要的基因家族DAZ1/DAZ2/DAZ3/DAZ4位于一组嵌套的回文重复序列中。据悉,该区域基因的部分缺失可能导致男性不育,因此解析非致病性结构变异在该扩增基因簇中的自然分布十分重要。DAZ基因包含缺失、插入和大的倒位等不同类型的结构变异。PGR-TK使用主丛分解算法在多个尺度上对DAZ基因中的变异进行识别和可视化,使人们能够直观地理解这种非常复杂的变异。
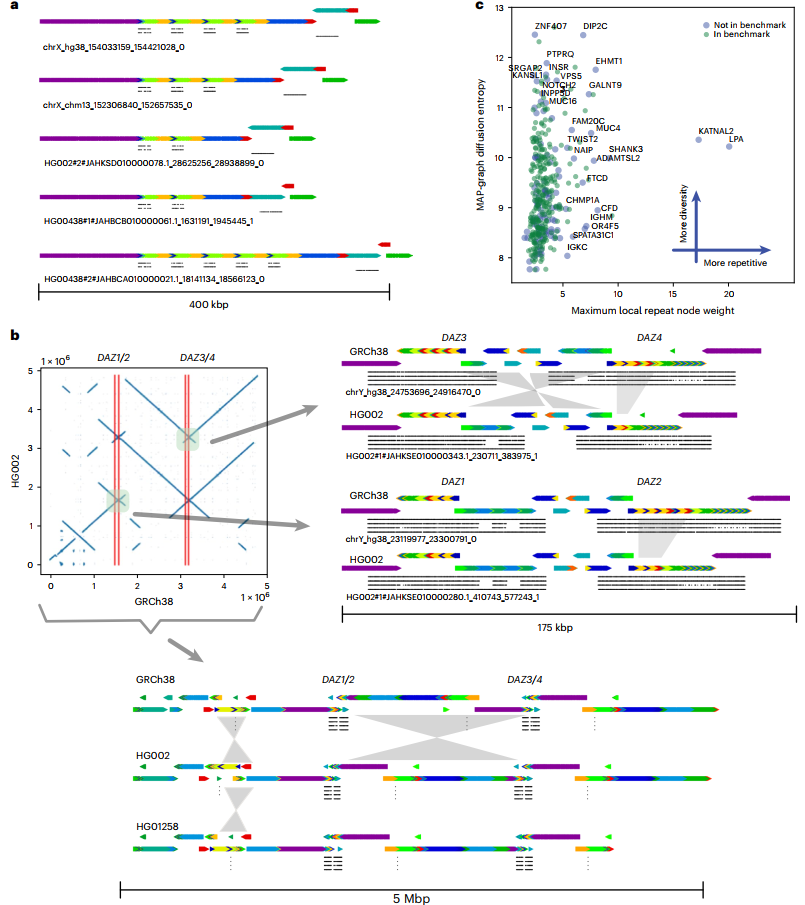
图4. 重复区域基因的主丛分解。来源:Nature Methods
结 语
综上所述,为有效解释许多人类基因组中各种尺度的变异,研究团队开发了PGR-TK,其能够可视化和更深入地分析重复基因的复杂变异,包括重复区域AMY1A和高度多态的HLA类II区域等。通过主丛分解,PGR-TK还可以自动可视化单倍型组合的重复和非重复组件,提供关于不同基因组排列体系结构的直观定性信息。在未来,研究团队希望扩展PGR-TK内置数据库,以利用泛基因组资源为基础来提供更多定量和基础水平的分析。
GeneDx公司首席技术官Gustavo Stolovitzky表示:“PGR-TK就像双筒望远镜一样,允许你调整焦距,看到不同尺度的特定结构。”
文章通讯作者兼第一作者Chen-Shan Chin说道:“目前已经有许多泛基因组工具包,它们专注于构建全基因组图谱,是计算密集型的。在很多情况下,我们只对某个地区感兴趣,我们的工具可以让你首先获取并专注于你感兴趣的几个区域,这种方法使其计算效率很高,原则上,可以允许研究人员同时分析一组基因组。”
参考文献:
1.Chin, CS., Behera, S., Khalak, A. et al. Multiscale analysis of pangenomes enables improved representation of genomic diversity for repetitive and clinically relevant genes. Nat Methods (2023).
2. Nurk, S. et al. The complete sequence of a human genome. Science 376, 44–53 (2022).
3. Eizenga, J. M. et al. Pangenome graphs. Annu. Rev. Genom. Hum. Genet. 21, 139–162 (2020).


