Genome Biol综述 | 多模态和单细胞/亚细胞水平空间组学技术的现状及发展前景
时间:2022-12-29 18:00:15 热度:37.1℃ 作者:网络
空间组学技术能够帮助人们更深入地了解细胞组织和目标组织内的相互作用。这些技术方法可以识别组织中具有差异转录物或蛋白质丰度的特定区段或区域,描述其相互作用,并补充定义细胞表型的其他方法。目前,各种空间方法正在开发和商业化,这些技术在空间分辨率、多路复用能力、规模和覆盖方面各有不同。
近日,美国威尔康奈尔医学院的研究团队回顾了从单细胞到亚细胞分辨率的空间组学技术和分析工具的现状及前景,为科研研究和临床应用提供了一个全面、详尽的综合分析。研究团队将分析结果发表Genome Biology上,文章题为“Spatial omics technologies at multimodal and single cell/subcellular level”。
 在原生空间背景下对单细胞进行分子分析的方法目前正迅速发展中。近年来,为覆盖更大数量的转录本或区域,传统的实验方法如条形码、免疫组化(IHC)和荧光原位杂交(FISH)等已让位于空间组学技术(图1)。不同空间组学技术在空间分辨率、覆盖范围、规模和通量以及多路复用能力等方面存在差异。根据研究目的,空间组学分析方法可以分为:1)基于靶向或多重探针、抗体的方法;2)基于转录组或下一代测序(NGS)的方法。
在原生空间背景下对单细胞进行分子分析的方法目前正迅速发展中。近年来,为覆盖更大数量的转录本或区域,传统的实验方法如条形码、免疫组化(IHC)和荧光原位杂交(FISH)等已让位于空间组学技术(图1)。不同空间组学技术在空间分辨率、覆盖范围、规模和通量以及多路复用能力等方面存在差异。根据研究目的,空间组学分析方法可以分为:1)基于靶向或多重探针、抗体的方法;2)基于转录组或下一代测序(NGS)的方法。
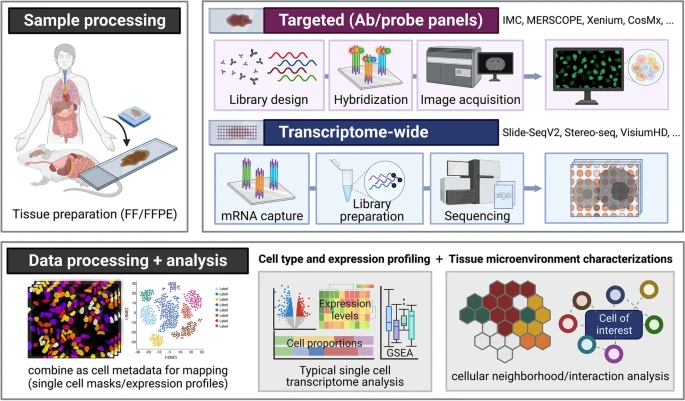
图1. 空间组学实验的典型工作流程。
大多数具有亚细胞水平分辨率的空间组学技术都是在载玻片(原位)上通过显微镜或NGS平台进行。目前,已有超50种不同的、可用的空间映射技术。在这篇综述中,研究团队重点介绍了在细胞和亚细胞水平(<10μm)研究细胞的最新技术(图2)。
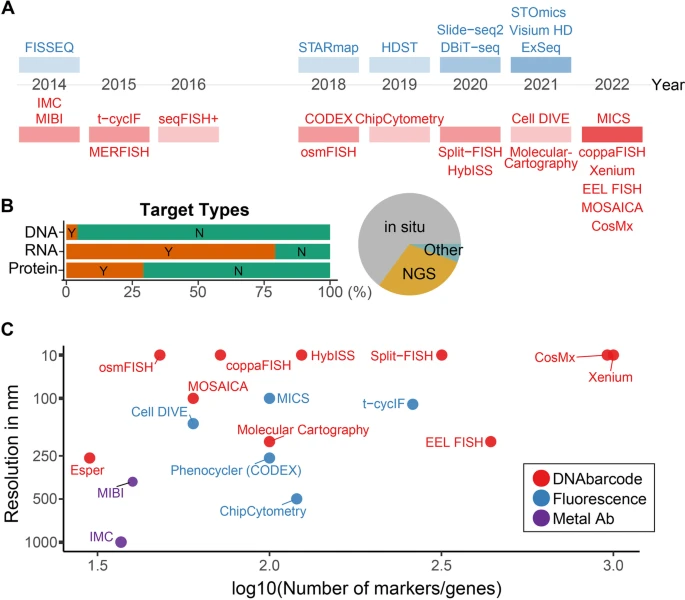
图2. 空间组学技术的时间轴、类型和规格。
使用抗体和RNA探针的靶向空间组学方法
当存在特定的、具有偏向性的分子实体可以识别细胞状态、身份和功能时,更适合使用靶向空间组学方法。传统的免疫荧光(IF)成像方法一次只能捕获4-5个通道,且受到光谱重叠的限制。使用含有可裂解linker的探针或抗体进行连续成像或条形码方案来区分同时或在同一波长内成像的多个探针,目前研究人员可以从一张玻片上分析50多个细胞实体。使用抗体进行连续染色、成像和漂白/剥离的技术包括循环免疫荧光(CyCIF)、迭代漂白扩展多重性(IBEX)、迭代间接免疫荧光成像(4i)等。
与抗体类似,mRNA探针(针对靶向转录本序列)也可以被设计用于靶向空间分析的panel。与基于抗体的方法相比,这类方法具有更短的protocol、更容易处理,并能够捕获更多的转录本(通常只有几百个转录本,最多可达10,000个)。此外,由于转录本的绝对数量会被计算为每个扫描点中的单个点,因此基于探针的方法比基于抗体的方法更具有定量性。
使用NGS平台进行转录组范围分析的空间组学
除了能够对细胞转录组进行全面观察的scRNA-seq方法外,近年来,科研人员还开发了几种以无偏倚方式对转录组进行空间分析的方法。早期方法(如LCM-seq、NICHE-seq和NanoString GeoMx DSP)侧重于有针对性地收集样本子集进行测序,并使用激光捕获显微镜(LCM)和基于光切割标记的方法,即收集由表面标志物或mRNA探针标记的特定区域并对其进行测序。这些技术允许用户定向分析只有10个细胞的特定ROI,可以解析每个样本的多个复制或组织类型。单细胞或亚细胞分辨率的方法依赖于空间条形码和原位测序,如荧光原位测序(FISSEQ)、STARmap、Slide-SeqV2、DBiT-seq、扩展测序(ExSeq)等,其测序位点的分辨率和化学性质因技术和扩增方法而异。
用于多模态研究的其他空间组学技术
与上面介绍主要关注基因表达谱(和表面标记蛋白)的空间组学技术类似,围绕空间基因组学、代谢组学、宏基因组学和表观基因组学的方法也正陆续出现。空间ATAC测序可通过原位Tn5转位和使用微流体设备进行探针结扎、标准消化和测序,然后进行染色质可及性分析。该方法的分辨率受微流体通道宽度(20μm)的限制,且分析依赖于核区域内的信号。
总体工作流程和实验设计标准
空间组学技术的总体实验和分析工作流程跨越了一些一致的特征。由于相关的探针和目标库已经商业化,实验通常使用小鼠或人体组织载玻片,但原则上,可以使用于任何带有注释基因组的物种,也可以使用来自特定培养平台的细胞培养物或切片(图3)。大多数技术兼容新鲜冷冻(FF)和福尔马林固定石蜡包埋(FFPE)样本。使用FF样本可以获得更好的RNA数据且提取过程更简单;FFPE样本能更完整地保存组织结构,更容易存储和运输。实验成功与否也取决于样本质量(RNA完整性和处理protocol)和技术(探针/抗体设计、透化和杂交的化学反应、成像和文库制备)的不同。
在选择方法和实验设计时要考虑的一个关键问题是,是否需要优先考虑特定的转录本、通路或细胞类型。如果已经知道特定的位点,那么选择基于原位捕获的方法往往能得到较好的实验效果;如果研究假设更多的是基于发现,那么基于NGS的方法可能更合适。
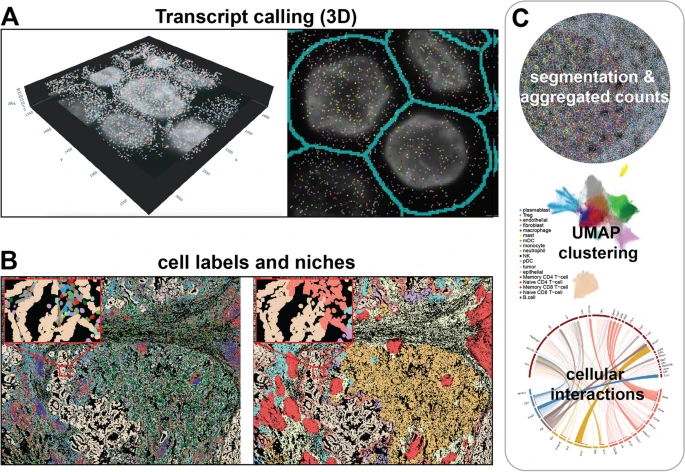
图3. 亚细胞空间组学数据和分析。
分析空间组学数据的计算方法
从亚细胞分辨率空间组学数据中捕获的信息主要被转化为单细胞格式(计数/细胞的量化)或csv文件,方便用于下游分析。研究团队介绍了用于一般空间分析的方法和已发布的软件包,以及用于高度多路复用图像可视化的现有多路复用图像查看器。
通常,大多数空间组学分析的输出涉及具有空间坐标信息的图像或条形码。这类空间信息可作为额外的元数据层来处理,有助于为下游分析提供信息。对空间组学数据的成功下游分析在很大程度上依赖于准确的细胞分割,这是一个依据捕获的坐标强度值推断细胞边界的过程。细胞分割根据可归纳的细胞特征进行。目前,基于监督学习的细胞分割算法主要有两大类:①基于计算机视觉的特征提取和机器学习模型(如随机森林)的组合;②基于卷积神经网络(CNN)的深度学习模型。
与传统的单细胞测序方法相比,高分辨率空间组学数据包含原生组织构象的成对空间信息,允许发现新的细胞类型、细胞相互作用和组织结构。目前,该领域的大多数方法都是构建细胞图,以细胞为节点、细胞间的阈值空间距离为边缘来利用空间信息。这一领域的两个大的分支是:①空间微环境分析,根据空间环境对细胞进行分组和分析;②细胞间相互作用的推断,研究一对细胞类型在原生组织构象中相互作用的频率。
此外,将空间组学数据与其他数据模式相结合(如单细胞RNA或ATAC-seq),可以在所研究的分子实体和细胞数量方面对空间检测进行补充,从而更全面地了解细胞系统。集成不同数据集的一种流行方法包括确定可变或“显著”的特征子集,并将其以作为跨两种数据模式的锚点。多组学因子分析(MOFA)为数据模态的整合引入了一个统计框架,特别是在来自同一组细胞的公共样本空间内。
综上所述,空间组学领域在过去几年蓬勃发展,并从根本上提高了原位实验的广度和分辨率。这些技术现在可以在50-100nm的空间分辨率下检测超过10,000个独特的基因靶点。探针化学、图像采集和商业化的发展推动了其成本的降低,正将空间组学技术转变为所有实验室都能使用的普通技术,有望为传染病、肿瘤学和基础科学的新发现提供动力。


