Nat Methods:何川团队开发RNA结合蛋白结合位点原位逆转录测序分析的新方法
时间:2024-01-12 21:19:23 热度:37.1℃ 作者:网络
RNA结合蛋白(RBPs)通过与RNA靶点动态相互作用来调节多种细胞过程。然而,仍然缺乏有效的方法来捕获RBPs与其RNA靶点之间的稳定和瞬时相互作用,特别是当相互作用是动态的或样品有限时。
2024年1月10日,芝加哥大学何川团队在Nature Methods 在线发表题为“Profiling of RNA-binding protein binding sites by in situ reverse transcription-based sequencing”的研究论文,该研究提出了一种基于逆转录的RBP结合位点测序(ARTR-seq)的检测方法,该方法依赖于抗体引导下RBP结合RNA的原位逆转录来识别RBP结合位点。
ARTR-seq避免了紫外线交联和免疫沉淀,允许从少至20个细胞或组织切片中高效和特异性地鉴定RBP结合位点。利用快速甲醛固定的优势,ARTR-seq能够在短时间内捕获RBP的动态RNA结合,正如在短至10分钟的时间范围内对应力颗粒组装过程中G3BP1的动态RNA结合的分析所证明的那样。

RBPs与RNA靶标动态相互作用,调控RNA转录、剪接、修饰、定位、翻译和降解等各方面的命运。RBPs的功能障碍或与RNA底物的结合可导致各种缺陷甚至疾病。捕获RBP-RNA相互作用的有效方法,特别是动态甚至瞬时相互作用,对于更好地理解RBP及其对靶RNA的功能作用至关重要。
广泛使用的识别RBP靶点的方法是基于特异性RBP及其结合RNA的免疫沉淀(IP),通过直接RNA IP (RIP)或共价捕获辅助的交联IP (CLIP) 。特异性RBP结合的底物RNA可以使用针对RBP的抗体通过RIP或CLIP富集,然后通过高通量测序(seq)在整个转录组中分析RBP靶点。CLIP-seq通过共价交联捕获底物RNA上的RBP结合位点。RNase处理可以消化RNA的无RBPs区域,提高结合位点检测的分辨率。CLIP-seq变体如PAR-CLIP或eCLIP可提高交联效率、特异性或结合位点分辨率。这些方法虽然有效且被广泛使用,但也有局限性。由于IP效率低,它们通常需要大量的起始材料;紫外交联是一种低效的化学反应。最近报道的tRIP-seq和LACE-seq可以应用于低输入样本,但代价是降低库的复杂性。
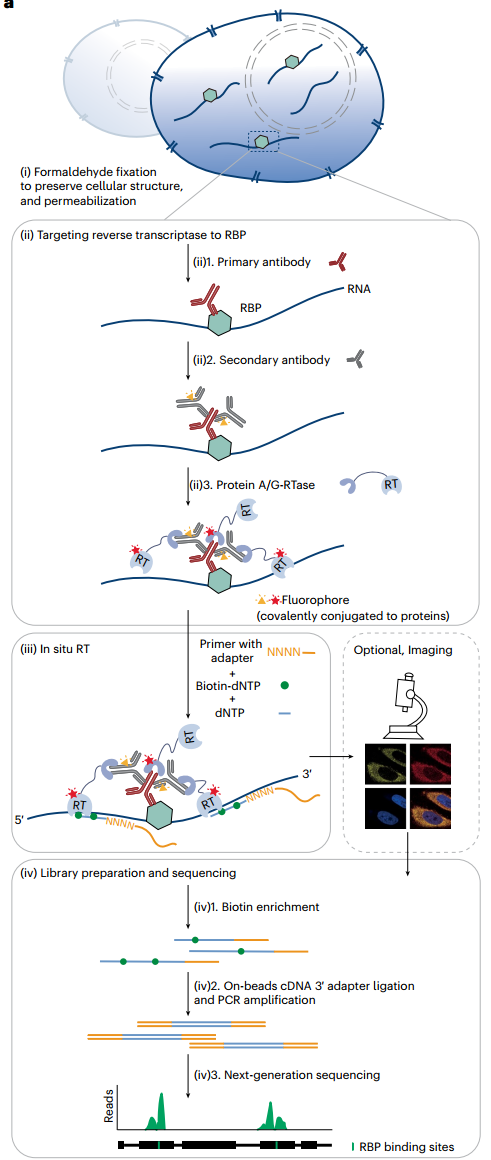
ARTR-seq方案(图源自Nature Methods )
TRIBE和STAMP型方法将RBP与RNA碱基编辑器融合,在RBP结合位点附近引入突变,绕过IP来识别RBP结合位点。这些方法可以很容易地应用于研究RBP在活细胞中的结合,并且在单细胞水平上使用有限的材料。它们在研究中的应用提供了新的机会;然而,这些基于编辑的方法仍然具有的局限性。它们需要通过在生殖系或细胞系中插入碱基编辑蛋白来操纵基因组,这阻碍了它们在原代细胞和组织中的应用。
诱导编辑蛋白表达通常需要大约24小时或更长时间,这不能用于监测RBP的动态RNA结合。这些碱基编辑器有自己的序列偏好,可能会改变目标RBP的本地结合特征。RT&Tag,一个从CUT&Tag策略衍生出来的方法,该方法通过寡核苷酸(dT)引物启动的逆转录(RT)和分离细胞核中全长RNA-互补DNA (cDNA)异双工的Tn5标记来分析RBP-RNA相互作用。RT&Tag可以识别RBP与多聚腺苷化RNA的结合,但对非多聚腺苷化RNA和细胞质RBP的结合无效。由于Tn5酶在异双工上的效率较低,需要25,000-100,000个细胞核才能获得足够的转录组范围内的结合信号。
为了克服现有方法的局限性,该研究引入了一种基于RBP结合位点测序(ARTR-seq)的检测方法,通过原位RT捕获RBP-RNA相互作用。研究证明,ARTR-seq可以使用少至20个细胞或单个组织切片,以良好的测序质量敏感地描述RBP靶点。此外,成像步骤可以很容易地内置到ARTR-seq程序中,提供RBPs的直接空间信息。通过ARTR-seq,发现剪接因子与RNA N6 -甲基腺苷(m6 A)修饰的YTH家族解读蛋白的结合模式不同。ARTR-seq可以无偏地检测RBP在细胞质和细胞核中的RNA结合,并测量RBP在RNA底物上的结合强度。此外,ARTR-seq可以在10分钟的小时间尺度上监测压力颗粒(SG)组装过程中G3BP1的动态RNA结合。
原文链接:
https://www.nature.com/articles/s41592-023-02146-w


