Scientific Reports:对新冠病毒采用新一代碱基序列分析方法“长读取测序“将大幅减少了时间和费用
时间:2021-07-05 11:01:54 热度:37.1℃ 作者:网络

近日,美国南加州大学(USC)医学院研究团队SungYong Park等开发出了更快、更准确的新型冠状病毒肺炎(COVID-19)基因分析(序列)技术。这项成果发表在《科学报告Scientific Reports》,文章整体介绍了采用新一代碱基序列分析方法“长读取测序(long-read sequencing)”,分析新冠肺炎病毒的方法,受到了学术界关注。
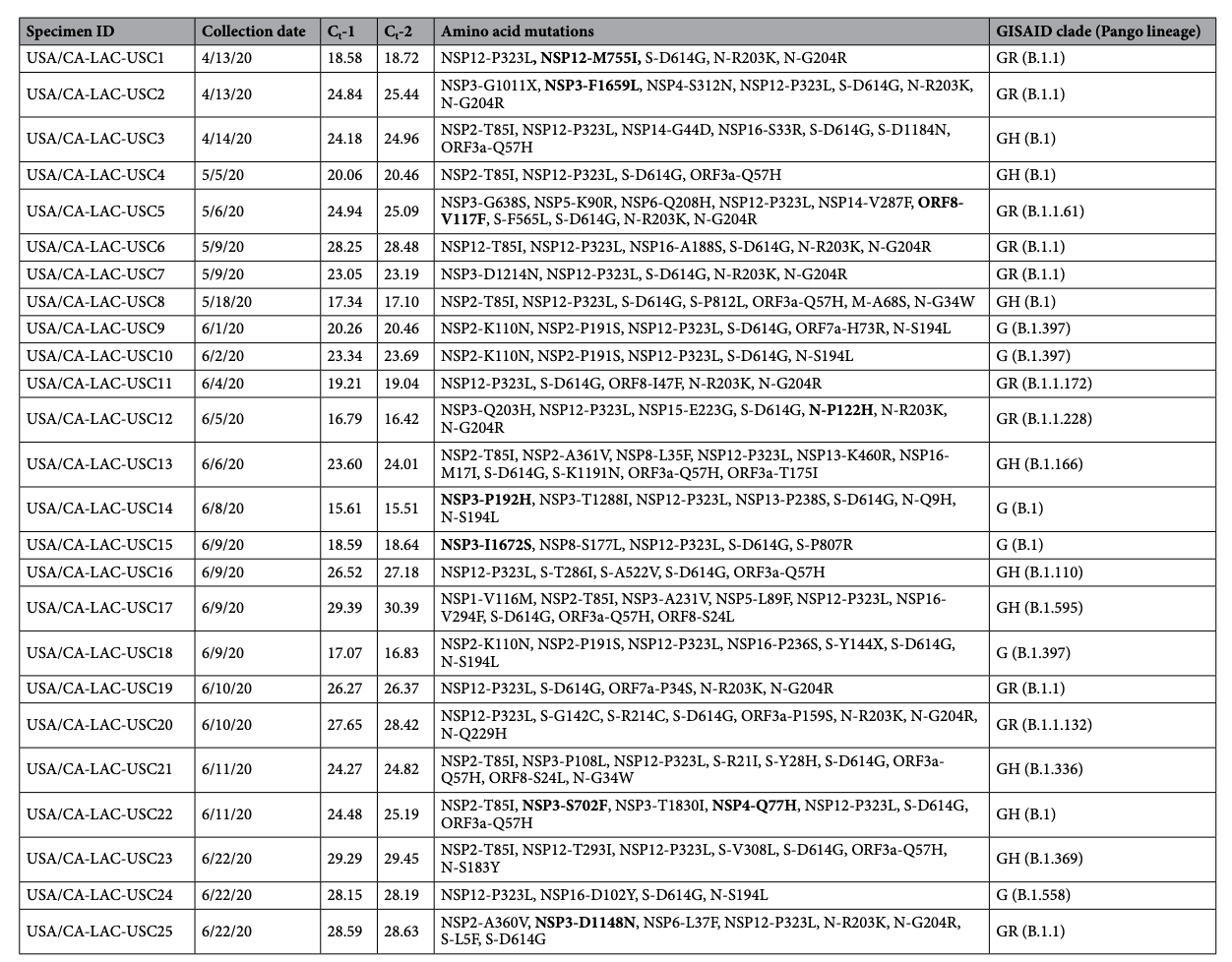 来自 COVID-19 残余物的 25 个全基因组序列的氨基酸突变、进化枝和谱系NP/OP 标本
来自 COVID-19 残余物的 25 个全基因组序列的氨基酸突变、进化枝和谱系NP/OP 标本
该研究,运用该技术,成功分析了去年4~6月新冠病毒在美国加利福尼亚州洛杉矶疫情高峰时感染的25人的病毒基因碱基序列。之前使用的是将新冠病毒基因细分成约100个后进行分析,然后掌握病毒的真面目及变异与否的“短读取测序(short-read sequencing)”。相反,研究组将基因大致分为三个部分进行分析,大幅减少了时间和费用。有期待称,将把之前需要约10天的测序时间缩短到数日内。
估计的低测序成本表明与当前基于短读长的 SARS-CoV-2 全基因组测序方法相比具有成本优势。 对于样品,CorvGenSurv 所需的时间约为 15 小时文库制备和 30 小时测序。 因此,我们具有成本效益的工作流程可能会加强实时大规模基因组 SARS-CoV-2 监测的实施。

团队开发了这种高精度、高性价比的用于 COVID-19 基因组监测的 SARS-CoV-2 全基因组测序平台称为 CorvGenSurv(冠状病毒基因组监测)。 CorvGenSurv 直接扩增来自 COVID-19 的病毒 RNA患者的鼻咽/口咽 (NP/OP) 拭子标本并对 SARS-CoV-2 进行测序通过长读长、高通量测序将整个基因组分为三个片段。测序三段全基因组显着减少测序数据浪费,从而防止基因组覆盖率下降。我们通过控制基因组验证了我们管道的精度RNA测序和桑格测序。研究对这些样本发现这些序列在 G 进化枝中高度多样化,具有九个新氨基酸突变包括 NSP12-M755I 和 ORF8-V117F。凭借其易于适应的设计,CorvGenSurv
授予广泛的基因组监测机会,允许对突发公共卫生事件立即做出反应威胁。
新冠肺炎病毒爆发后,世界卫生组织(WHO)一直敦促各国加快新冠病毒测序的速度。但是,从现有方法来看,全球测序平均比率仅超过1%,在应对前所未有的传染病大流行方面存在局限性。有观测认为,考虑到这一点,此次研究将对新冠病毒的终结做出巨大贡献。特别是,研究组分析的基因中,有属于最近迅速扩散的德尔塔变异、贝塔变异等,因此对变异病毒的研究也会起到相当大的作用。
参考资料:Park, S.Y., Faraci, G., Ward, P.M. et al. High-precision and cost-efficient sequencing for real-time COVID-19 surveillance. Sci Rep 11, 13669 (2021). https://doi.org/10.1038/s41598-021-93145-4


