NAR:中山大学杨建华/屈良鹄团队发表RMBase v3.0平台:解码RNA修饰图谱、机制和功能
时间:2023-12-16 15:42:38 热度:37.1℃ 作者:网络
2020年表观遗传学研究的是RNA在转录后发生的化学修饰,目前已经鉴定出了170多种RNA化学修饰,这些修饰在RNA的加工、转运、翻译和降解中具有多种调节作用。但大多数RNA修饰的发生、生物学作用和功能机制仍不清楚。
2015年,中山大学杨建华教授、屈良鹄教授团队构建了RMBase平台,对高通量测序产生的公共RNA修饰位点进行大规模整合,并提供了多种细胞类型的RNA表观遗传图谱,帮助扩大人们对RNA修饰潜在功能的理解。2017年,研究团队对RMBase平台进行了升级改进,发布了RMBase 2.0,涵盖了100多种转录产物的RNA修饰,揭示了RNA修饰参与的复杂转录后调控网络。
近日,杨建华/屈良鹄团队对该平台再次进行了升级,发布了RNA修饰综合分析平台RMBase v3.0,可用于探索RNA修饰的转录组图谱、生物发生、相互作用组和功能。该研究已发表在Nucleic Acids Research,文章题为“RMBase v3.0: decode the landscape, mechanisms and functions of RNA modifications”的文章。
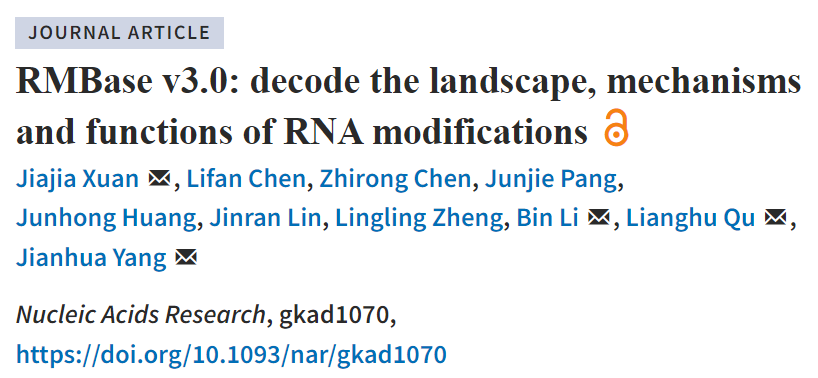
RMBase v3.0含有八个模块:“RNA Modifications”模块使用新的流程挖掘了数千个表观转录组数据集,揭示了62个物种73种RNA修饰的图谱。“Genes”模块支持通过基因和转录本检索RNA修饰图谱和簇。“Mechanisms”模块探索了23,882个酶催化或snoRNA引导的修饰位点,阐明了生物发生机制。“Co-localization”模块系统地阐述了各种细胞系中14种组蛋白修饰和6种RNA修饰之间的潜在相关性。“RMP”模块研究了146种RNA修饰蛋白在18种癌症中的差异表达谱。“Interactome”模块整合了73种RNA修饰与RBP结合事件、miRNA靶点和SNP之间的相互作用关系。“Motif”模块阐明了从表观转录组数据集中鉴定的11种RNA修饰的富集基序。“Tools”模块引入了新颖的网页版“modGeneTool”,可用于注释修饰。总的来说,RMBase v3.0为研究RNA修饰提供了各种资源和工具。
此外,RMBase 3.0还提供了两个用户友好的界面,“Download”和“Help”,允许用户下载数据库中存储的所有数据。此外,用户可以从主页进入每个模块页面,深入了解RMBase提供的原始数据信息和汇总结果。
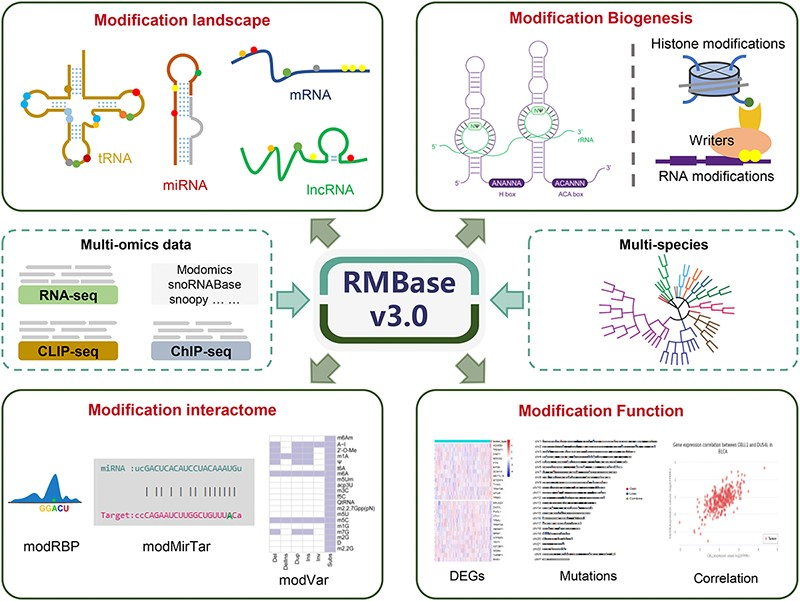
图:研究概要
利用ModFinder流程揭示62个物种的73个RNA修饰图谱
研究团队收集并整合了来自62个不同物种的数千个RNA修饰测序数据集,并整合了来自100多项研究和5个公共数据库的经鉴定的修饰。同时,为了进行RNA修饰的鉴定和功能分析,研究人员还收集了所有62个物种的基因组序列和基因注释。
为了全面破译RNA修饰图谱,研究团队开发了一个统一的计算管道“ModFinder”。研究人员分析了1,880个表观转录组测序数据集,检测了精确的m6A修饰位点,并构建了基于公共数据库的RNA修饰比对和分布特征。经过严格的质量控制和过滤,鉴定了62个不同物种73种RNA修饰的100多万个准确的RNA修饰位点,涵盖哺乳动物、植物、脊椎动物、真菌、昆虫、后生动物、原生生物、细菌和病毒(图1)。研究人员对鉴定的修饰位点进行了全面的注释,包括基因名称、基因类型、生物型特征、PWM评分、序列背景、RNA二级结构、生物发生机制和细胞来源。
转录组中RNA修饰的图谱显示了不同RNA修饰的偏好。例如,与其他RNA相比,m6A修饰在mRNA中最丰富。此外,使用RMBase v3.0的“modMetagene”生成的不同RNA修饰的元基因(metagene)图显示出不同的分布模式,这与先前的报道一致。总之,在各种RNA中观察到的不同RNA修饰的不同模式可能与其不同的生物发生机制和生物功能密切相关。
图1. RMBase v3.0的工作流。
各种RNA修饰簇的鉴定
鉴于基因内成簇的m6A修饰在生物过程中起着关键作用,因此研究团队对RNA修饰的分布模式进行了全面分析。研究人员共发现了5,871个m6A簇、98个m5C簇、18个2′-O-Me簇、17个Ψ簇、59个A-I簇。m6A簇分布在mRNA、lncRNA和假基因中,其中大多数在mRNA中。进一步的统计分析显示,单个mRNA可以包含一个或多个m6A簇,长度从120nt到3000nt不等。这些RNA修饰簇已被整合到“modGene”中,并用基因本体(GO)术语进行了注释,为用户提供了一种方便的方法来探索其生物学功能。
不同RNA类型中snoRNA引导的RNA修饰
真核Ψ和2′-O-Me修饰主要由H//ACA snoRNA和C//D snoRNA催化。研究人员开发了新流程“Sno2Psi”(图2A),可以系统地识别各种RNA分子上snoRNA引导的Ψ。最终共检测到141个人类snoRNA引导的Ψ,包括rRNA、tRNA、mRNA、lncRNA和snRNA中的94个新型snoRNA引导的Ψ和47个已知snoRNA引导的ψ(图2B)。
此外,研究团队进一步开发的“Sno2Nm”流程,可以系统地检测各种人类RNA分子上C//D snoRNA引导的2′-O-Me位点。结果显示,Sno2Nm鉴定了444个snoRNA引导的2′-O-Me位点,这些位点位于rRNA、tRNA、lncRNA、mRNA、snRNA和假基因内。以上结果为Ψ和2′-O-Me修饰的生物合成提供了有价值的机制见解。

图2. 使用Sno2Psi鉴定由snoRNA引导的Ψ修饰位点。
组蛋白修饰与RNA修饰的共定位研究
研究团队利用基于多项式回归的“modHistone”流程对14种组蛋白修饰的ChIP-seq数据和12种不同细胞系的表观转录组测序数据进行综合分析。研究人员通过研究组蛋白修饰在RNA修饰位点附近的分布模式,推测了组蛋白修饰和RNA修饰之间的共定位关系。当组蛋白修饰在RNA修饰位点有显著富集峰时为正相关,当组蛋白修饰在RNA修饰位置的分布有明显的低谷时为负相关。从该流程获得的结果已集成到“Co-localization (modHistone)”模块中。

图3. 组蛋白修饰与RNA修饰的分布相关性。
RNA修饰蛋白在肿瘤中的表达谱和突变图谱的探索
RNA修饰蛋白(RMP)包括催化(Writer)、去除(Eraser)和识别(Reader)RNA修饰的RBP,其失调和突变对各种癌症的发生和发展有重要影响。为了确定RNA修饰在肿瘤中的潜在功能,研究人员系统地阐明了18种肿瘤中146个RMP的异常表达谱和突变模式。差异表达谱显示,在所有16种类型的肿瘤中,都存在RMP失调,85.6%的RMP在大多数肿瘤中出现上调或下调(图4A)。以上发现表明,RMP基因在肿瘤中通常异常表达,且更容易下调。虽然RMP在肿瘤中表现出频繁的异常表达和突变,但部分研究确定的SCNA驱动RMP和体细胞突变驱动RMP的数量相对有限(分别为26%和11%),这提示RMP的异常和突变可能作为调控因素而非驱动因素,在肿瘤的发展中发挥关键的调节作用。
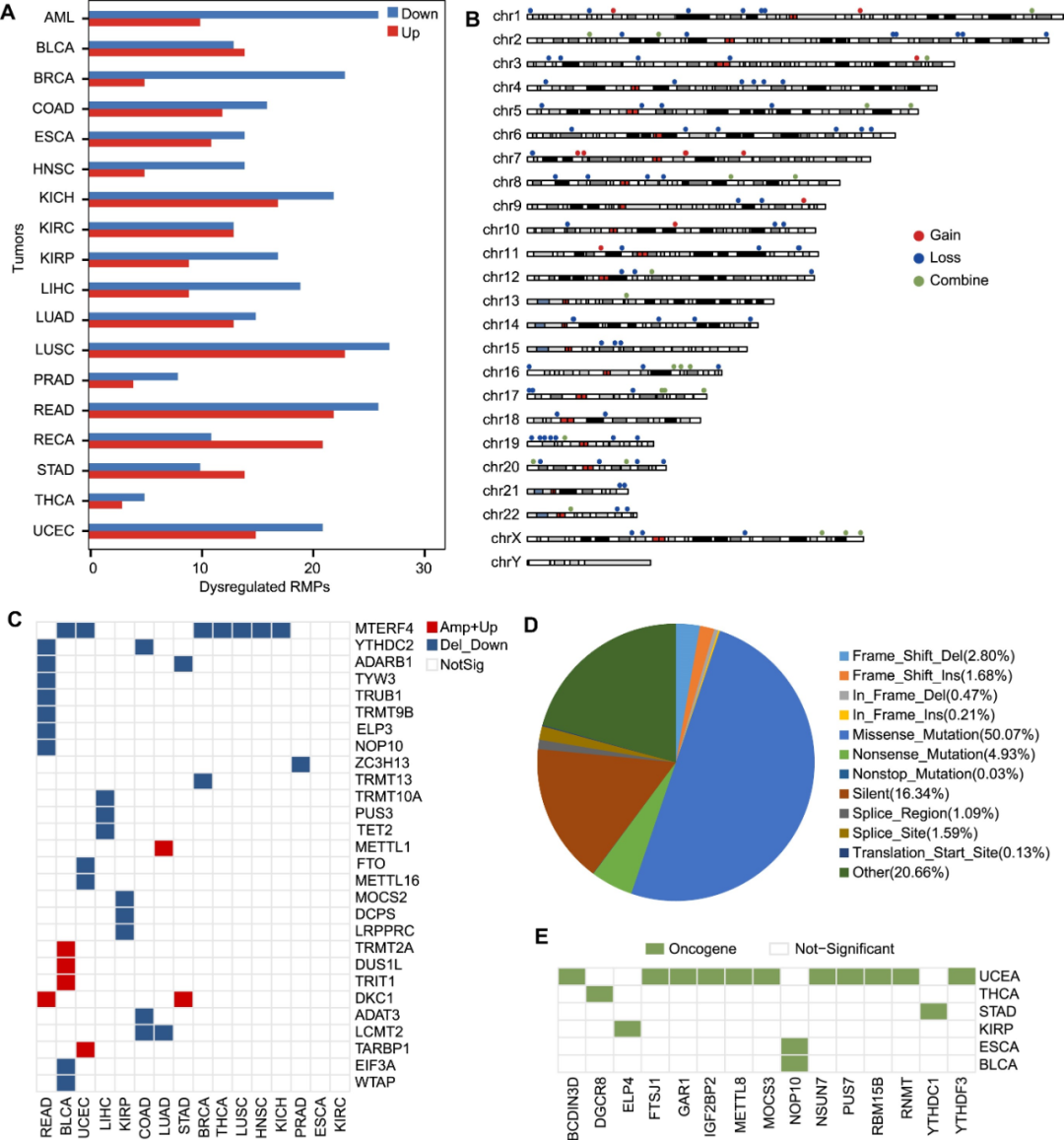
图4. RNA修饰蛋白在肿瘤中失调表达谱和突变图谱的探索。
综上所述,RMBase v3.0采用多种标准化方法和流程解码了62个物种73种RNA修饰的最全面图谱、潜在机制和功能。与其他数据库和之前的RMBase v2.0版本相比,RMBase v3.0有显著的进步,有望成为RNA表观转录组研究领域的标准资源库。
中山大学生命科学学院杨建华教授、屈良鹄教授、李斌特聘副研究员为文章共同通讯作者,Jiajia Xuan为第一作者。
论文原文:
Xuan J, Chen L, Chen Z, et al. RMBase v3.0: decode the landscape, mechanisms and functions of RNA modifications. Nucleic Acids Res. Published online November 13, 2023. https://doi.org/10.1093/nar/gkad1070


