Genome Biology:基于长读长RNA测序鉴定RNA编辑位点的新算法L-GIREMI
时间:2023-08-24 17:49:10 热度:37.1℃ 作者:网络
腺苷到肌苷(A-to-I)RNA编辑是一种重要的转录后修饰,也是人类细胞中最普遍的RNA编辑方式之一,其在编码区和非编码区都会发生,极大地丰富了转录组的多样性。编码区RNA编辑会直接影响蛋白质功能,非编码区RNA编辑可通过影响选择性剪接或RNA稳定性来影响基因表达。此外,RNA编辑还会影响微小RNA的成熟,导致RNA编辑和RNA干扰之间的串扰。
近年来,长读长RNA-seq方法逐渐成为研究RNA生物学的有力工具,该方法克服了短读长RNA-seq固有的转录本组装歧义,促进了人们对转录组多样性的理解。目前已有多种方法用于解析长读长RNA-seq数据,但多数方法主要集中在转录本异构体鉴定及其丰度分析上,在鉴定和分析RNA中的单核苷酸变异(SNV)方面,如遗传突变、RNA编辑位点,尚未得到充分探索。
近日,美国加州大学洛杉矶分校、欧文分校的研究团队合作在Genome Biology发表了题为“L-GIREMI uncovers RNA editing sites in long-read RNA-seq”的文章。研究团队提出了一种无需基因组信息即可在长读长RNA-seq中检测和分析RNA编辑位点的新方法——L-GIREMI。L-GIREMI可有效地处理测序错误和reads偏差,并使用基于模型的方法对RNA编辑位点进行评分;能够分析单个RNA分子的RNA编辑模式、多个RNA编辑事件的共现情况以及检测等位基因特异性RNA编辑。该方法为研究长读RNA-seq中的RNA核苷酸变异提供了新的机会。

文章发表在Genome Biology
对于不同类型的RNA变异,mRNA中RNA变异的alternative allele之间的连锁模式也不同。在使用长读长RNA-seq进行检测时,同一mRNA中的多个变异可以被每个read覆盖,因此其在捕获等位基因连锁方面具有主要优势。利用长读长RNA-seq这一特性,研究团队开发了L-GIREMI方法来识别RNA编辑事件。
L-GIREMI算法主要由四个步骤组成(图1)。1)检查每个read的链,并在必要时进行校正。2)根据使用RNA-seq数据检测RNA编辑位点的常见操作,获取BAM文件中的错配位点并进行预过滤。3)计算同一基因中错配位点对之间的互信息(MI),即计算每个未知错配相对于相同reads覆盖的假定SNP的平均MI,以及假定杂合SNP对的MI。对两种MI值进行比较,预测未知错配中的RNA编辑位点。4)将预测的RNA编辑位点作为训练数据,导出广义线性模型(GLM)。将候选位点的序列特征和等位基因比率纳入GLM中作为预测变量,并计算每个错配的得分。
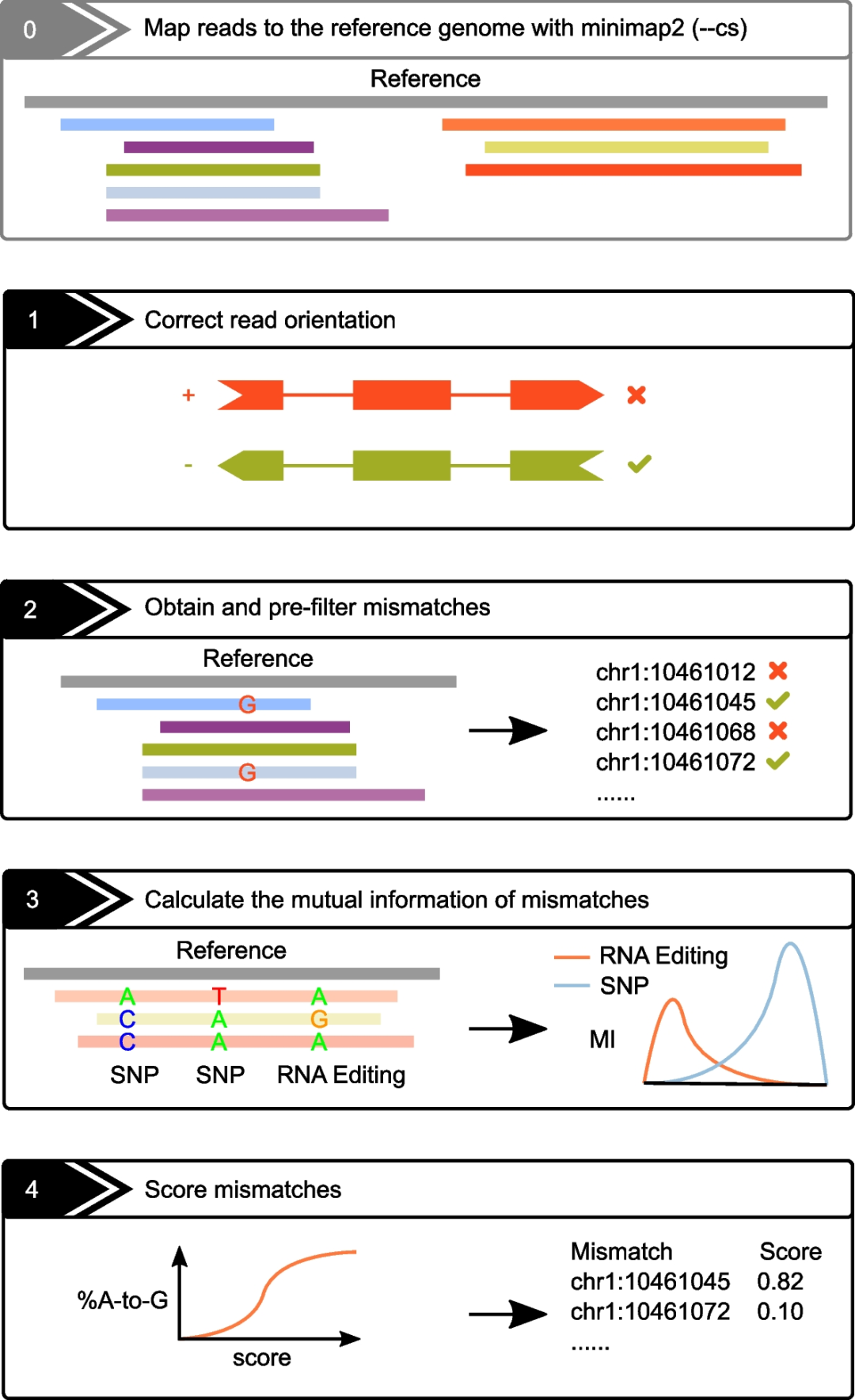
图1. L-GIREMI算法的原理图。
研究团队使用来自阿尔茨海默病(AD)患者大脑样本的数据集测试了L-GIREMI的性能(图2)。与预期一致,大多数reads包含至少一个错配或插入/缺失(InDel),平均每个read有14个错配、38个缺失和11个插入。这表明L-GIREMI克服了长读长RNA-seq在解决真正核苷酸变异时遇到的挑战,能够从数据集中有效地检测RNA编辑位点。
通过L-GIREMI得到SNP的MI分布,研究团队计算了每个错配位点的经验p值,并将p<0.05的位点作为候选RNA编辑位点。该步骤共检测到13,442个编辑位点,其中83.3%为A-to-G类型(可能由A-to-I编辑所致)。进一步,将上述位点当作GLM模型的训练数据进行分析,最终在AD数据集中总共检测到28,584个RNA编辑位点,其中A-to-G位点占比较高,表明L-GIREMI具有高准确性。综上,L-GIREMI能够在较大的总reads覆盖范围内高精度捕获长读长RNA编辑位点。
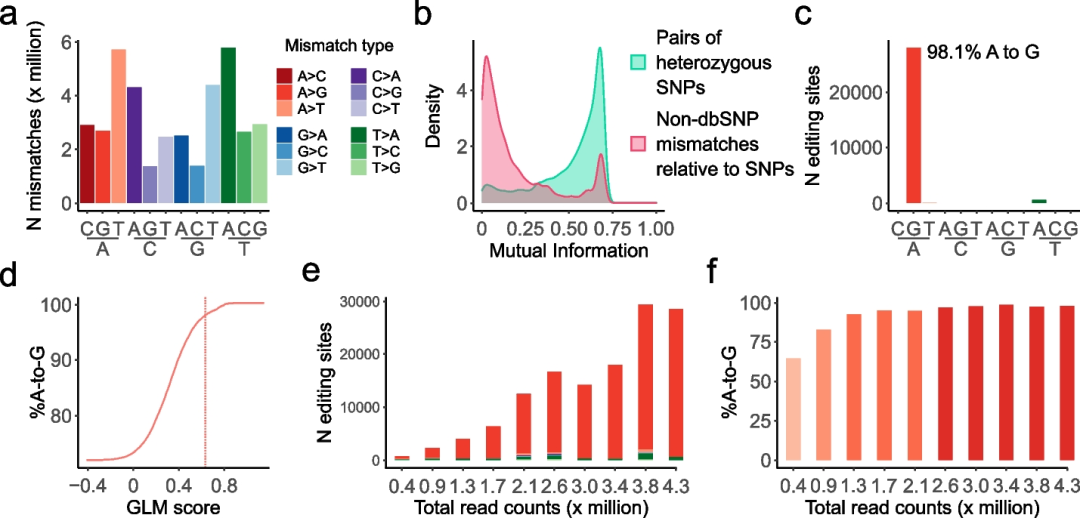
图2. 阿尔茨海默病患者大脑样本的长读长RNA-seq数据中RNA编辑位点的鉴定。
研究团队使用L-GIREMI对GM12878数据集进行了检测(图3)。结果显示,与AD数据集一致,L-GIREMI在GM12878数据集中检测到的大多数RNA编辑位点位于非编码区和Alu元件中。为探究一个基因的多个编辑位点倾向于同时出现在RNA分子的一个子集中还是相互独立,研究团队使用长读长RNA-seq数据进行分析。首先检测了Alu元件的每个read中观察到的编辑位点数量的基尼指数,发现同一Alu元件中编辑位点存在共存。
接下来,研究团队计算了基因中成对编辑位点的MI值和随机排列编辑位点的MI值,发现前者MI值显著高于后者。值得注意的是,编辑位点的MI远低于SNP对之间的MI。上述结果表明,相同RNA分子存在RNA编辑位点共存现象,但其水平显著低于遗传连锁。

图3. L-GIREMI检测到Alu元件中A-to-I RNA编辑位点的共存现象。
为探究等位基因特异性RNA编辑是否影响大多数编辑位点,研究团队使用GM12878长读长RNA-seq数据进行了分析(图4)。具体而言,研究团队计算了REDIportal数据库中所有已知RNA编辑位点相对于GM12878中已知SNP的MI值,这些SNP可在长读长RNA-seq数据中检测到(图4)。结果显示,REDIportal定义的多数已知编辑位点的MI值相对较低,只有一小部分MI值大于0.3,表明等位基因特异性编辑可能只影响少数编辑位点。
随后为排除假阳性的可能,研究团队使用Sanger测序测试了6个可能的等位基因特异性编辑位点。结果显示,其中四个位点被确认是RNA编辑位点,但剩余两个位点既未被编辑也没有形成SNP。这一结果表明等位基因特异性RNA编辑确实存在,但相对罕见。综上,一般不建议使用L-GIREMI来检测新编辑位点的等位基因特异性编辑,但L-GIREMI中的MI计算可用于揭示已知RNA编辑位点的等位基因特异性编辑。
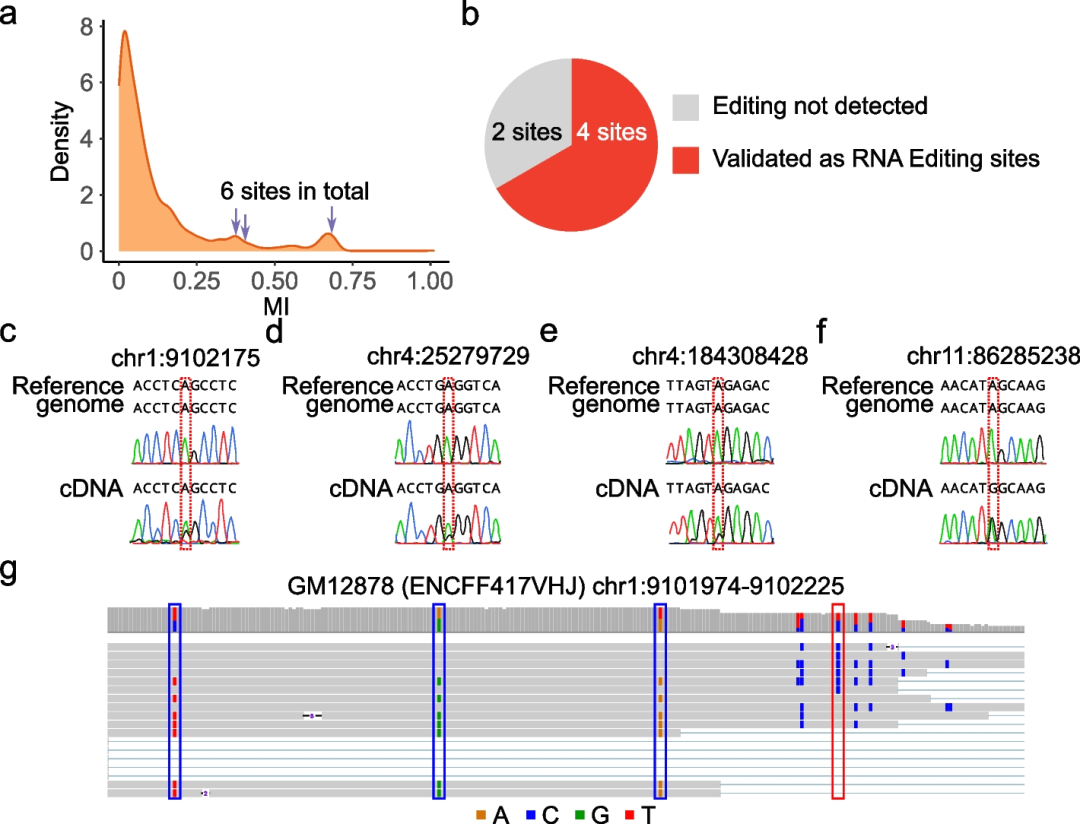
图4. GM12878长读长RNA-seq数据中反映的等位基因特异性编辑。
在检测RNA-seq read中的RNA编辑位点时,研究团队观察到一些长reads会跳过一个区域,其通常在RNA编辑位点附近。为探究长read中的区域跳跃是否由RNA高度结构化所致,研究团队分析了AD数据集和GM12878数据集,鉴定了含有编辑富集区域的dsRNA结构(图5)。
结果显示,上述两个数据集中分别至少有一个read覆盖了36,166和17,293个预测的dsRNA。在这些预测的dsRNA中,大约20%的read与跳跃区域重叠。在AD和GM12878数据集中,分别有34.4%和31.6%的dsRNA在≥50%的read出现跳跃模式。跳跃区域的中位长度约为600-800个bp,约是两个相邻Alu重复序列的长度。
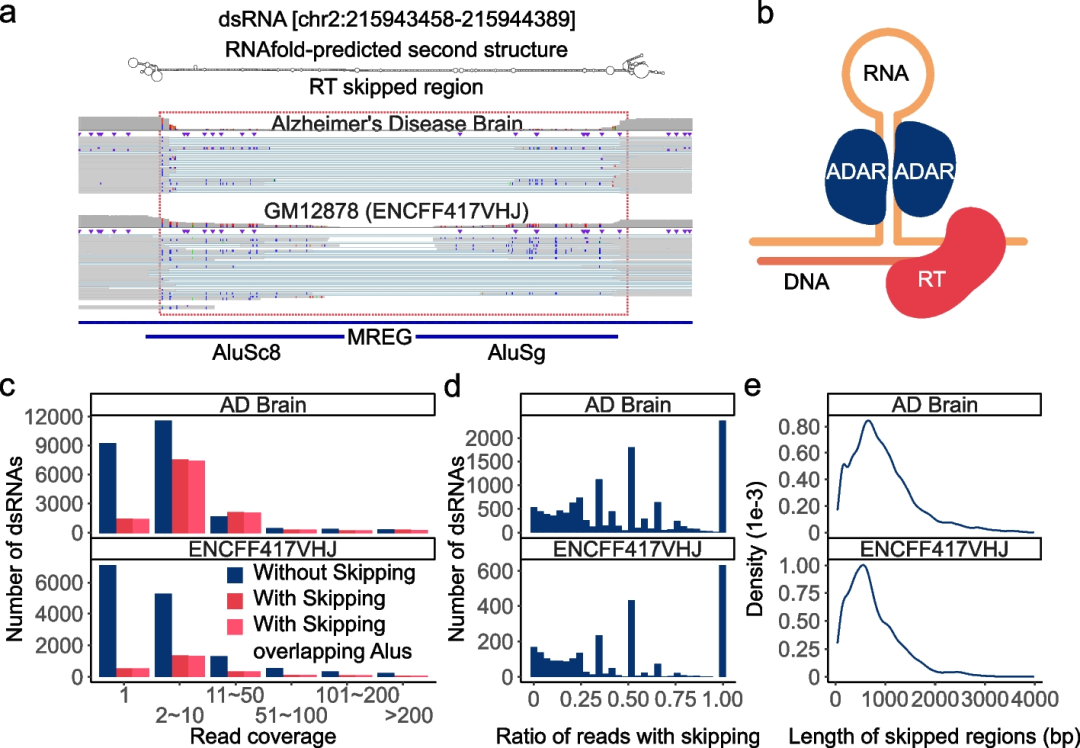
图5. 长读长RNA-seq检测到高度结构化的区域。
综上所述,研究团队开发了一种名为L-GIREMI的算法,可应用于检测单分子中的RNA编辑位点、等位基因特异性RNA编辑以及由dsRNA结构存在而导致的区域跳跃。该研究揭示了有关单分子和双链RNA结构中RNA编辑发生的新见解,L-GIREMI方法为研究长读长RNA-seq中的RNA核苷酸变异提供了一种强大的手段。
参考文献:
1.Liu, Z., Quinones-Valdez, G., Fu, T. et al. L-GIREMI uncovers RNA editing sites in long-read RNA-seq. Genome Biol 24, 171 (2023). https://doi.org/10.1186/s13059-023-03012-w


