中国青年学者联手发Science:推出三维人类脑细胞图谱!
时间:2023-10-15 14:29:12 热度:37.1℃ 作者:网络
理解大脑内多样化细胞类型的复杂基因调控机制对于理解大脑在健康和疾病中的功能至关重要。最近高通量表观基因组分析的兴起带来了对这些基因调控机制的重大突破性认识。
2023年10月13日,美国索尔克生物学研究所Joseph R. Ecker博士的团队,全面地考察了人类大脑中细胞的表观基因组,通过在三名成年男性大脑的46个区域中以单细胞分辨率检测DNA甲基化和染色质构象,共涵盖了51.7万个细胞(其中39.9万个神经元和11.8万个非神经元)。研究鉴定了188种不同的细胞类型,并描述了它们的分子特征。综合分析结果显示,不同细胞类型、大脑皮层区域以及基底神经节结构之间的DNA甲基化、染色质可及性、染色质组织和基因表达都呈现出协调的变化。此外,研究还开发了单细胞甲基化条形码(scMCodes),利用特定基因组位点的甲基化状态可可靠地预测大脑细胞类型。该论文成果以“Single-cell DNA methylation and 3D genome architecture in the human brain”为题,发表在Science期刊上。论文共同第一作者是美国索尔克生物学研究所的田伟和周景天博士。

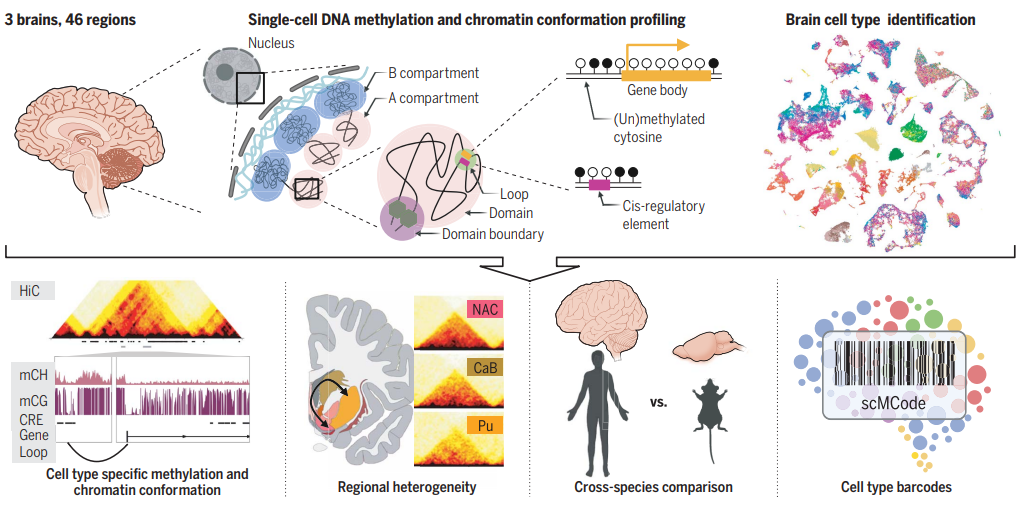
在人类大脑中进行DNA甲基化和染色质构象分析。这项研究在三名成年人大脑的46个区域中以单细胞分辨率检测了51.7万个细胞的DNA甲基化和染色质构象。这一全面的调查使得能够深入分析细胞类型的复杂性,基因调控的表观遗传多样性,人类与小鼠大脑之间的比较,以及构建大脑细胞类型的甲基化条形码。
【基于表观基因组的大脑细胞类型分类】
研究涉及46个不同的大脑区域,包括大脑皮层、基底前脑、基底核、海马、丘脑、中脑、脑桥和小脑。研究使用了分子标记技术将神经元和非神经元分离,然后对这些细胞进行DNA甲基化和染色质构象的单细胞分析。研究通过分析DNA甲基化数据将细胞分为不同的主要类型和子类型,其中一些类型具有空间特异性。这些细胞类型的DNA甲基化特征与基因表达数据相一致,并且与其他分子数据相吻合,验证了细胞类型的确定。此外,研究还探讨了不同细胞类型之间的DNA甲基化和染色质构象的差异,以及它们在不同脑区域的分布情况。
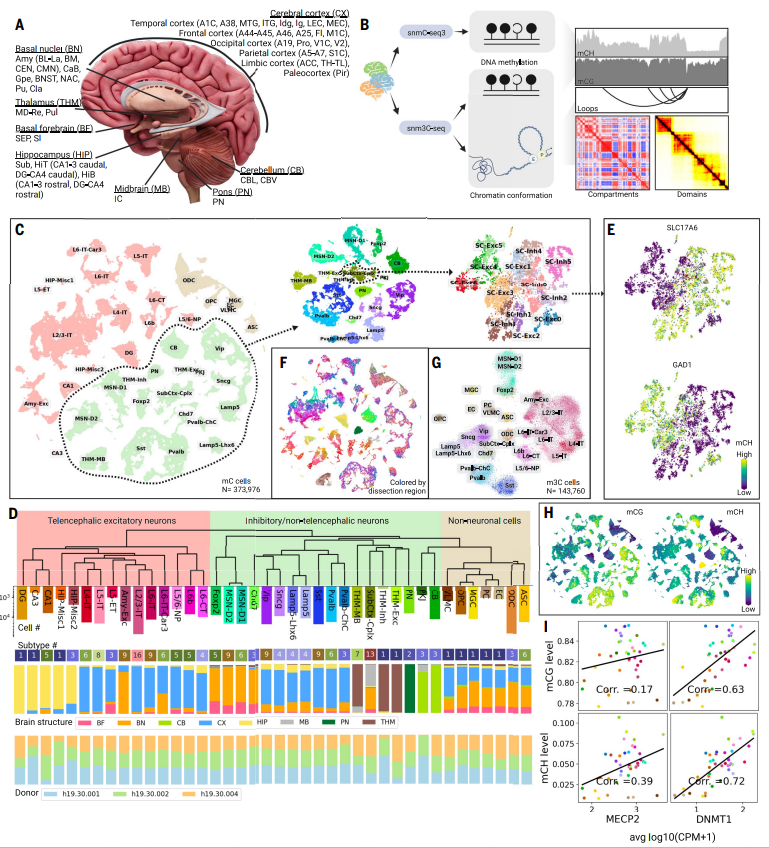
图1. 人类大脑细胞的表观基因组分析,使用snmC-seq3和snm3C-seq。(A) 覆盖的人类大脑结构和区域。(B) snmC-seq3和snm3C-seq的分析模式示意图。(C) 人类大脑细胞核的迭代聚类和注释。依次使用t-SNE可视化整个mC数据集、抑制性/非前脑神经元细胞类以及SubCtx-Cplx主要类型的细胞,按照相应迭代中注释的细胞组别进行着色。(D) 主要类型的稳健树状图以及亚型数量、大脑结构和捐赠者来源的元信息。
颜色调色板在整个研究中都是共享的。(E) 主要类型SubCtx-Cplx的兴奋性和抑制性标记物(SLC17A1和GAD1)的CH甲基化。(F) 人类大脑细胞按解剖区域着色。(G) 由snm3C-seq分析的大脑核的二维可视化。(H) 不同大脑细胞类型中全局CG和CH甲基化的变化。(I) 主要类型之间全局DNA甲基化与MECP2和DNMT1基因表达之间的相关性。
【神经元和非神经元之间的接触距离差异】
研究分析了不同细胞类型之间在基因组不同尺度上的染色质相互作用。结果显示,神经元在较短距离(200 kb至2 Mb)上的相互作用丰富,而成熟的少突胶质细胞和非神经元细胞在较长距离(20 Mb至50 Mb)上的相互作用丰富。星形胶质细胞和少突胶质前体细胞则在两个范围内均表现出相互作用丰富。在神经元内部,大脑皮层的兴奋性神经元和皮层抑制性神经元相比,具有更多的较短距离相互作用。研究还观察到这些模式在小鼠的前期数据和不同技术的数据中也存在,表明这些模式在细胞间保守。与细胞的全局基因表达活动密切相关,并与细胞核的大小一致。研究进一步探讨了这些相互作用与染色质区域和结构的关系,发现不同细胞类型中的染色质相互作用模式存在差异,这可以提供关于细胞类型特异性基因组折叠的重要见解。
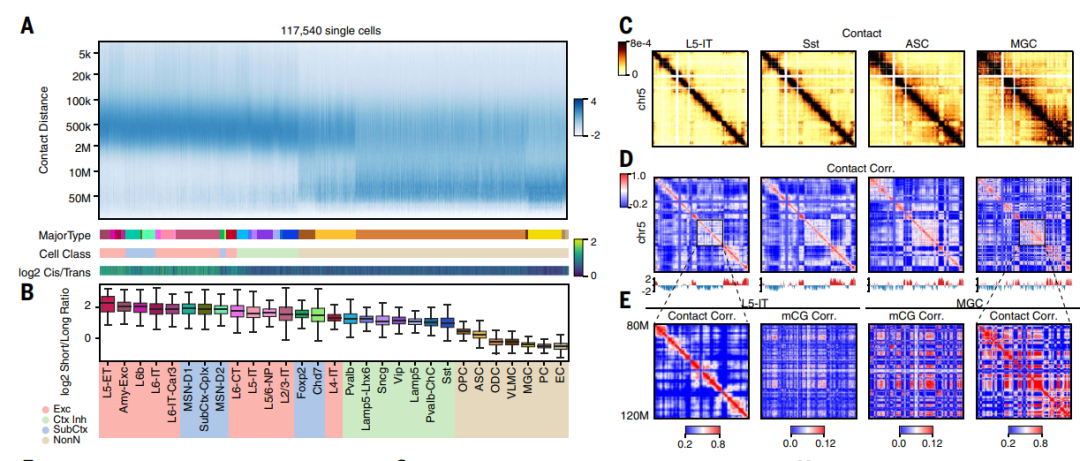
图2. 主要类型之间的3D基因组结构多样性。(A) 在每个单细胞中的基因组距离下的接触频率,在每个细胞内进行Z分数归一化(列)。细胞按主要类型分组,然后按细胞上的中位数log2短/长比例排序。y轴按log2比例划分。(B) 主要类型的log2短/长比例,按照与(A)相同的顺序排序。(C) 四个主要类型的预测接触图。(D) 显示(C)中距离归一化接触图的相关矩阵的热图,线图显示相关矩阵的PC1。(E) 放大(D)中两个矩阵以及跨细胞的mCG相关矩阵。(F和G) FOXP2位点上兴奋性IT神经元(L4-IT细胞类型的标志物)
【大脑细胞类型中的区域、区域和环路】
研究表明,基因组中不同位点的甲基化状态会协同变化,如果它们在物理上靠近的话。甲基化相关系数矩阵显示了跨单个细胞的基因组区域之间甲基化的相关性,呈现出与染色质互联系结构相似的格状模式。这暗示基因组被分隔成具有相反甲基化多样性的局部共甲基化区域,这两组区域之间存在相互作用。研究还观察到DNA甲基化和三维基因组结构之间的相关性,以及染色体区室的关联。研究还对单细胞的染色质域进行了详细研究,结果发现神经元具有更多的染色质域,但其平均大小较小,导致了染色质域所覆盖的基因组比例相似。研究还探讨了染色质域边界的概率和与三维基因组互作的相关性。此外,研究还描述了单细胞中的染色质环的发现,这些环通常是远程甲基化区域(DMRs)与基因启动子之间的相互作用。
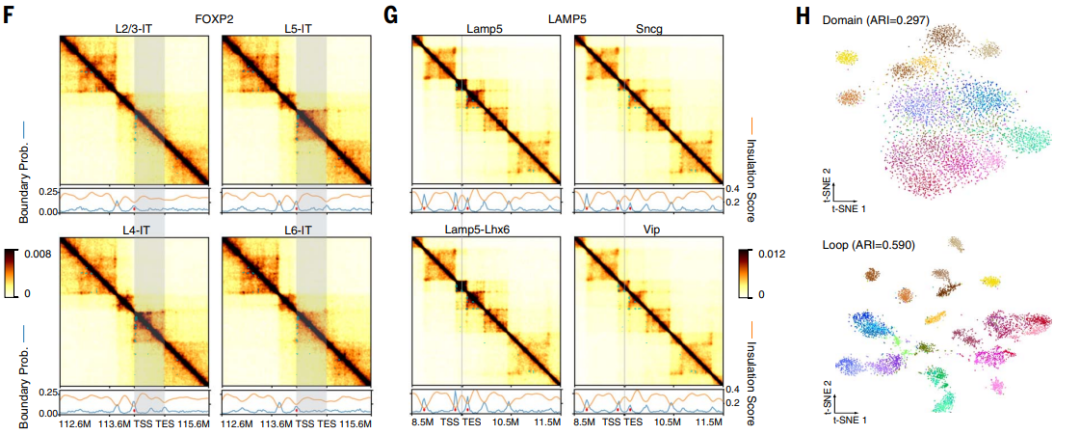
图2. 主要类型之间的3D基因组结构多样性。(A) 在每个单细胞中的基因组距离下的接触频率,在每个细胞内进行Z分数归一化(列)。细胞按主要类型分组,然后按细胞上的中位数log2短/长比例排序。y轴按log2比例划分。(B) 主要类型的log2短/长比例,按照与(A)相同的顺序排序。(C) 四个主要类型的预测接触图。(D) 显示(C)中距离归一化接触图的相关矩阵的热图,线图显示相关矩阵的PC1。(E) 放大(D)中两个矩阵以及跨细胞的mCG相关矩阵。(F和G) FOXP2位点上兴奋性IT神经元(L4-IT细胞类型的标志物)(F)或LAMP5位点上的CGE源抑制性神经元(Lamp5和Lamp5-Lhx6的标志物)(G)的预测接触矩阵(热图),边界概率(蓝线),绝缘分数(橙线),不同边界(线图中的红点)以及不同环路(热图中的青点)。灰色阴影表示基因本体(TSS到TES)。(H) 使用区域(上)或环路(下)作为特征的细胞(n = 5707)的t-SNE图,按主要类型着色。
【3D基因组特征的细胞类型特异性】
这一部分讨论了在不同细胞类型中的三维基因组特征的特异性。通过使用染色体区室评分、域边界概率和环强度,研究人员能够确定不同细胞类型之间的差异,并表明这些三维结构在不同细胞类型之间具有特异性。具体来说,染色体区室可以区分不同类型的神经元和非神经元,但对于更具体的细胞类型区分能力较差。相比之下,染色质域和环对于细化的细胞类型区分更为有效,其中环表现最佳。这强调了不同尺度的三维特征在不同细胞类型之间的基因调控中的不同作用。不同的三维特征具有不同的特异性。染色质区室主要用于区分神经元和非神经元,而染色质域和环更适用于区分更细化的细胞类型。
【基因组组织与其他分子模态之间的关系】
在神经细胞类型中,mCG和mCH与染色体区室评分、域边界概率和环强度呈反相关,而开放染色质信号则呈正相关。这些关系表明了活跃染色体区室、强结构域和环互相协调,同时伴随着开放染色质和DNA甲基化降低,对应于活跃的染色质状态。在细胞类型间的不同三维结构特征(DCs、DBs和DLs)之间,DLs与mCG、mCH和开放染色质的(反)相关性更强。基因表达与三维基因组结构之间也存在关联,尤其是对于细胞类型特异的基因。
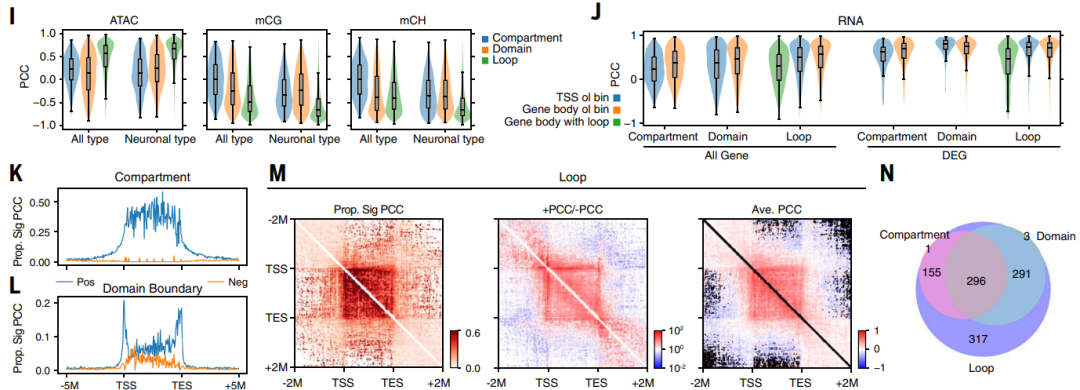
图2. 主要类型之间的3D基因组结构多样性。(I) 跨所有主要类型的所有基因(左)或仅顶部差异表达基因(右)的区域分数、边界概率或环路相互作用强度与ATAC信号、mCG和mCH分数之间的PCC。(J) 不同重叠类别(x轴)下,不同主要类型的所有基因(左)或顶部DEG(右)之间的区域分数、边界概率或环路相互作用强度与基因表达之间的PCC。(K和L) 正相或负相相关区域(K)或区域边界(L)在相对于基因的不同位置的所有bin中所占的比例,平均值在顶部神经DEG中。(M) 在相对于基因的不同位置的所有环路像素中,显着相关的环路像素(左)、正负相关的环路像素比率(中),或显着相关的环路像素的平均PCC(右)所占比例,平均值在顶部神经DEG中。(N) 在顶部神经DEG中,有显着正相关区域、与基因本体重叠的区域边界或基因本体内的环路像素,或者至少有一个锚点与基因的TSS或TES重叠的基因的数量。其中,35个基因没有包含在这三个圆圈中。
【细胞类型特异的DNA甲基化模式和相关的基因调控网络】
研究中鉴定了不同的CH-和CG-差异甲基化基因(DMGs)以及CG-差异甲基化区域(DMRs),总共覆盖了188个脑细胞亚型。这些差异甲基化模式不仅展示了大脑细胞的不同表观遗传特征,还为理解大脑细胞中的基因调控程序提供了重要的见解。这些研究还将转录因子(TFs)分配给特定的细胞类型,如果它们是CH-DMGs并且其模体在相同细胞类型中的CH-DMRs中富集,共有612个TFs分配给主要的神经细胞类型和亚型,其中它们可能在塑造和维持细胞特性方面起重要作用。将DMGs、DMRs和差异环路整合起来,可以确定每个细胞类型的潜在CREs(启动子区域)。
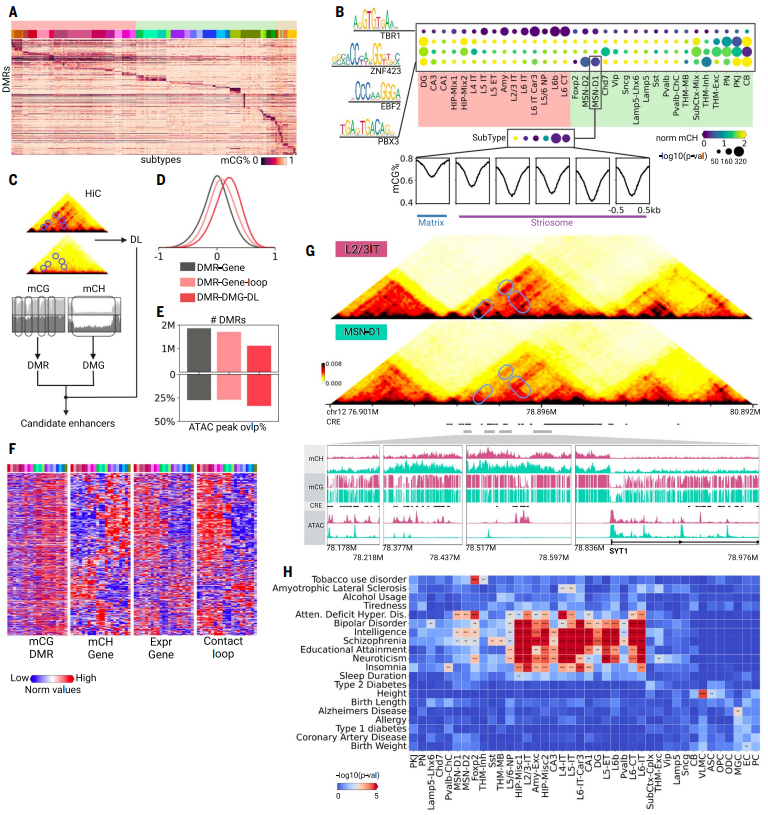
图3. 大脑细胞中的基因调控。(A) 在188个细胞亚型中,细胞类型特异的DMR(差异甲基化区域)的mCG。(B) CH-低甲基化的转录因子(TFs)以及它们的富集在CG低甲基化DMRs中的结构。底部面板显示了TF PBX3在其潜在结合位点上整个基因组的平均甲基化分数。(C) 确定潜在CREs(增强子、调节元素)的工作流程。(D) 来自不同筛选的潜在CREs的甲基化与相应基因之间的相关性分布。(E) 不同筛选条件下的潜在CREs数量和与开放染色质区域的重叠比例。(F) 热图显示潜在CREs的mCG、mCH,以及目标基因的表达,以及相应环路的接触强度。(G) L2/3-IT和MSN-D1主要类型中SYT1基因周围的基因本体mCH、DMR mCG和3D染色质结构。(H) 显示与不同指示性状或疾病相关的变异体在从主要人类细胞类型中鉴定的DMR中的LDSC分析结果的热图。
【大脑皮层和基底神经核中的区域异质性】
研究发现,除了细胞类型多样性外,在大脑皮层的更广泛区域和亚皮层区域之间存在明显的DNA甲基化差异。特别是,皮层兴奋性神经元表现出显著的区域差异,而皮层抑制性神经元也显示出一定程度的区域差异。基底神经节神经元也表现出显著的区域多样性。通过单细胞轨迹分析,研究人员构建了皮层和基底神经节细胞类型的区域轴,揭示了区域DNA甲基化效应。这些区域差异可能与大脑功能和发育中的区域化特异性相关。同时,研究还发现大量的转录因子(TFs)的结合位点富集在区域甲基化区域,这些TFs可能在不同脑区的细胞类型中调控特定的基因表达。
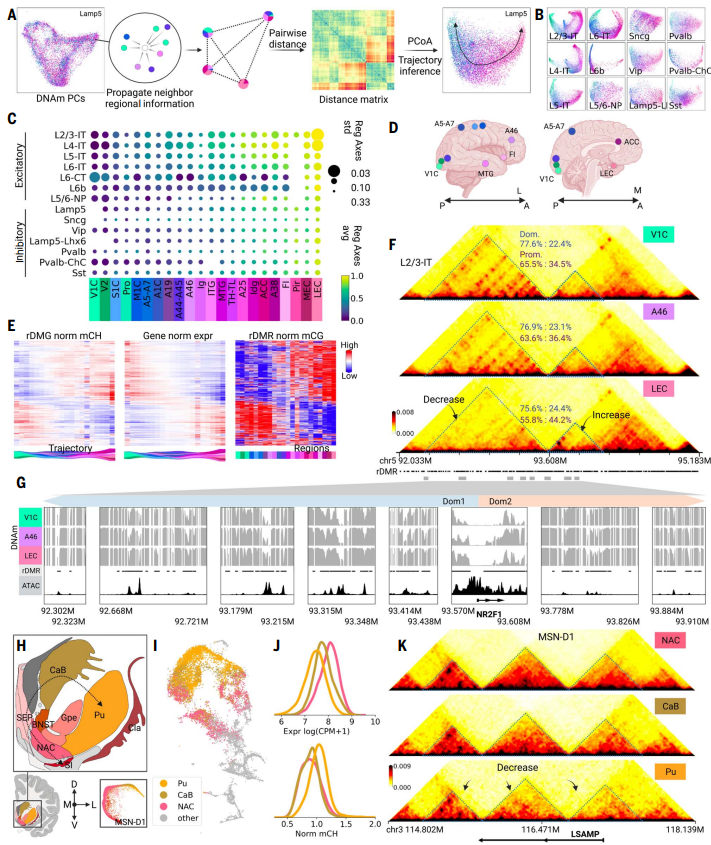
图4. 大脑皮层和亚皮层细胞的区域轴。(A) 从单细胞DNAm(DNA甲基化)确定区域轴的工作流程。(B) 皮层神经元在区域空间中的二维可视化,按解剖位置着色。(C) 皮层神经元中的共同区域轴。散点图显示区域指数在每个皮层区域中的变化。(D) 示例皮层解剖位置的示意图。(E) L2/3-IT细胞中rDMRs(区域特异性差异甲基化区域)的mCG、rDMGs(区域特异性差异甲基化基因)的mCH和表达中的区域梯度。(F) NR2F1周围的染色质构象中的区域差异。蓝色和紫色数字分别表示每个区域的相对区域强度和启动子强度。(G) 放大示例区域差异环路重叠的rDMRs,标记在(F)中。在减少的区域(左侧),甲基化分数从V1C到A46再到LEC增加,而在增加的区域(右侧),甲基化分数减少。(H) 基底神经核中的抑制性神经元显示DNA甲基化的侧向-背侧-腹侧轴。(I) MSN-D1的2D t-SNE可视化。来自NAC、CaB和Pu的细胞被突出显示。(J) MSN-D1中基因本体mCH甲基化和LSAMP表达的区域差异。(K) LSAMP周围的染色质构象中的区域差异,位于MSN-D1中。
【人类和小鼠之间的大脑细胞类型和DMRs的保守性】
研究人员比较了人类和小鼠的单细胞DNA甲基化数据,发现在更广泛的脑区域中,细胞类型的分类在人类和小鼠之间存在差异。虽然某些细胞类型在两个物种中没有直接对应,但总体上,在不同脑区域之间,人类和小鼠的细胞类型分类是相对保守的。然而,人类的DNA甲基化水平(包括CG和CH甲基化)在相应的细胞类型中普遍高于小鼠。研究还通过将人类和小鼠的甲基化区域进行比对,找到了一些高度相关的区域,这些区域在两个物种中可能具有功能上的保守性,特别是与前脑发育和突触相关的生物过程。
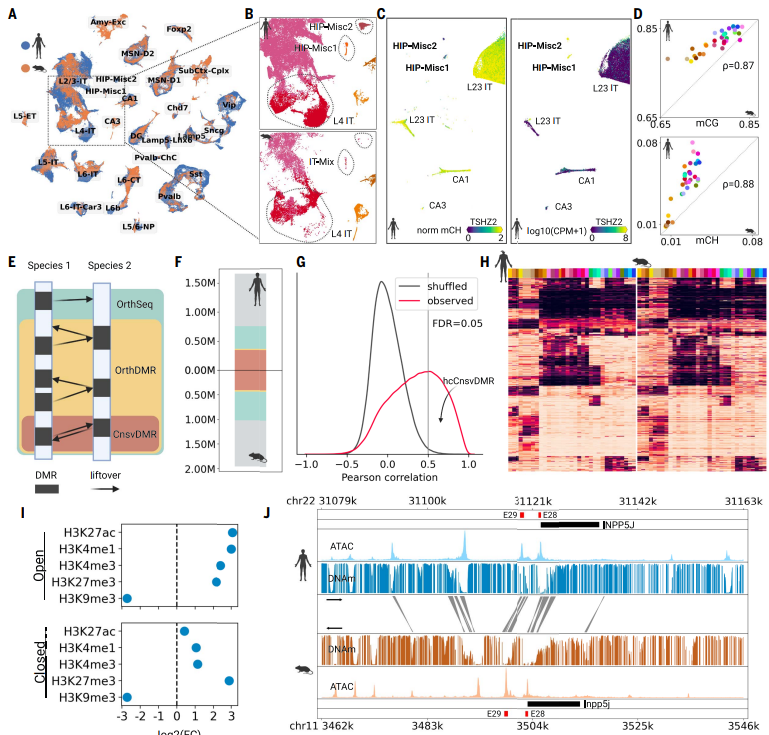
图5. 人类和小鼠大脑细胞甲基组之间的跨物种比较。(A) 用2D t-SNE可视化的人类和小鼠大脑之间的单细胞甲基组整合。(B) 人类和小鼠大脑中细胞类型L4-IT、HIP-Misc1和HIP-Misc2之间的细胞类型差异。(C) 细胞类型HIP-Misc1和HIP-Misc2中CH低甲基化和TF TSHZ2的基因表达。(D) 人类和小鼠之间保守细胞类型的全局mCH和mCG之间的相关性。(E) 细胞类型DMRs的跨物种匹配示意图。(F) 总体上,约50%的DMRs在另一物种中有同源序列,其中约25%是相互的DMRs。(G) DMR甲基化的跨物种相关性分布(红色)和随机重排的背景分布(黑色)。(H) hcCnsvDMRs甲基化分数的示例。(I) hcCnsvDMRs在组蛋白修饰标志中的富集。(J) 在Pvalb主要类型中,hcCnsvDMRs周围基因INPP5J的浏览器视图。
【单细胞甲基化条形码可靠地预测人类大脑细胞身份】
研究人员选择了一些高度特异于大脑细胞类型的CpG位点,然后使用这些位点的DNA甲基化状态来生成scMCodes。这些scMCodes在机器学习模型中表现出很高的细胞类型预测准确性,大约为93%。研究还进行了跨个体的测试,结果显示scMCodes方法具有跨个体的鲁棒性,可在不同个体之间实现高预测准确性,约为92%至96%。尽管单细胞测序具有基因组覆盖范围有限,但平均每个细胞只能检测到大约200个scMCodes中的CpG位点,但这些方法仍然非常有效,可使用数百个选定的甲基化位点来准确识别人类大脑细胞类型。
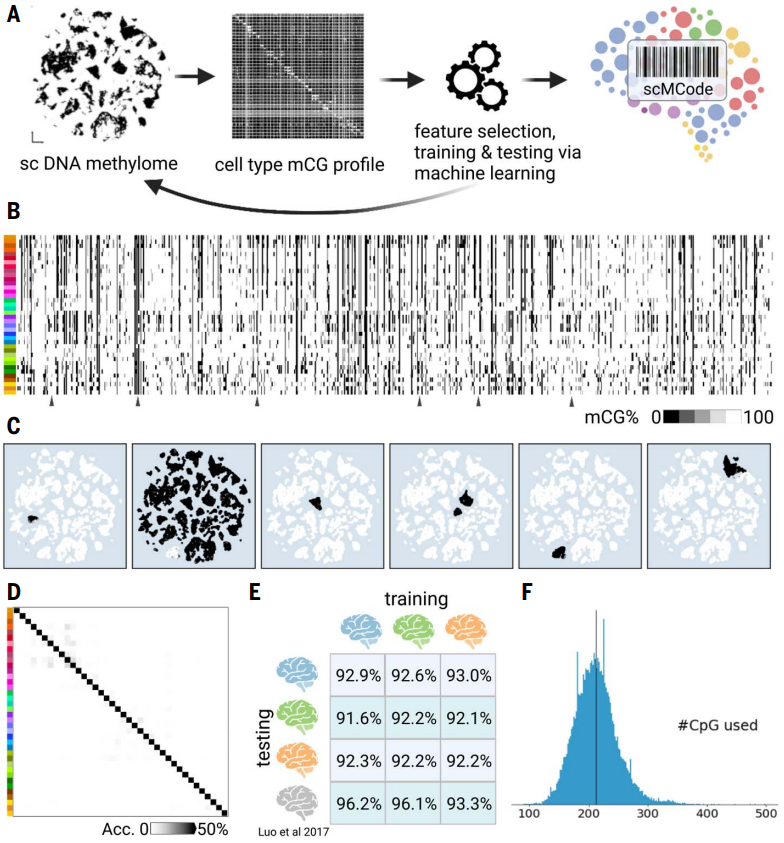
图6. 用于大脑细胞类型的snMCodes。(A) 获得snMCodes的工作流程。(B) 从三位捐赠者获得的snMCodes。(C) snMCode特征的细胞类型特异性示例。(D) 显示预测细胞类型的snMCodes的混淆矩阵的热图。(E) 跨捐赠者测试中的细胞类型预测准确性。(F) snMCodes以单细胞分辨率的有限CpG位点预测人类细胞类型。
【小结】
通过分析表观遗传特征,可以对大脑细胞进行分类,发现神经元和非神经元之间的染色质接触距离差异,并了解大脑细胞的三维基因组结构。此外,研究还揭示了不同细胞类型的三维基因组特征以及DNA甲基化模式的细胞特异性。研究还发现了不同大脑区域之间的差异,并在人类和小鼠之间比较了大脑细胞类型和差异甲基化区域。最后,研究还提到了一种新的方法,称为单细胞甲基化条形码(scMCodes),用于精确识别人类大脑细胞类型。总之,这项研究为我们提供了关于人类大脑细胞的DNA甲基化和三维基因组结构的详细图谱,有望成为有价值的资源,推动我们对大脑细胞多样性、基因调控机制和新遗传工具的研究。
原文链接:
https://www.science.org/doi/full/10.1126/science.adf5357


