Genome Medicine:刘琦团队提出单细胞组学驱动的泛癌肿瘤个体化组合用药的AI计算解决方案
时间:2023-12-03 15:35:48 热度:37.1℃ 作者:网络
精准医学作为肿瘤个体化治疗的未来发展趋势,强调根据每位肿瘤患者的个体化组学特征制定合适的治疗方案。如何在肿瘤的临床治疗中推进精准医学和个体化治疗具有重要的意义,但存在巨大挑战。领域内已有大量计算模型关注于肿瘤药物治疗效果的有效预测,但其预测建模大多基于体外的细胞系药敏数据或通过量化肿瘤样本的整体转录组测序信息(Bulk-seq)来获得患者肿瘤样本的平均转录学特征进而进行药效预测。然而,癌症作为一种高度异质性的复杂疾病,不同患者间的癌细胞、乃至同一患者体内的癌细胞间,均存在显著差异。故细胞系或者Bulk组学均无法体现肿瘤的组织异质性。
此外,肿瘤微环境中肿瘤细胞和免疫细胞的相互作用对于癌症治疗的效果也具有重要影响,这些特征均无法通过细胞系实验或者Bulk组学来进行模拟,而研究中常用的模式动物模型如PDX模型等在模拟肿瘤免疫微环境层面差强人意。另外,组合用药具有高协同作用、低毒性,有望解决单药治疗的耐药问题,但如何从海量的药物组合空间中筛选有效的组合难度巨大。综上,肿瘤的这种耐药性、异质性和免疫微环境的复杂性,使得肿瘤精准医学在临床应用中面临巨大挑战(图1)。
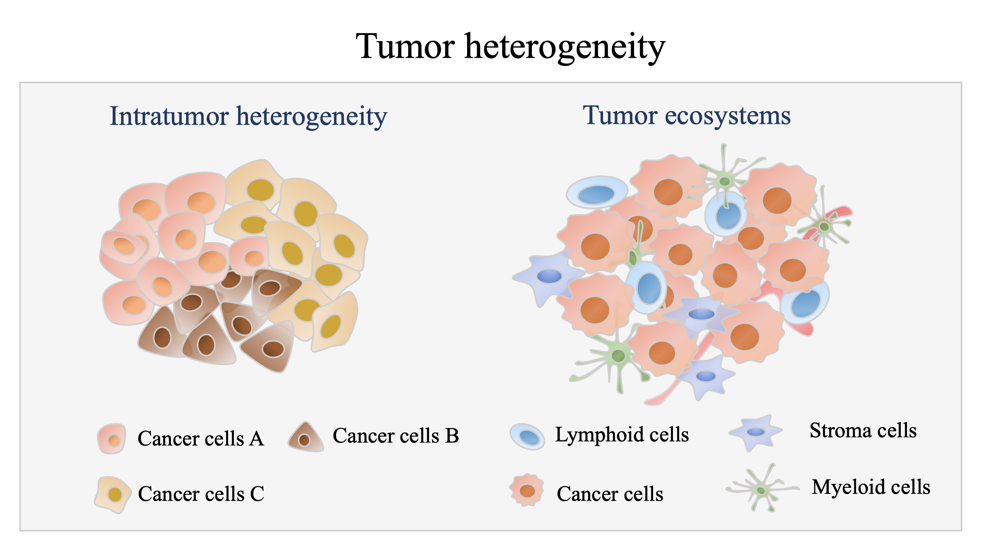
图1. 肿瘤的异质性和免疫微环境的复杂性
近日,同济大学生命科学与技术学院生物信息学系、同济大学附属第十人民医院、同济大学附属肺科医院、同济大学-上海自主智能无人系统科学中心等团队联合攻关,在Genome Medicine期刊上发表了题为“Personalized tumor combination therapy optimization using the single-cell transcriptome”的研究论文,首次提出了一种面向泛癌种、具有普适性、可拓展性的单细胞组学数据驱动的肿瘤个体化治疗的AI计算解决方案和计算框架comboSC,尝试克服上述肿瘤精准医学中面临的挑战。该计算框架基于单细胞RNA测序进行个性化肿瘤组合治疗方案的优化和推荐,旨在最大限度地发挥已知的体外细胞系药敏数据在单细胞水平的转化潜力,识别协同的药物小分子组合或可与免疫检查点抑制剂配对以增强免疫疗法疗效的小分子组合。
具体来说,comboSC首先通过单细胞RNA测序数据定量评估患者个体的肿瘤微环境并进行精准分型,再基于肿瘤细胞和免疫细胞的转录组特征,采用图组合优化的算法优化免疫治疗和靶向治疗的组合。该研究将comboSC应用于包括实体瘤和血液瘤在内的15种癌症类型的119个肿瘤样本,证明了其在不同癌症类型中进行组合治疗优化的广泛实用性和性能优越性,从而为肿瘤临床的精准治疗提供了一个具有可借鉴和潜在临床应用价值的创新性计算平台,为如何发挥单细胞组学在精准医学领域的转化价值提供了有益的借鉴。为了便于推广comboSC在临床和研究领域的应用,研究团队开发了一个一站式的交互式网站(http://www.combosc.top),用户可以通过上传患者肿瘤的单细胞RNA测序数据快速使用comboSC计算平台进行可能的个体化组合用药识别。

文章发表在Genome Medicine
comboSC创新性的提出单细胞转录组学驱动的泛癌种的个体化组合治疗方案推荐和治疗反应预测的计算框架。需要特别指出的是,comboSC旨在通过单细胞组学对于肿瘤体内的免疫微环境进行整体刻画,并给出in-vivo场景下的肿瘤个体化组合用药的优化方案,最大程度的发挥了单细胞组学对于肿瘤异质性刻画的能力,有别于领域内常见的基于细胞系(in-vitro),基于PDX小鼠模型或者是仅面向特定细胞类型的治疗方案推荐。
该计算平台具有以下若干特点:(1)comboSC基于单细胞转录组学对于肿瘤免疫微环境进行定量评估和精准分型,通过定义一种T cell resilience (Tres) 驱动的打分函数对于病人的免疫微环境进行评估,该打分函数基于免疫抑制信号和T细胞增殖信号来对免疫治疗反应进行预测。根据Tres的评分,comboSC将病人分成高,中,低三个免疫评分等级:对于高免疫得分的样本,因为样本的免疫功能得到了充分激活,comboSC推荐采用ICI(Immune Checkpoint Inhibitor)单药治疗的方案;对于中等评分的样本,comboSC推荐ICI与某些小分子/药物的组合策略,以增强样本对于ICI治疗的应答;对于低免疫得分且对免疫治疗反应潜力有限的样本,comboSC倾向于推荐直接以小分子/药物组合策略清除肿瘤恶性细胞。(2)comboSC定义了一种图组合优化驱动的AI计算框架。通过二部图模型推断候选药物与病人细胞异质性群体间的相互关系,为中、低免疫评分的患者优化组合用药方案(图3)。二部图模型能够在细胞种群规模、发育轨迹、药物协同作用、副作用和毒性等多重信息和约束下,为肿瘤细胞与免疫细胞群的多样分子细胞表型识别出简约(Parsimony)且全面(Complete)的药物组合覆盖。

图2. comboSC的整体计算框架
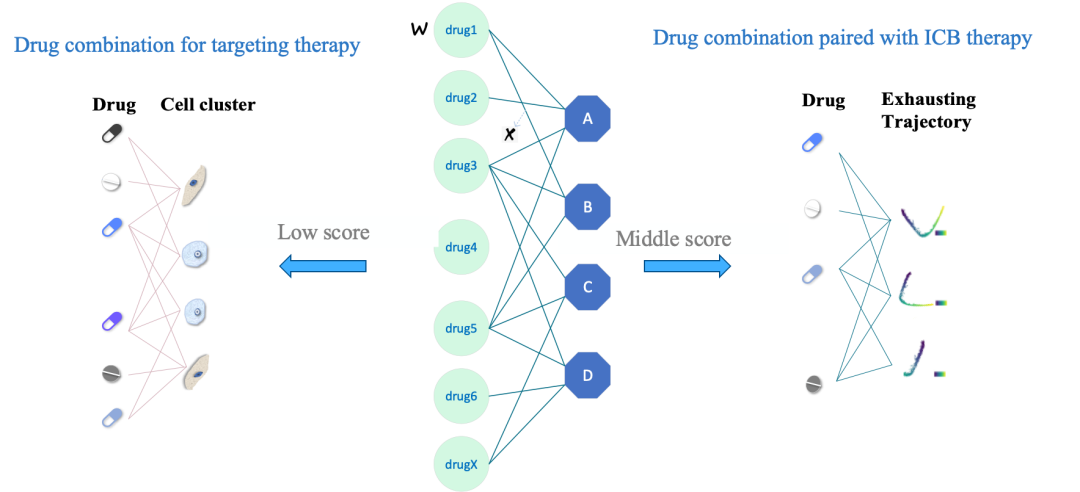
图3. 中低免疫评分样本中药物与细胞簇关系的二部图建模
目前,该版本comboSC支持15种癌症类型,并在119个肿瘤样本单细胞转录组样本中得到应用。结果表明,comboSC能以较高的AUC值(0.76)对于样本进行免疫评分(高,中,低三级)。此外,通过文献、临床试验数据挖掘、公开的细胞系数据以及具有预后标签的单细胞测序样本等多维度的系统性验证,证明了comboSC对中、低免疫得分患者药物组合推荐的初步可靠性。例如,在公开报道的某一例肾细胞癌患者(RCC)临床案例中,comboSC预测患者免疫得分为中级,并在没有任何先验信息的前提下,成功预测了临床上报道对于该病人有效的组合治疗方案(伊马替尼/imatinib联合ICI治疗);在公开报道的另外一例非小细胞肺癌患者(NSCLC)的临床案例中,comboSC评估患者免疫得分为低级,并预测帕比司他(panobinostat)和吉西他滨(gemcitabine)对于该病人具有良好的药物协同效应,而多项临床前研究均已证实这一组合可以增强抗肿瘤作用。
综上所述,comboSC创新性的提出了一种单细胞组学驱动的泛癌种肿瘤个体化组合用药的计算框架和解决方案,在单细胞组学驱动的精准医学领域进行了有益的探索,其可行性和有效性经过了Proof-of-Concept验证。该计算框架致力于进行组学数据驱动的AI建模和药物组合计算识别,缩短庞大药物组合的筛选空间,从而使得个性化肿瘤治疗过程更为高效和有效。通过配合PDX等药物筛选实验模型,将有望大大缩短肿瘤病人个体化试药的成本和时间,为肿瘤患者的精准治疗提供计算框架层面的有效支撑。
同济大学刘琦教授课题组长期致力于发展人工智能和生物组学交叉融合的研究范式(AI for Omics),以组学人工智能赋能复杂生命系统的智能解析和精准干预,进行数据驱动的精准医学研究和转化。近年来关注的研究方向之一是基于人工智能及高通量组学挖掘的肿瘤个体化治疗和药物发现。此次工作是刘琦教授课题组联合同济大学附属第十人民医院,同济大学附属上海市肺科医院在肿瘤精准治疗领域的重要探索,该论文第一作者是同济大学生命科学与技术学院的博士生汤晨和博士后傅沙镠,通讯作者是同济大学生命科学与技术学院的刘琦教授,同济大学附属第十人民医院的王平教授和同济大学附属上海市肺科医院的何雅億教授。
论文链接:
https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-023-01256-6


