Commun Biol:基于表观遗传因子表达的泛癌AI模型可准确预测多种癌症类型患者预后
时间:2023-12-07 16:38:40 热度:37.1℃ 作者:网络
表观遗传是指在基因的DNA序列没有发生改变的情况下,基因功能发生了可遗传的变化,并最终导致了表型的变化。表观遗传过程包括DNA甲基化、组蛋白修饰、染色质可及性和高阶染色质结构的变化,在基因的表达调控中发挥重要作用。这些状态的改变由染色质相关蛋白因子(表观遗传因子或表观因子)介导,这些因子可以添加、移除和读取DNA和组蛋白修饰,并重塑染色质。
癌症历来被认为是一种遗传性疾病,其致癌基因或抑癌基因中存在驱动突变。近年来,NGS技术的出现使人们认识到基因突变和表观遗传变化可以协同作用于癌症的发展。表观遗传因子本身可能在肿瘤中发生基因改变,从而导致广泛的表观遗传失调;在表观遗传因子基因没有突变的情况下,癌细胞也会发生表观遗传变化。此外,癌细胞中表观遗传因子基因的异常表达或抑制可导致表观遗传变化,进而导致癌症生长。
近日,美国加州大学的研究人员在Communications Biology上发表了题为“Pan-cancer landscape of epigenetic factor expression predicts tumor outcome”的文章。研究团队根据表观遗传因子(epifactors)的基因表达模式对24种TCGA成人癌症类型中的原发性肿瘤进行分组,与癌症分级和分期等传统方法相比,这些分组能够更好地预测各种癌症类型患者的预后。基于五种癌症类型的表观遗传因子表达数据,研究团队开发了一个泛癌机器学习模型,可准确预测患者预后。总之,该研究提供了癌症类型的表观遗传图谱,为发现靶向泛癌的表观遗传因子奠定了基础。

文章发表在Communications Biology
研究团队分析了TCGA数据库中24种成人癌症类型,并根据来自表观遗传因子数据库中720个表观遗传因子基因的表达模式,将这些肿瘤划分为不同亚群;多数表观遗传因子基因(556个)在癌症组织中未发生遗传改变(图1)。使用非负矩阵分解(NMF)算法,研究人员基于患者肿瘤中表达变化最大的表观遗传因子基因,对每种癌症类型患者的肿瘤进行聚类;对24种癌症类型进行独立评估,将肿瘤分成两个分离良好的聚类,每种癌症类型的两个聚类由一组具有不同表达模式的标志性顶级NMF基因表征。
结果显示,在上述24种癌症类型中,顶级NMF基因的数量从低级别胶质瘤(LGG)的76个到结直肠癌(CRC)的9个不等,中位数为43个基因;基于顶级NMF基因表达模式的泛癌图谱显示,肿瘤主要依据其起源组织聚类,并在一定程度上也取决于组织相似性或邻近性;在部分癌症类型中,顶级NMF基因之间存在高度重叠。
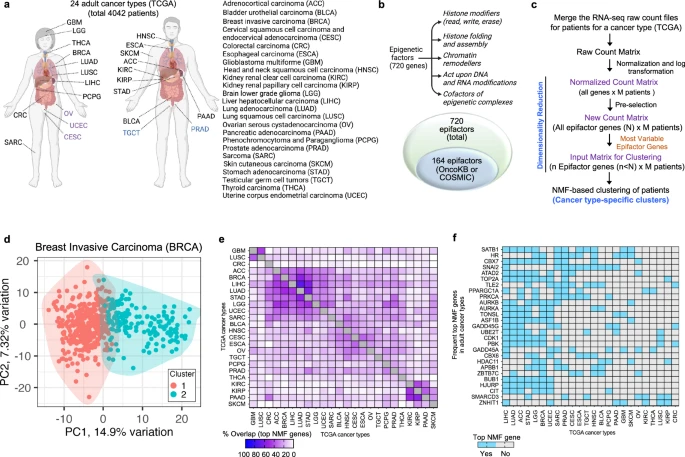
图1. 表观遗传因子的表达水平在24种TCGA癌症类型中形成两个不同的簇。
为确定表观遗传因子的表达水平是否会影响患者的临床结果,研究团队比较了不同癌症类型中两组患者的无进展期(PFI)、疾病特异性生存(DSS)和总生存期(OS)(图2)。结果显示,有10种癌症类型的聚类在至少一个指标的临床结果中存在显著差异,即肾上腺皮质癌(ACC)、结直肠癌(CRC)、肾透明细胞癌(KIRC)、肾乳头状细胞癌(KIRP)、LGG、肝癌(LIHC)、肺腺癌(LUAD)、前列腺癌(PRAD)、胃癌(STAD)和子宫内膜癌(UCEC)。
其中,5种癌症类型(ACC、KIRC、LGG、LIHC和LUAD)的聚类,在三个指标的临床结果上均存在显著差异;预后较差的肿瘤聚类往往具有较高的癌症分期、较大的肿瘤体积或更严重的扩散指标。此外,与肿瘤分级或上皮细胞-间充质转化(EMT)相比,基于表观遗传因子表达水平对这5种癌症类型进行肿瘤聚类的预后效果更好。
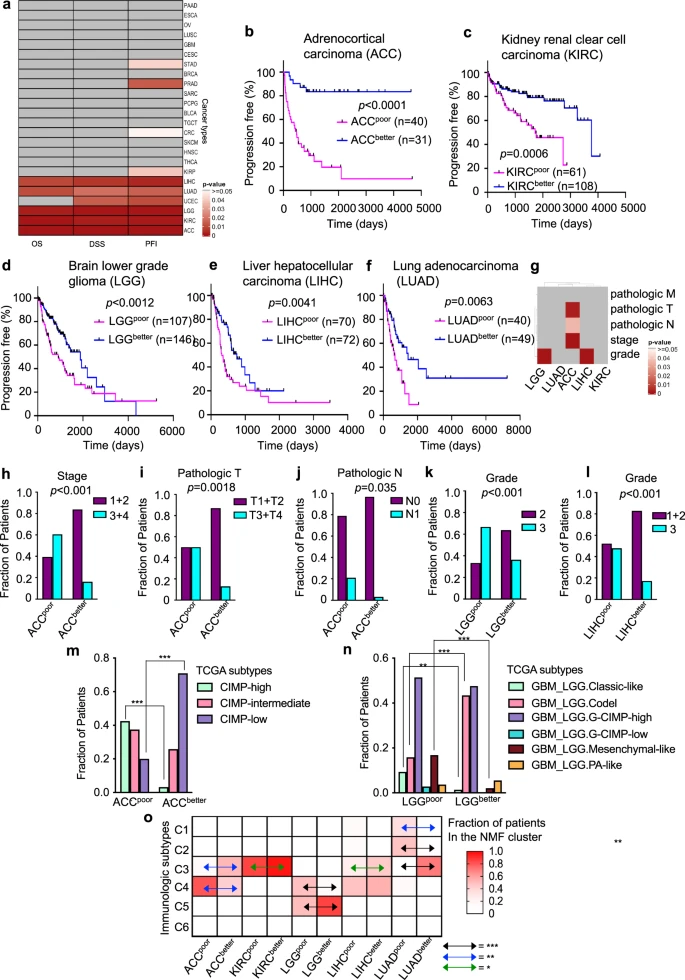
图2. 5种TCGA癌症类型基于表观遗传因子表达的肿瘤集群与临床结果密切相关。
研究团队对肿瘤进行加权相关网络分析(WGCNA),以识别与具有类似表达模式的基因组(模块)相关联的GO terms,这些模块与不良或较好预后聚类的顶级NMF表观遗传因子基因相似(图3)。结果显示,与不良预后聚类相关模块的GO terms富集细胞周期基因和发育基因,表明增殖率或干细胞样特征差异造成了聚类之间的临床差异。在上述5种癌症类型中,与细胞周期相关的模块中顶级NMF表观遗传因子形成的蛋白-蛋白相互作用(PPI)网络显著富集,表明可能存在协同作用机制;不良和较好预后聚类中与特征基因相关的GO terms不同,表明不同肿瘤类型涉及不同的生物学机制。
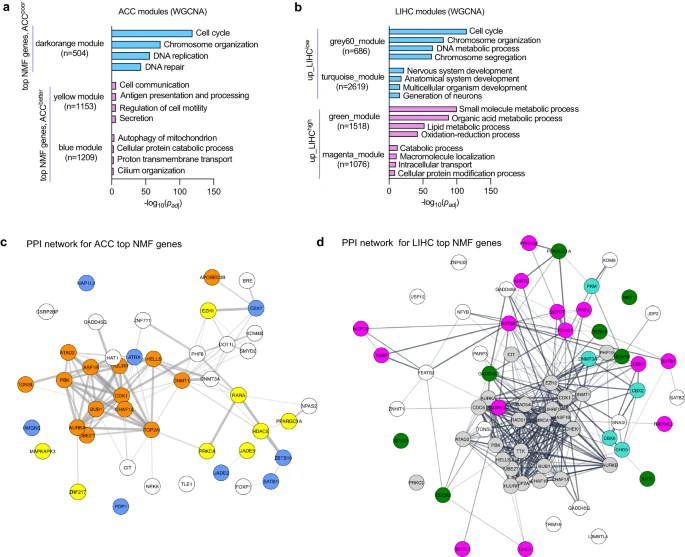
图3. 临床不同簇的顶级NMF基因特征揭示了丰富的生物学功能。
接下来,研究团队对24种癌症类型中每个可变表观遗传因子基因表达水平的预后价值进行了系统分析(图4)。结果显示,排名前十的预后性表观遗传因子基因为DPF1、TOP2A、AURKA、BUB1、CDK1、CHEK1、GSG2、MSH6、SMYD2和USP49,其参与染色质重塑、组蛋白磷酸化、甲基化和去泛素化等过程;预后性表观遗传因子的比例因癌症类型而异,从KIRC的77%到TGCT的0.4%不等,中位数为21%。此外,在不同的癌症类型中,一个基因的预后方向并不总是相同,在肿瘤中的表达水平也并不一致地与突变或拷贝数变异(CNA)相关。
在24种癌症类型中,预后性表观遗传因子和非表观遗传因子的比例高度相关;不同肿瘤类型中具有预后作用的可变表观遗传因子的比例与总突变数或CNA呈弱负相关,但无统计学意义。与其他癌症类型相比,五种癌症组中具有预后意义的顶级NMF基因比例较高;除PPARGC1A外,常见顶级NMF基因的预后方向在五种癌症组中一致。
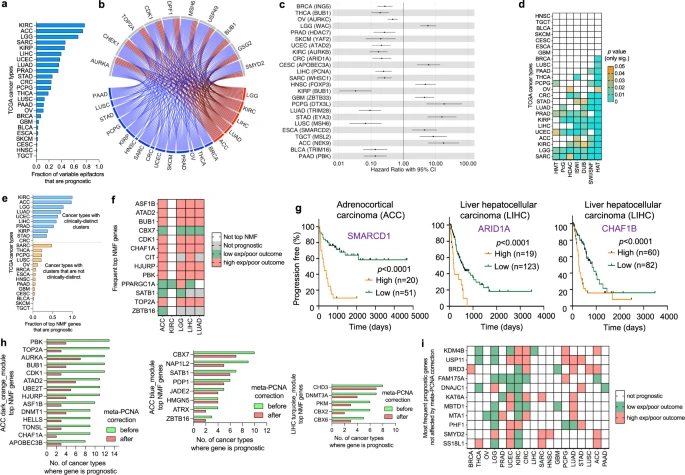
图4. 表观遗传因子基因的预后潜力取决于癌症类型。
为探究泛癌表观遗传特征能否预测患者预后,研究团队利用Cox-nnet人工神经网络(ANN)框架,为五种癌症组开发了一个预测模型,将患者随机分成训练组和测试组,使用患者的年龄和性别、epifactor表达数据对模型进行训练(图5)。结果显示,该模型成功地将患者分为预后较差和预后较好的两组,可高度准确预测患者预后;测试集的高预后指数(PI)组和低PI组的PFI存在显著差异。此外,研究团队还发现对人工智能模型最关键的基因与聚类定义的特征基因有显著重叠。上述结果表明,表观遗传因子的表达水平(共同或单独),具有依据临床结局对肿瘤进行分类的强大能力。
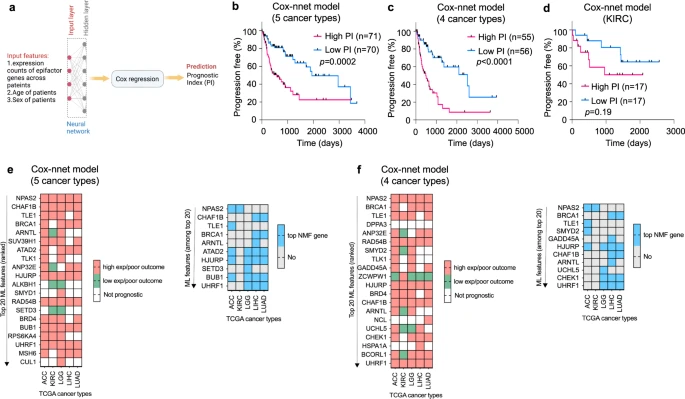
图5. 基于表观遗传因子基因表达模式的泛癌神经网络模型可预测患者预后。
综上所述,驱动癌症进展的致癌途径同时反映了遗传变化和表观遗传调控,研究团队通过比较表观遗传因子的表达,分析了来自24种不同癌症类型的原发性TCGA患者肿瘤的表观遗传异质性;并使用机器学习,为五种癌症组合开发了一个神经网络模型,可以高度预测患者预后。该研究增加了人们对癌症类型之间因起源组织和空间位置而导致的临床差异得理解,以及同一部位肿瘤的表观遗传因素对患者结果的影响。这项泛癌研究的结果可作为针对表观遗传调控因子进行合理药物设计的基础。
参考文献:
Cheng, M.W., Mitra, M. & Coller, H.A. Pan-cancer landscape of epigenetic factor expression predicts tumor outcome. Commun Biol 6, 1138 (2023). https://doi.org/10.1038/s42003-023-05459-w


