Nature Methods:肿瘤微环境精准解析的新途径:CyLinter在多重成像中的应用
时间:2024-11-04 12:00:18 热度:37.1℃ 作者:网络
引言
肿瘤由细胞和非细胞成分构成,其结构的空间尺度从微米到厘米不等。近年来,伴随着高度多重空间分析技术的发展,对这些复杂结构的理解得到了显著推进。基于图像的空间分析方法,使研究人员能够在单个样本中对多达百万数量级的细胞进行百种蛋白质的空间分布和强度的解析。这些方法对于揭示肿瘤微环境中的细胞异质性以及细胞间的复杂相互作用具有重要意义。然而,组织图像不可避免地存在诸如折叠、碎屑、抗体聚集和光学畸变等伪影,这些伪影会严重干扰单细胞数据的质量和生物学解释,导致错误的结论。为了应对这些挑战,10月30日Nature Methods的研究报道“Quality control for single-cell analysis of high-plex tissue profiles using CyLinter”介绍了一种交互式质量控制工具——CyLinter,用于识别并去除图像中的伪影数据,从而显著提高单细胞分析的质量,特别是针对长期保存的存档样本。

高度多重成像技术(例如MxIF、CyCIF、CODEX等)使得研究人员能够在保存的二维和三维组织环境中解析20至100种蛋白质和其他生物分子。与解离方法如单细胞RNA测序(scRNA-seq)相比,这些成像方法能够更好地保留细胞的空间背景信息,尤其在福尔马林固定、石蜡包埋(FFPE)样本中,能够从丰富的存档人类活检样本中提取高价值的数据。这些数据对于理解肿瘤细胞在其微环境中的行为,以及免疫细胞如何渗透和与肿瘤细胞相互作用,具有极高的应用价值。
然而,从高度多重图像生成单细胞数据,需要进行图像分割,得到类似于scRNA-seq中的“空间特征表”。这个分割过程易受到多种伪影的干扰,包括组织折叠、抗体聚集和光学畸变,从而降低数据质量和影响生物学推断的准确性。伪影的存在不仅会导致对某些细胞类型的错误分类,还会掩盖真正具有生物学意义的细胞间相互作用。因此,有效的质控对于保证单细胞水平的数据质量至关重要。
CyLinter工具的开发与模块化应用
为了解决高度多重组织成像中的伪影问题,研究人员开发了一个名为CyLinter的质控工具。CyLinter是一款交互式软件,集成于Python环境下的Napari图像查看器,能够辅助用户识别并去除由显微伪影导致的单细胞数据偏差。CyLinter的目标是通过多种质控手段,尽可能提高数据的准确性,使研究人员能够对肿瘤样本中的细胞特征和空间关系进行可靠的分析。
CyLinter的工作流程包括若干关键模块
区域选择模块(ROI selection):用户可以手动选择图像中的伪影区域,后续分析时这些区域的数据将被去除。对于伪影较多的数据集,CyLinter还提供了正选择模式,允许用户选择没有伪影的区域予以保留。此外,CyLinter集成了自动化算法,能够程序化地标记出潜在的伪影,以供人工审核。通过这种半自动化和手动相结合的质控方式,CyLinter确保用户能够准确标记数据中所有可能影响分析结果的伪影区域。
DNA强度分析模块(dnaIntensity):用于检查每个细胞核的平均强度直方图。强度过低可能表示细胞核被切片截断或未处于焦平面内,强度过高可能表示染色过饱和或组织折叠。用户可以调整阈值来去除这些异常数据。DNA强度的分布对于判断组织的完整性和分割的精确度具有重要参考价值,尤其在样本处理过程中可能会引入变异的情况下。
细胞分割面积过滤模块(dnaArea):用于识别和去除在分割过程中过度或不足分割的细胞。该模块在结直肠癌(CRC)图像中,能够有效地去除过度分割的细胞。面积的异常分布可能由成像伪影、分割错误或样本制备中的问题引起,因此该模块对于保证分割后数据的准确性至关重要。
成像周期相关性模块(cycleCorrelation):在循环成像中,由于组织移动或降解,某些细胞可能会在多轮成像过程中丢失。该模块通过计算第一个和最后一个成像周期之间的DNA强度比值来识别不稳定的细胞。这种周期相关性检查对于多轮成像的数据集尤为重要,因为组织移动或成像质量变化可能导致细胞的信号丢失或偏移,从而影响定量分析。
通道离群值修剪模块(pruneOutliers):用于可视化每个细胞的信号,基于上下百分位数阈值去除残留的伪影(例如小的抗体聚集)。离群值的存在往往代表了染色过程中抗体非特异性结合或样本中的技术性噪音,通过去除这些离群信号,可以显著提高数据的准确性。
这些模块的协同应用,使得CyLinter能够全面地执行质量控制,确保去除伪影后的数据具有更高的可靠性和准确性。每一个模块都可以独立用于特定的质控需求,同时它们的组合使用使得质控过程更为灵活和高效。
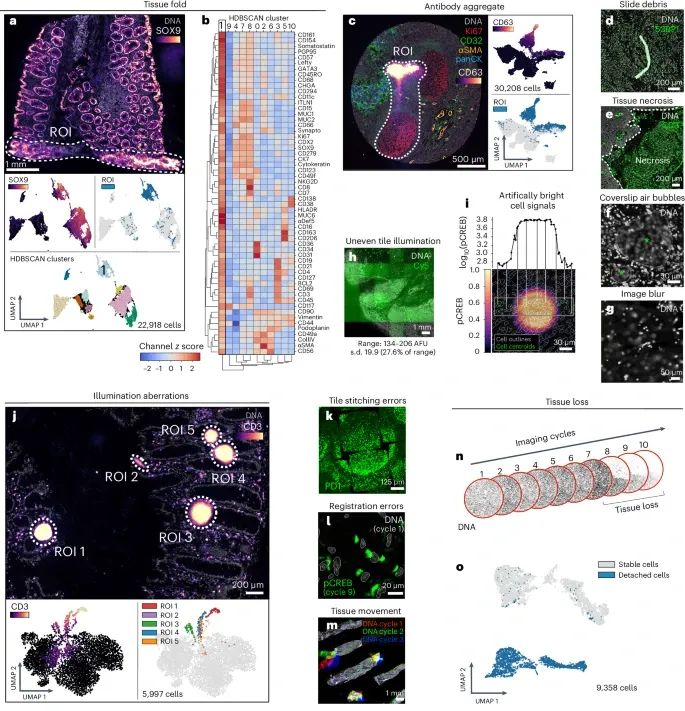
多种影响组织切片免疫荧光图像质量的伪影,这些伪影影响了基于组织衍生的单细胞数据的分析(Credit: Nature Methods)
组织折叠:在大肠(CODEX,标本1)中,标出了含有组织折叠的区域,并显示出受影响的细胞在UMAP嵌入图中的表现,显示了伪影导致的异常细胞聚类。
抗体聚集:在正常扁桃体的CD63通道中观察到抗体聚集,这些聚集导致单细胞数据中形成了伪影影响的簇。
自体荧光纤维:在TOPACIO标本中发现了自体荧光纤维,这种伪影也影响了细胞的检测。
坏死组织区域:在TOPACIO标本中的CD3通道中观察到坏死区域,这些坏死组织的存在导致了单细胞数据中的噪音。
盖玻片气泡:在某些组织切片中,由于盖玻片安装时形成的气泡被观察到,这些气泡也成为图像伪影的一部分。
图像模糊和离焦区域:在TOPACIO标本中观察到的离焦区域,以及在HNSCC标本中观察到的非均匀的瓷砖照明,均导致了图像质量的降低。
CyLinter的应用效果与实证分析
在该研究中,CyLinter被应用于多个数据集,包括来自TOPACIO临床试验的乳腺癌和卵巢癌样本,以及结直肠癌(CRC)样本。通过CyLinter的质控处理,研究人员显著提高了单细胞数据的质量。例如,在CRC数据集中,约23%的细胞被移除,其中绝大部分是由于过度分割导致的伪影。质控后的数据集生成了78个细胞簇,比质控前的22个细胞簇更为细致,表明去除伪影后的数据更能准确地反映生物学特征。
在TOPACIO数据集中,CyLinter去除了约84%的细胞,其中53%的细胞通过正选择模式被去除。此外,约14%的细胞因抗体聚集导致信号过强而被移除,12%的细胞因成像周期中的组织移动而丢失,4%的细胞因分割错误被去除,1%的细胞由于DNA染色过饱和或强度过低而被去除。质控后的数据集包含43个细胞簇,聚类质量的Silhouette评分大部分为正值,表明质控后的数据具有较好的聚类效果。
质控后的TOPACIO数据集展示了显著的细胞簇间差异,肿瘤细胞通常按患者进行分组,而免疫细胞群体则在不同患者之间表现出更大的异质性。通过CyLinter的各个模块,研究人员确认了大多数细胞的形态和标记表达与已知细胞类型的一致性。例如,质控后簇42主要由不含DNA损伤的肿瘤细胞组成,而簇21则包含具有γH2AX标记的DNA损伤肿瘤细胞。这些细胞簇的分离能够为后续分析提供有力的基础,尤其在研究治疗反应和肿瘤进展机制时。
通过CyLinter的过滤模块,研究人员识别并去除了多个数据集中的伪影。例如,在TOPACIO数据集中,FOXP3通道中超过30%的图像瓦片受到了非特异性抗体信号条纹的影响。通过结合手动和自动化的质控过程,成功去除了这些伪影,使得后续的单细胞分析更加可靠。此类质控过程在临床样本的研究中尤为重要,因为样本的异质性和处理步骤中的变化可能导致大量的技术性噪声,从而影响数据的解释和生物学推断。
CyLinter的开发为高度多重组织成像数据的质控提供了一种可靠且高效的方法。通过去除实验和成像过程中的伪影,CyLinter显著提高了单细胞数据的准确性,使研究人员能够更为精准地解读复杂的生物学现象。尤其是在癌症微环境的研究中,高质量的单细胞数据对于理解肿瘤进展、免疫逃逸以及细胞间相互作用至关重要。CyLinter的应用不仅可以改善现有数据集的质量,还为未来的实验设计提供了质控思路和工具支持。
此外,CyLinter为开发自动化伪影检测的深度学习模型提供了理想的训练数据。随着人工智能技术的发展,基于深度学习的伪影检测方法有望实现更高效、更准确的质控过程。未来,研究人员计划将CyLinter质控报告中的伪影库公开,以扩展伪影检测能力,进一步推动该领域的发展。这一举措将为整个研究领域提供宝贵的资源,促进标准化质控流程的建立,并提高多重成像技术的再现性和可靠性。
总体而言,CyLinter的应用显著提升了高度多重组织成像数据的分析精度,为揭示复杂组织结构中的生物学机制,特别是在肿瘤微环境的研究中,提供了重要的技术支持和方法学创新。通过不断改进和扩展,CyLinter将进一步促进单细胞水平的空间生物学研究,助力解析肿瘤和其他复杂组织中的动态变化,为精准医疗提供科学依据。未来,随着更多数据集的质控和伪影库的完善,CyLinter有望成为高度多重成像领域的标准工具,为复杂组织的单细胞研究开辟新的路径。
参考文献
Baker GJ, Novikov E, Zhao Z, Vallius T, Davis JA, Lin JR, Muhlich JL, Mittendorf EA, Santagata S, Guerriero JL, Sorger PK. Quality control for single-cell analysis of high-plex tissue profiles using CyLinter. Nat Methods. 2024 Oct 30. doi: 10.1038/s41592-024-02328-0. Epub ahead of print. PMID: 39478175.


