Genome Biol:大规模食管癌甲基化综合分析揭示细胞类型和癌症特异性表观遗传调控
时间:2023-09-12 17:00:07 热度:37.1℃ 作者:网络
食管癌的发病率居世界第七位,死亡率居世界第六位。食管癌侵袭性强,患者预后差,5年生存率低于20%。食管癌包括两种主要的组织学亚型:鳞状细胞癌(ESCC)和腺癌(EAC),这两种亚型具有不同的临床特征。
在表观遗传学上,已有多项研究报道了食管癌的分子变化,特别是DNA甲基化水平的变化。但这些研究主要集中在基因启动子区域的DNA甲基化分析上,该区域仅占人类基因组中所有CPG位点的6%。已知DNA甲基化在其他非编码区域也发挥重要作用,如增强子、部分甲基化结构域(PMDs)以及重复元件。因此,食管癌的DNA甲基化组有待通过全基因组亚硫酸盐测序(WGBS)等单碱基分辨率方法进行进一步全面表征。
近日,美国Samuel Oschin综合癌症研究所的研究团队与以色列耶路撒冷希伯来大学研究团队合作,对来自两种不同食管癌亚型及其相应的非恶性组织的45例食管样本的WGBS数据进行分析,并开发了一种新的sequence-aware方法来识别大的PMD,揭示了肿瘤样本中PMD的甲基化水平和基因组分布的高度异质性。研究鉴定了与抑制性转录、染色质B区室和高体细胞突变率相关的亚型特异性PMD。此外,利用泛组织单细胞和泛癌症表观基因组数据集,研究团队证明食管癌中的大部分细胞类型特异性PMDs和差异甲基化区域(DMRs),实际上是源自相关细胞类型的其他癌症中共同发生的标记物。该研究结果发表在Genome Biology上,文章题为“Comprehensive analyses of partially methylated domains and differentially methylated regions in esophageal cancer reveal both cell-type- and cancer-specific epigenetic regulation”。

主要研究内容
开发新的PMDs识别方法
虽然已经开发了用于识别PMD的计算工具,但对于具有非常轻微的低甲基化的组织或几乎完全甲基化丢失的肿瘤WGBS样本无法进行跟准确的预测。该研究团队使用深度学习神经网络方法来建立通用序列特征,这些特征几乎完全可以预测人类基因组PMD区域的CpG甲基化丢失或保留。最终,研究团队基于MethylSeekR中使用的Hidden Markov模型(HMM)开发了一种sequence-aware PMD调用方法,命名为MMSeekR(Multi-model PMD SeekR)。数据显示,MMSeekR成功地在所有样本中一致地识别出PMDs,并具有较高的稳定性和一致性。值得注意的是,MMSeekR几乎完全分离了不同的癌症类型。
研究团队将MMSeekR应用于研究用的所有食管样本,通过三种不同方法识别PMDs的主成分分析再次证实MMSeekR优于MethylSeekR和MethPipe,可完全分离EAC和ESCC样本。
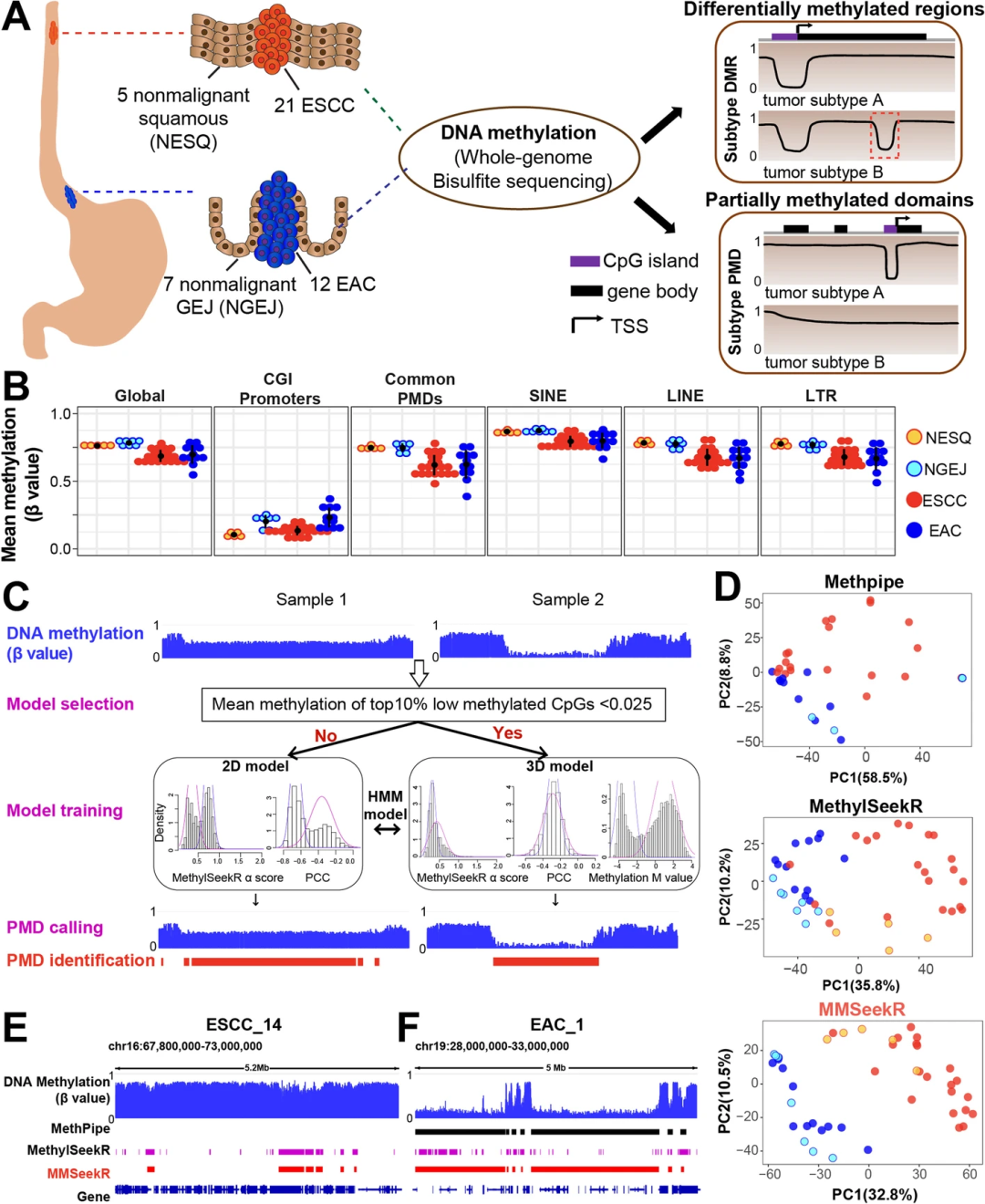
图1.利用MMSeekR识别食管样本中的PMD
食管样本中共有和亚型特异性PMD特征
研究团队逐个对PMDs进行了全基因组注释(图1),发现PMDs在非恶性样本中显示DNA甲基化轻微下降,在肿瘤中甲基化进一步消失。值得注意的是,PMDs在深度(即DNA甲基化β值)和广度(即基因组位置)上都表现出高度的样本间异质性。研究团队根据甲基化结构域在队列中的出现频率将其分为4组:共享PMDs、EAC特异性PMDs、ESCC特异性PMDs和共享HMD。
为了验证研究的结果,研究团队使用比其他CPG丢失甲基化更快的solo-WCGW CPGs来检测4个结构域组内的平均甲基化丢失。结果显示,在EAC样本中,共享PMDs和EAC特异性PMDs的甲基化水平低于其他两组。反过来,在ESCC样本中,共享PMD和ESCC特异性PMD的甲基化水平较低。来自TCGA或其他个体研究的独立队列进一步验证了这些DNA甲基化丢失的亚型特异性模式。PMD与Hi-C B区室相关,分析发现亚型特异性PMDs以亚型特异性的方式在B区室中富集。相比之下,共享PMD显示没有这种特异性。与全基因组突变相比,癌症特异性PMDs内的突变显示出更低的年龄相关特征。
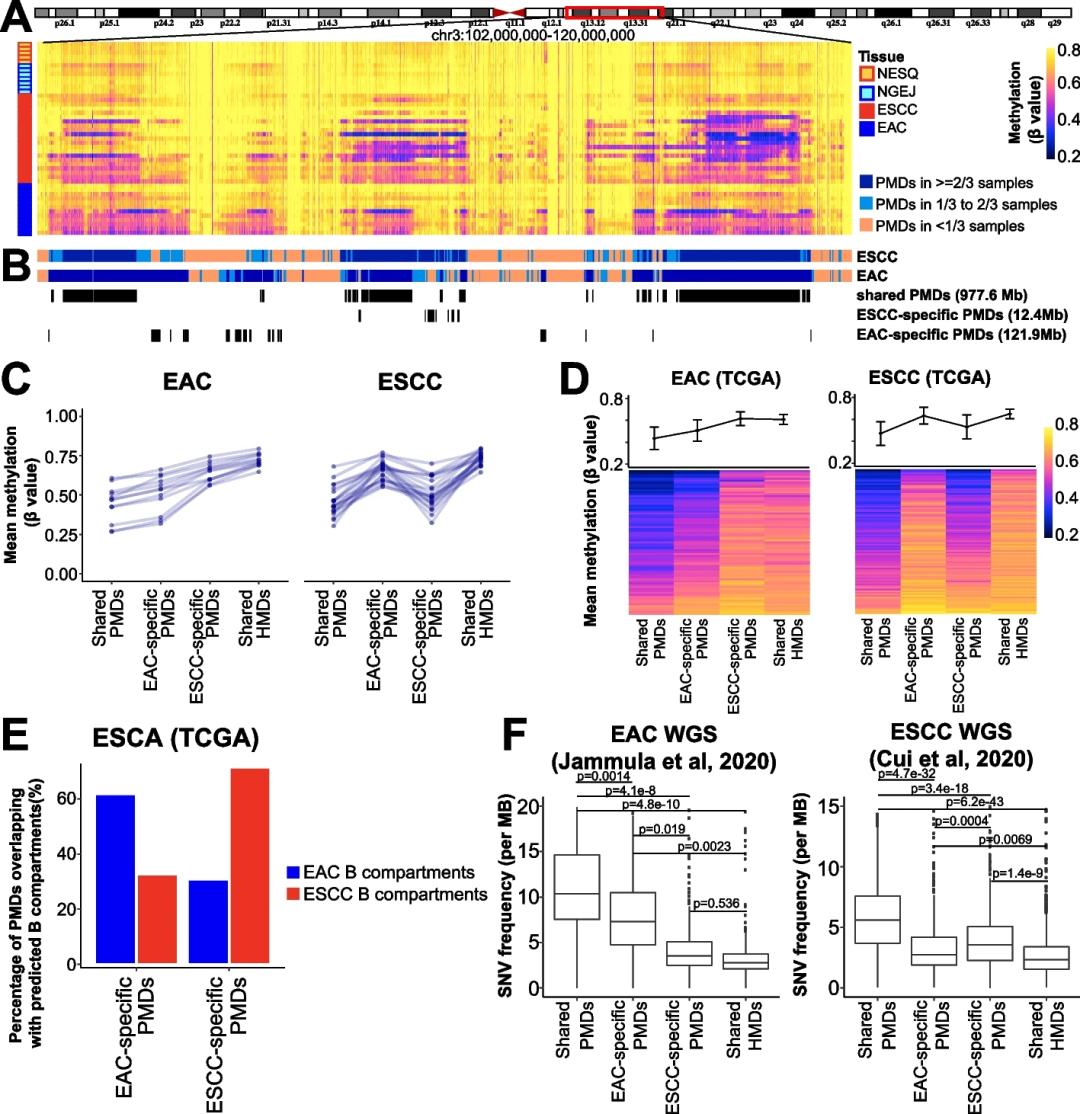
图2.共享和亚型特异性PMD的特征。
此外,研究团队在EAC和ESCC细胞系中进行了H3K36me2芯片测序,并查询了HNSCC细胞系的公开H3K36me2芯片序列数据,发现H3K36me2以细胞类型特异性的方式与PMDs呈负相关。具体来说,在HNSCC细胞系中,共享PMD和ESCC特异性PMD都具有低信号,在HMD(极高或低甲基化结构域)和EAC特异性PMD中发现了高水平的H3K36me2。
食管癌亚型特异性DMRs
在研究PMDs以外的癌症相关甲基化特征时,去除整体低甲基化的影响是至关重要的。为了排除PMD低甲基化的影响,研究团队掩盖了在三分之二的EAC或ESCC样本中发现的任何PMD,随后鉴定了EAC和ESCC样本之间的DMRs。
EAC中共有7734个低甲基化DMRs,ESCC中有5470个低甲基化DMRs(图3)。正如预期的那样,低甲基化DMRs(hypoDMRs)在相应亚型中具有较低的平均甲基化水平。大多数DMRs长约1 - 2 kb,主要位于内含子区和基因间区,与随机背景相似。hypoDMRs区域的染色质可及性分析表明,hypoDMR区域与可接近的染色质和活性组蛋白标记有关。与亚型特异性PMD相似,临床病理参数均未显示对亚型特异性DMRs的影响。
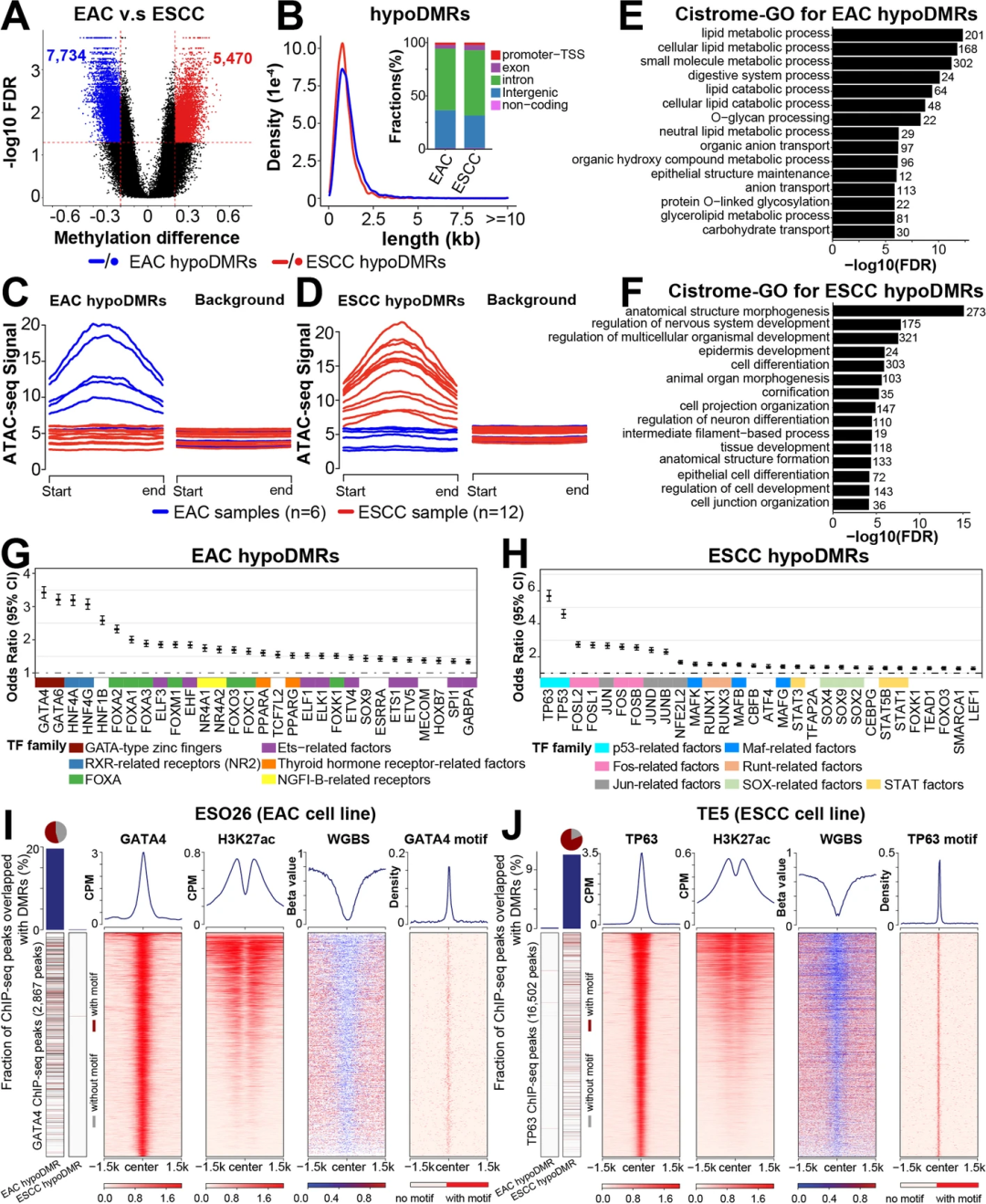
图3.食管癌亚型特异性DMRS。
肿瘤特异性hypoDMRs的鉴定
为了从上述亚型特异性DMR中识别肿瘤特异性的hypoDMR,研究团队对每种hypoDMR的肿瘤及其相应的非恶性样本进行了甲基化比较。(图4)研究发现,25.5%(1972/7734)的EAC hypoDMRs和12.0%(654/5470)的ESCC hypoDMRs在肿瘤中的甲基化水平显著低于相应的非恶性样本,这被称为“肿瘤特异性hypoDMRs(ts-hypoDMRs)”,其余的则被称为“细胞类型特异性DMRS (cts-hypoDMRs)”。ts - hypodrm分布在基因间域和内含子域,与总体上的hypoDMRS和随机背景相似。此外,ts-hypoDMRs在癌症相关通路中富集。以上数据表明,ts-hypoDMRs与促进肿瘤特异性功能的基因有关。

图4.肿瘤特异性hypoDMRs的鉴定。
PMDs和hypoDMRs表现出强烈的细胞类型特异性表观基因组特征
研究团队接下来分析了肿瘤PMDs是否同样具有以上两种类型的甲基化差异。在仅基于肿瘤甲基化组定义的亚型特异性PMDs中,非恶性组织明显表现出与恶性组织相同的甲基化变化模式(图5)。在各自的非恶性样本中,很大一部分亚型特异性PMDs已经低甲基化。对hypoDMRS的相同分析证实,超过80%的亚型hypoDMRS在相应的非恶性样本中显著降低了DNA甲基化。以上数据表明,从肿瘤样本中鉴定出的亚型特异性PMDs和hypoDMRS的很大一部分反映了正常对应物中存在的甲基化差异,肿瘤中甲基化丢失的程度明显更高。对正常样本的分析也表明PMDs和hypoDMRs都具有很强的正常细胞类型特异性。
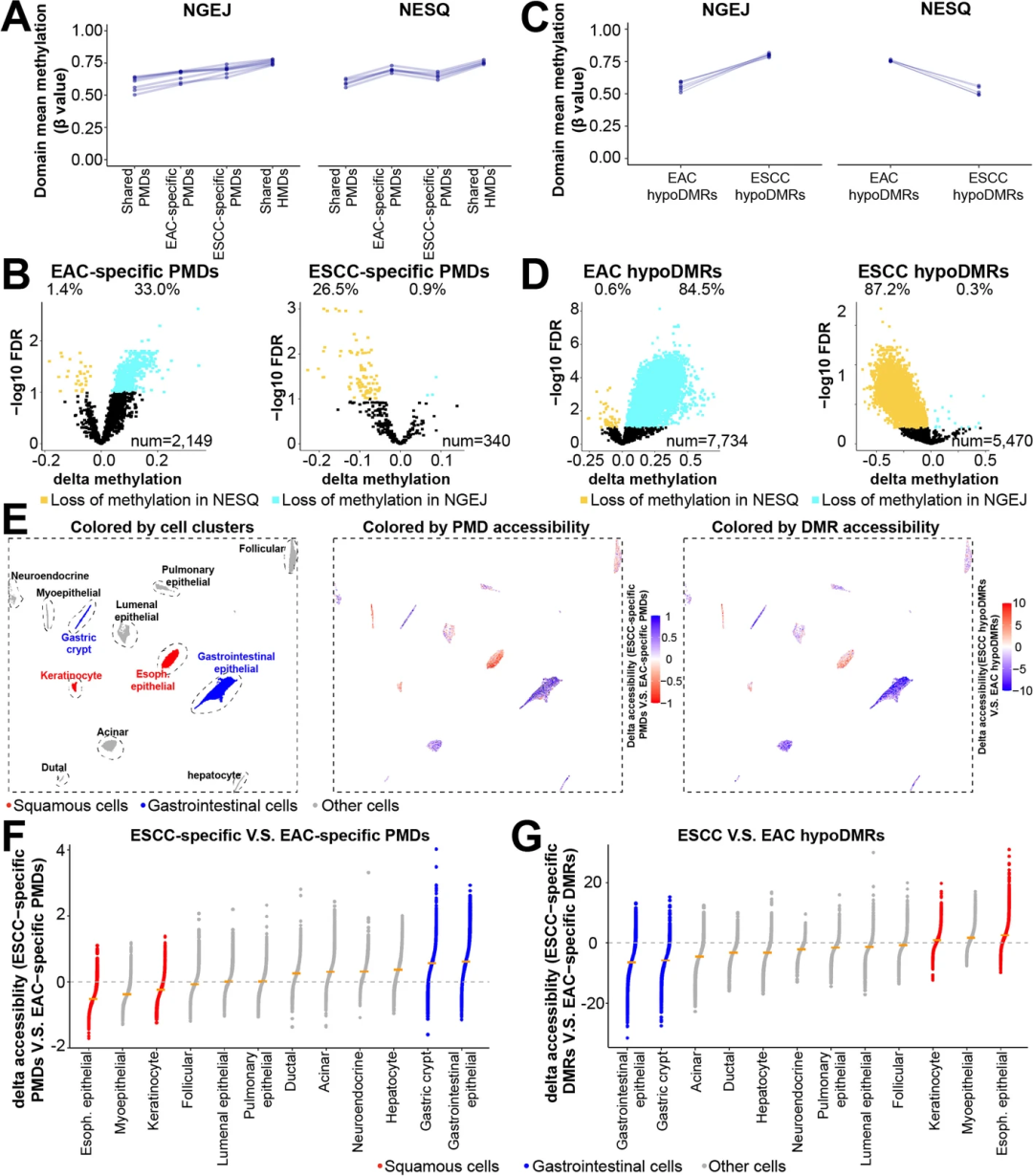
图5.PMDs和hypoDMRS表现出强烈的细胞类型特异性表观基因组特征。
亚型特异性PMDs和hypoDMRs的泛癌分析
基于上述研究结果,研究团队猜测发现的PMDs和hypoDMRs可能分别与其他鳞状上皮和胃肠道腺癌共有,并进行了泛癌分析。(图5)研究人员首先分析了所有33种癌症类型中亚型特异性PMDs和hypoDMRs之间的甲基化变化,发现大多数泛胃肠道肿瘤在EAC特异性PMD中丢失DNA甲基化,大多数泛鳞状肿瘤在ESCC特异性PMD中甲基化降低。在hypoDMRS亚型中观察到高度一致的结果。在共享的PMD和HMD中没有发现特定的模式。随后的数据分析表明,PMDs和DMRs的甲基化水平对特定细胞类型具有高度预测性,并且可以作为癌症诊断的潜在生物标志物,尤其是当这两种特征结合时。

图5.泛癌症数据集中PMDs和hypoDMRs的分析。
结 语
综上所述,研究团队们生成了迄今为止食道癌中最大的WGBS数据集之一,集中分析了PMDs(大规模)和DMRs(小规模)的分析,并揭示了这些区域新的表观遗传学特性。该研究强调了正常细胞类型中细胞类型特异性PMDs和DMRs的存在,它们在恶性细胞中得以保留。研究团队表示,这是第一次证明PMDs在正常、前驱和恶性状态下具有显著的细胞类型特异性。
参考资料:
Zheng, Y., Ziman, B., Ho, A.S. et al. Comprehensive analyses of partially methylated domains and differentially methylated regions in esophageal cancer reveal both cell-type- and cancer-specific epigenetic regulation. Genome Biol 24, 193 (2023). https://doi.org/10.1186/s13059-023-03035-3


