Nature:不止蛋白质结构!AlphaFold3可高精确度预测生物分子结构及相互作用
时间:2024-05-26 06:02:39 热度:37.1℃ 作者:网络
准确预测的生物分子复合物结构,对于人们理解细胞功能和开发合理治疗方法至关重要。随着AlphaFold的发展,人们在蛋白质的结构预测方面已经取得了巨大的进步,AlphaFold2使得蛋白结构预测达到实验层面的精度,此外,在AlphaFold2思想和技术基础上开发的一系列新算法和研究新进展也使这一领域得到了长足发展。AlphaFold在问世不久后,依靠简单的输入修改就可实现蛋白质相互作用预测,并且科研人员专门为蛋白质相互作用预测训练AlphaFold 2产生了一个高度准确的系统。
上述成功的结果引发了一个问题,即是否有可能在深度学习框架内准确预测包含更广泛生物分子的复合物的结构,包括配体、离子、核酸和修饰残基等。目前,针对各种特定相互作用类型的多种深度学习预测方法已被开发,但这些方法的准确性参差不齐,通常低于物理启发方法,并且几乎所有方法都高度专注于特定的相互作用类型,无法预测包含多种实体类型的一般生物分子复合物的结构。
为解决上述难题,谷歌DeepMind AlphaFold团队联合Isomorphic Labs公司在Nature杂志发表了题为“Accurate structure prediction of biomolecular interactions with AlphaFold 3”的文章,推出了全新的预测工具AlphaFold3(AF3),这是一个能够高精度预测蛋白质数据库(PDB)内几乎所有分子类型复合物的模型,包括准确预测蛋白质、DNA、RNA、小分子配体结构以及其相互作用模式。
与先前许多专业工具相比,AF3模型准确性显著提高:预测蛋白质-配体相互作用的准确性远高于最先进的对接工具,预测蛋白质-核酸相互作用的准确性远高于核酸特异性预测工具,抗体-抗原预测精度显著高于AlphaFold-Multimer v2.3。这些结果表明,在一个统一的深度学习框架内,可实现跨生物分子空间的高精度建模。综上,AF3的发布在生物学界具有革命性的意义,将使结构生物学研究变得更加普及,并有望推动药物研发。

文章发表在Nature
主要研究内容
01 模型架构与训练
AF3的整体结构与AlphaFold 2相似,均有一个大主干,用于演化化学复合物的配对表示,然后是一个结构模块(Structure Module),使用配对表示来生成明确的原子位置,但每个主要组件存在较大差异。
AF3减少了多序列比对(MSA)模块的数量(减至4个),MSA表示的处理使用配对加权平均法,并且只有配对表示用于后面的处理步骤;更简单的Pairformer模块取代AlphaFold 2的Evoformer模块成为主要的处理模块,减少对MSA信息的依赖,更多地依靠pair信息。特别地,AF3引入了扩散模块(Diffusion Module)取代AlphaFold 2中的结构模块,可直接预测原始原子坐标。扩散过程的多尺度性质(低噪声引导神经网络、改善分子的局部结构)也能消除立体化学损失,还能减少网络中键合模式(bonding patterns)的特殊处理。
在模型训练数据上,AlphaFold 3模型根据PDB中包含的生物分子结构数据进行训练,能够处理其中99%以上的已知生物分子复合物。
研究团队还开发了预测最终结构中原子水平和配对误差的置信度措施,这一过程在AlphaFold 2中通过训练期间回归结构模块输出中的误差直接完成,但其不适用于扩散训练。为此,研究团队开发了一个“rollout”过程,用于在训练过程中生成完整结构预测,使用该预测结构来排列对称的地面真值链和配体,并计算性能指标来训练置信度。
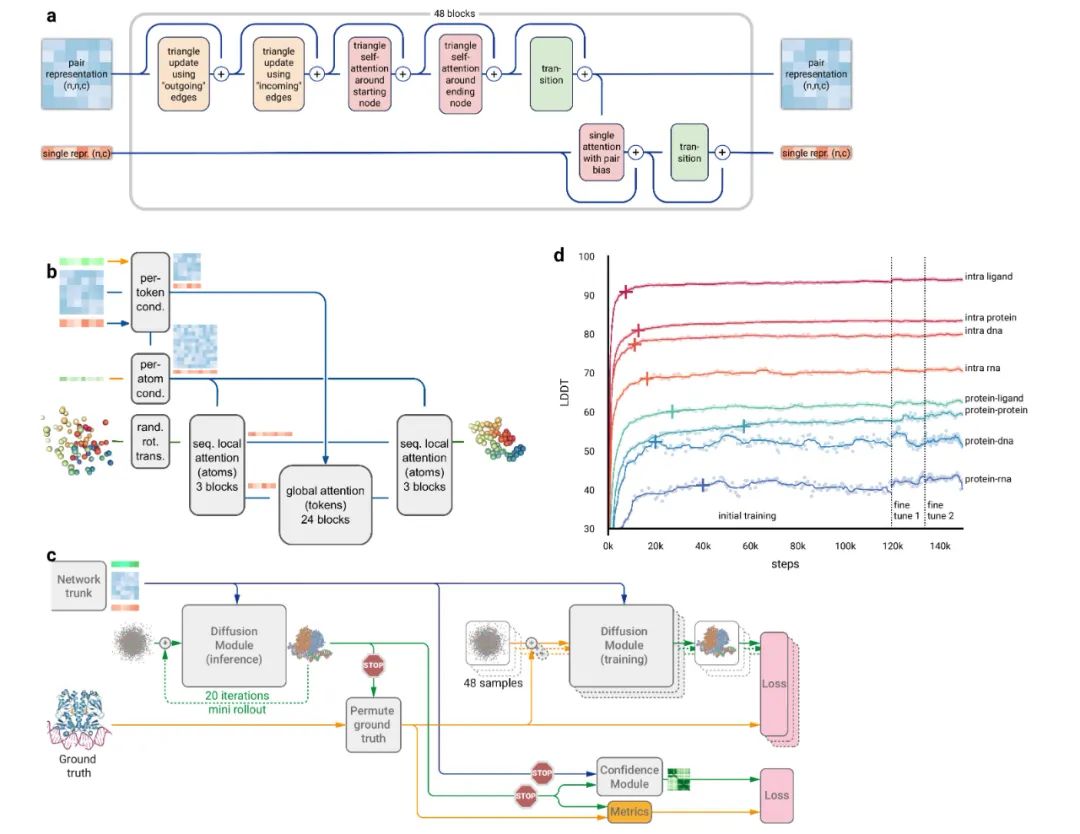
图1. AF3架构和训练细节
02 模型预测的准确性
AF3可以预测输入的聚合物序列、残基修饰和配体SMILES的结构,在图2中研究团队展示了一些例子,凸显了该模型具有推广到许多生物学上重要和治疗相关模式的能力。此外,AF3还比各种单一模型表现出更高的性能,包括蛋白质-小分子、核酸、共价、抗原抗体等各种分子的相互作用。
在PoseBusters基准数据集上,研究团队评估了AF3预测蛋白质-配体相互作用的性能,该数据集由428个蛋白质-配体结构组成。结果显示,在不使用任何结构输入的情况下,AF3预测准确率为76%,性能显著优于传统对接方法Vina以及最新开发的RFAA模型(基于深度学习方法预测生物大分子结构)。对于蛋白质-核酸复合物和RNA结构,AF3预测准确性也高于RoseTTAFold2NA(目前最好的预测方法),AF3能够预测具有数千个残基的蛋白质-核酸结构。此外,AF3还可以准确预测共价修饰,包括对任何聚合物残基(蛋白质、RNA或DNA)的修饰。
在扩展建模能力的同时,相对于AlphaFold-Multimer v2.3,AF3也提高了对蛋白质复合物的预测准确度。具体而言,AF3对蛋白质-蛋白质预测成功率有所提高,特别是对于抗体-蛋白质相互作用的预测有了显著提高,准确率达62.9%;对蛋白质单体LDDT的预测则有显著改善。上述结果表明,AF3在预测类药物相互作用(包括蛋白质-配体结合以及抗体-靶蛋白结合)方面展现出前所未有的准确性,可加速药物发现过程,大幅度减少传统药物发现方法所需的时间和成本。
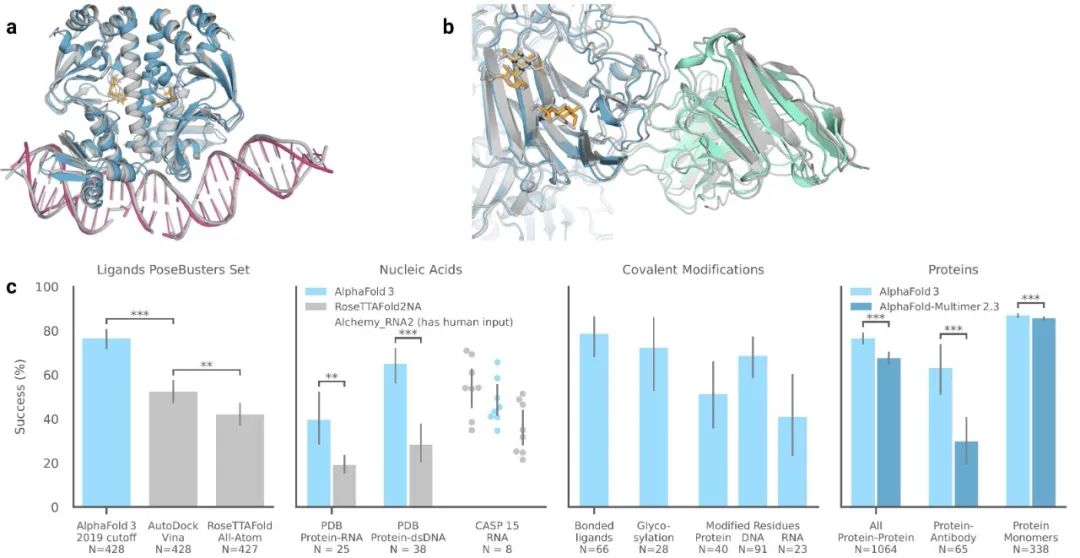
图2. AF3可以准确预测生物分子复合物的结构
03 AF3预测准确性与置信度
与AlphaFold 2一致,AF3的置信度也经过精确校准。研究团队在最近的PDB评估集上进行了置信度分析,使用模型预测的相互作用界面TM分数(ipTM)作为置信度指标;随后,将ipTM分数进行分段处理,统计每个区间内的样本数量,并将其与蛋白质-蛋白质匹配的DockQ分数、蛋白质-核酸匹配的iLDDT分数以及蛋白质-配体匹配的成功率进行比较。
分析结果显示,ipTM分数与上述指标之间存在显著正相关性,表明其是一个有效的模型预测结果评估指标。此外,研究团队还对另一个置信度指标pLDDT进行了类似的实验,发现其同样与模型预测的准确性之间存在正相关性。因此,ipTM分数、pLDDT均能很好地评估模型预测结果,并为模型的预测准确性提供有力的证据。
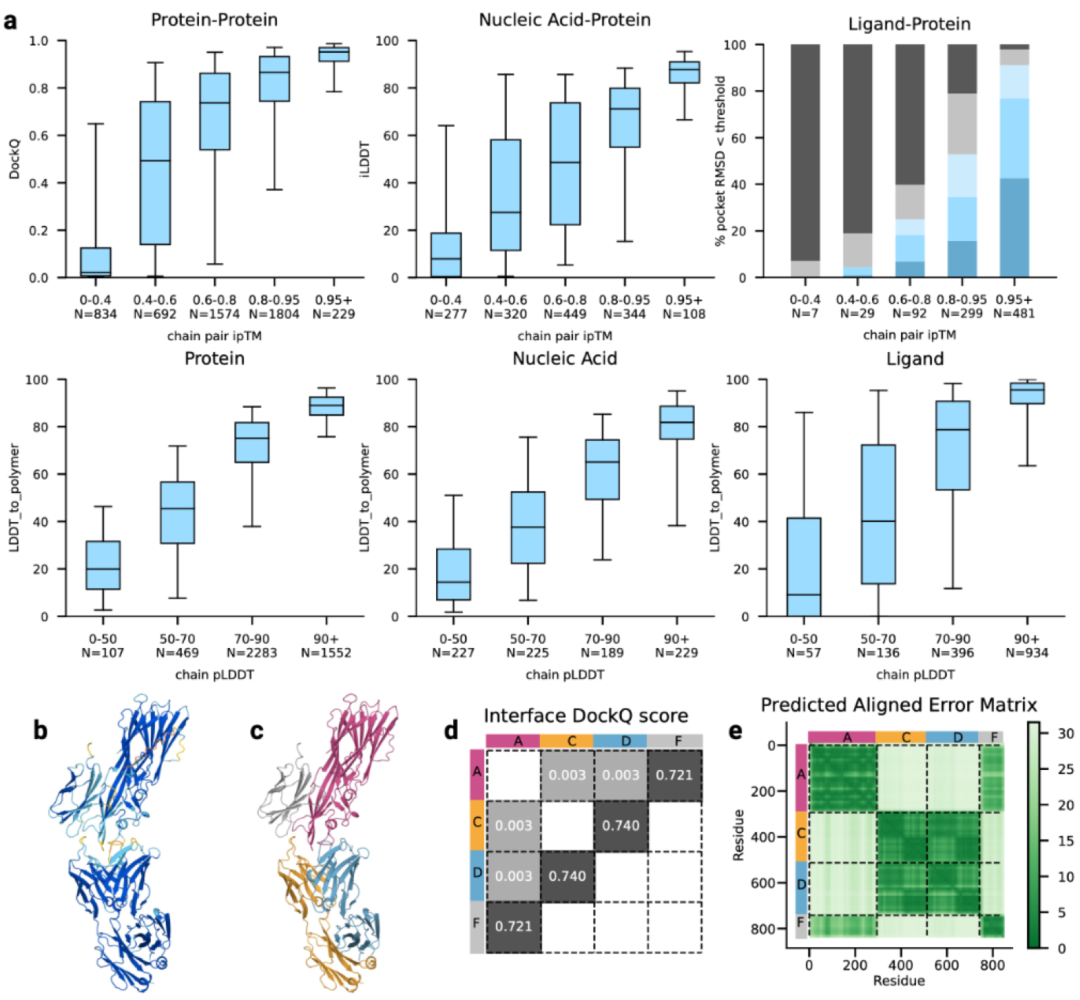
图3. AF3置信度指标与准确性相关
04 AF3模型局限性分析
AF3在蛋白质结构预测领域取得了显著的成就和空前的精度,但在立体化学、幻觉(hallucinations)、动力学和特定目标的准确性等方面仍存在一定局限性。即便如此,现有环境下,AF3仍是蛋白质结构预测领域的一个重要工具,其进步和革新也为未来的研究和应用奠定了基础。当然,这些挑战也是研究人员开发新算法或改进现有算法的动力,最终有望实现更精确和全面的结构预测。
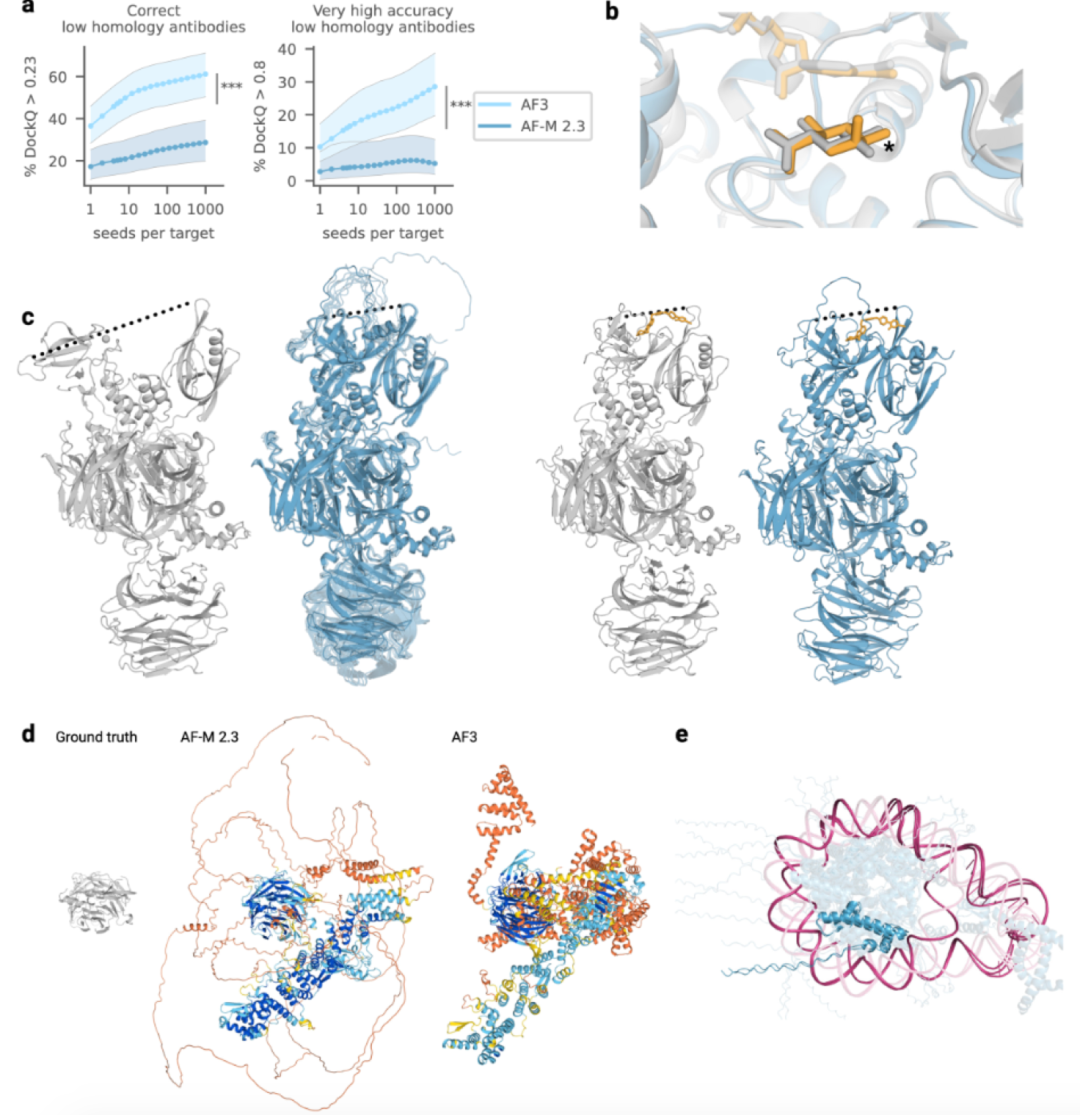
图4. AF3模型局限性
目前,AlphaFold3的访问次数有限,科研人员每天只能进行10次预测,研究团队也表示未有开源AlphaFold 3完整代码的计划。但研究团队推出了一个支持非商业性研究的工具AlphaFold Server,可以免费访问AlphaFold3的大部分功能,界面使用简单、可视化程度良好。
Alphafold Server链接:
https://golgi.sandbox.google.com/about
结 语
分子生物学的核心挑战是理解并最终调节生物系统中复杂的原子相互作用,AlphaFold 3的问世在这一方向上迈出了一大步,其将人们从蛋白质带到广泛的生物分子,证明了在一个统一的框架中准确预测不同生物分子系统的结构是完全可能的,并有望开启更多变革性的科学。 该研究提示,基于实验技术解析结构会带来更多高质量蛋白质复合体结构,而这些可作为AI模型的训练数据,进一步提高模型的泛化能力和准确性。因此,实验技术的发展和计算方法的发展是相互补充的,两者共同发力,可帮助人们更好地理解生物世界。
参考原文:
Abramson, J., Adler, J., Dunger, J. et al. Accurate structure prediction of biomolecular interactions with AlphaFold 3. Nature (2024). https://doi.org/10.1038/s41586-024-07487-w


