论文解读| Xianjun Zhu/Xiaohui Zhao教授团队揭示Emc6对视紫红质定位和光感受器细胞存活的重要性
时间:2024-08-29 23:02:01 热度:37.1℃ 作者:网络
视网膜色素变性是一种导致视力严重损害并可能最终致盲的异质性疾病,其主要特征是光感受器细胞的逐渐退化。视紫红质是杆状光感受器中的关键蛋白质,其突变在常染色体显性视网膜色素变性病例中约占25%。内质网膜蛋白复合体(EMC)负责监管具有尾锚定或多个跨膜结构域的膜蛋白的生物合成和质量控制。其中,Emc6是EMC的的不可或缺亚单位。然而,EMC6在视网膜光感受器细胞中的功能仍然知之甚少。
中国电子科技大学/中国科学院西北高原生物研究所的Xianjun Zhu/Xiaohui Zhao教授团队在本刊发表了题为"The endoplasmic reticulum membrane protein complex subunit Emc6 is essential for rhodopsin localization and photoreceptor cell survival"的研究论文。该研究通过构建Emc6在杆状和锥状光感受器细胞中特异性敲除的小鼠模型,深入探讨了Emc6在视网膜光感受器细胞中的功能,及其缺失如何导致视紫红质定位异常和光感受器细胞退化。
01 Emc6 敲除引发视网膜退化
研究团队构建了Emc6杆状光感受器细胞特异性敲除小鼠(RKO)和锥状光感受器细胞特异性敲除小鼠(CKO)模型。结果表明,RKO小鼠出现早期视网膜退化,光感受器细胞减少,外核层变薄,视觉功能受损(图1)。CKO小鼠则表现为锥状光感受器细胞数量减少以及视锥细胞视蛋白的定位异常。
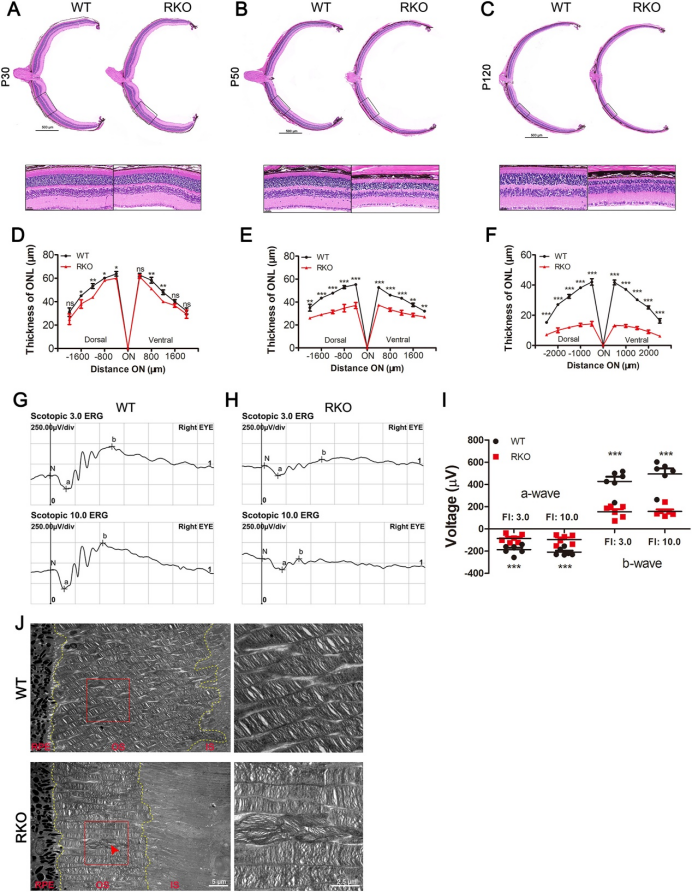
图1.Emc6 敲除小鼠的进行性退化和功能受损(原文中Figure 2)。
02 Emc6 缺失导致视紫红质在光感受器外段(OS)定位异常和表达降低
通过免疫荧光实验,研究团队观察到P30 RKO小鼠的视紫红质蛋白定位异常,并伴随OS的缩短。免疫印迹分析进一步揭示,RKO小鼠的视紫红质蛋白表达量较野生型(WT)小鼠下降了约35%(图2)。这些结果表明Emc6对于视紫红质的正确定位和光感受器的稳定维持具有至关重要的作用。
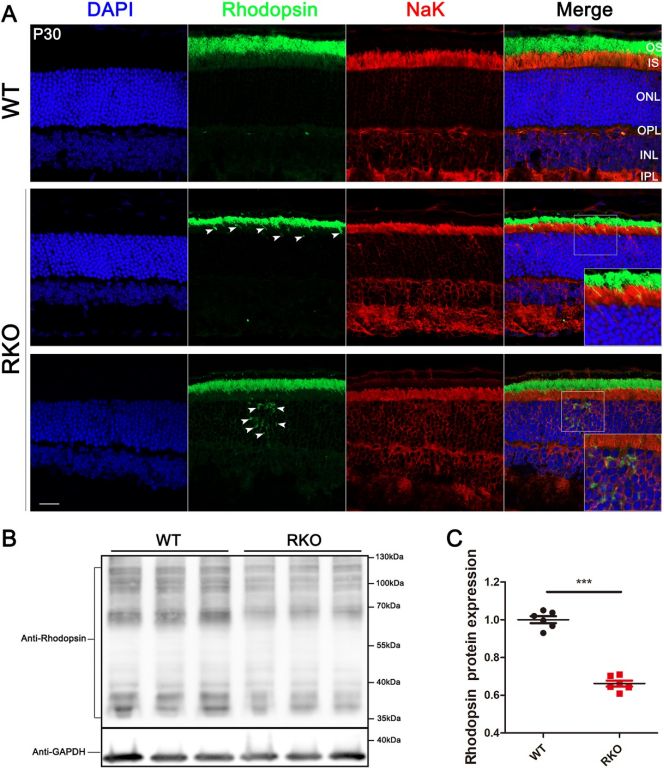
图2.Emc6敲除小鼠视网膜中视紫红质蛋白的错位和表达降低(原文中Figure 3)。
03 Emc6 缺失引发反应性星形胶质化和光感受细胞凋亡
定位错误的视紫红质蛋白具有细胞毒性,可触发细胞凋亡。在P30 RKO小鼠中,视网膜出现凋亡现象,同时星形胶质细胞被激活,表现为反应性星形胶质化,这是视网膜损伤的标志。GFAP抗体染色显示,RKO小鼠视网膜中GFAP表达增加,荧光信号在多个视网膜层中增强,表明存在病理损伤。TUNEL检测进一步证实了RKO小鼠光感受器细胞正在发生凋亡(图3)。
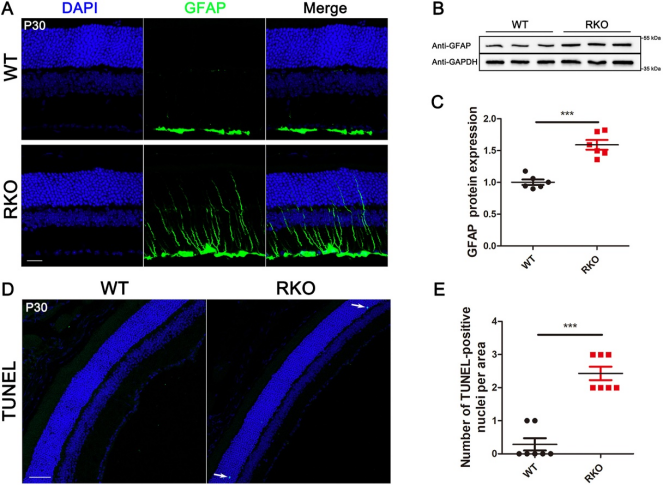
图3.Emc6敲除小鼠的视网膜中星形胶质细胞增生和光感受细胞凋亡(原文中Figure 4)。
04 Emc6 缺失导致杆状光感受器细胞纤毛缺陷
蛋白质组学分析显示,RKO小鼠视网膜中与纤毛发育或成熟相关的蛋白质表达下调,特别是ANO2和TMEM67。免疫荧光结果显示,ANO2和TMEM67主要定位于杆状光感受器细胞纤毛,并且在RKO小鼠中的表达降低。电镜结果显示,RKO小鼠杆状光感受器细胞纤毛长度和数量均减少(图4)。总体而言,Emc6缺失导致纤毛相关蛋白水平下降和纤毛缺陷。
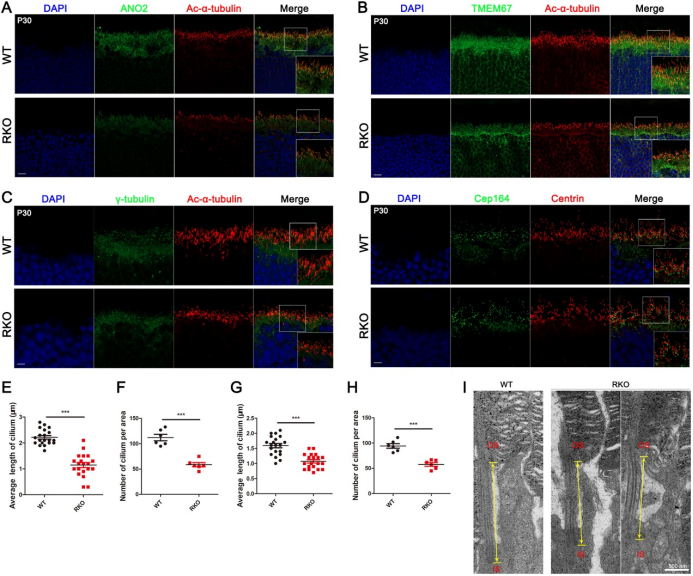
图4.杆状光感受器细胞纤毛在Emc6敲除小鼠中异常(原文中Figure 7)。
05 Emc6 缺失导致膜盘蛋白运输受损
研究还发现,RKO小鼠中参与光转导的膜盘蛋白运输受损,GNAT1和GNB1在细胞内段定位错误,GNAT1表达下降。PDE6B和PDE6G的运输受阻,PDE6B表达显著降低(图5)。这些结果表明Emc6缺失导致纤毛缺陷,进而影响膜盘蛋白定位。
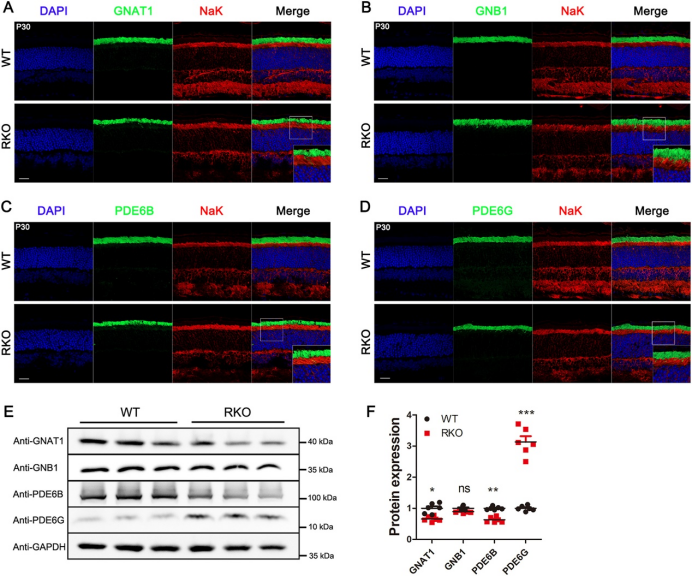
图5.Emc6敲除小鼠中膜盘蛋白错位(原文中Figure 8)。
Emc6蛋白在视网膜光感受器细胞中扮演着至关重要的角色。Emc6的缺失不仅会导致杆状光感受器细胞纤毛缺陷,影响膜盘蛋白的运输,还会导致光感受器细胞退化和死亡。此外,Emc6的缺失同样会导致锥状光感受器细胞功能障碍。这些发现为理解视网膜疾病的发生发展提供了新的视角,并为开发新的治疗策略提供了潜在的靶点。
免费全文下载链接:
https://www.sciencedirect.com/science/article/pii/S2352304223002076
引用这篇文章:
Sun K, Liu L, Jiang X, et al. The endoplasmic reticulum membrane protein complex subunit Emc6 is essential for rhodopsin localization and photoreceptor cell survival. Genes Dis. 2024;11(2):1035-1049.


