Nature:蛋白质测序技术突破:PASTOR单分子水平精准解读蛋白质突变和修饰
时间:2024-09-14 06:02:18 热度:37.1℃ 作者:网络
引言
蛋白质的多样性在生物学过程中发挥着至关重要的作用,尤其在疾病状态的识别和治疗开发中具有关键意义。然而,现有的技术在完整蛋白质的测序方面面临着诸多挑战,例如检测灵敏度、动态范围、分析通量以及设备成本等问题。为了解决这些问题,研究者们提出了基于纳米孔(nanopore)技术的新型蛋白质分析平台(9月11日Nature “Multi-pass, single-molecule nanopore reading of long protein strands”)。纳米孔技术最初用于核酸测序,其通过感知单分子穿过纳米孔时的电流变化,能够直接、无标签地读取分子信息。这项技术不仅能够区分肽段和蛋白质,还能实时监测蛋白质-蛋白质和蛋白质-配体相互作用,并检测氨基酸和翻译后修饰(post-translational modifications, PTMs),如磷酸化(phosphorylation)和糖基化(glycosylation)。
该研究中,研究者开发了一种基于纳米孔的长链蛋白质单分子读取方法,通过结合ClpX解折酶(unfoldase)的作用,使蛋白质以可控的方式通过CsgG纳米孔,实现了对长达数百个氨基酸的完整蛋白质链进行测序。这项技术能够识别单个氨基酸的突变以及蛋白质链上的翻译后修饰。为提高测序准确性,研究者还展示了多次重读同一蛋白分子的能力,并探讨了蛋白条形码(barcode)测序的潜力。此外,研究者开发了一个物理模型,用于模拟基于氨基酸残基体积和电荷的原始纳米孔信号,从而增强了信号数据的解释能力。该研究证明了这一平台在单分子水平上识别和表征完整蛋白质形式的潜力,为未来蛋白质测序和条形码标记技术的发展奠定了基础。

研究开发了一种基于纳米孔(nanopore)和解折酶ClpX的蛋白质测序技术——PASTOR(Protein Annotation by Single-molecule Translocation and Overwriting),其核心在于通过纳米孔读取单分子蛋白质链的电流信号,并结合机器学习和物理建模技术实现蛋白质序列的精准测序。研究通过设计特殊的蛋白质结构块(每块59个氨基酸)并引入单个氨基酸突变,使用双酪氨酸标记(tyrosine mutation)来分割电流信号,进而实现对氨基酸序列的识别和翻译后修饰的探测。
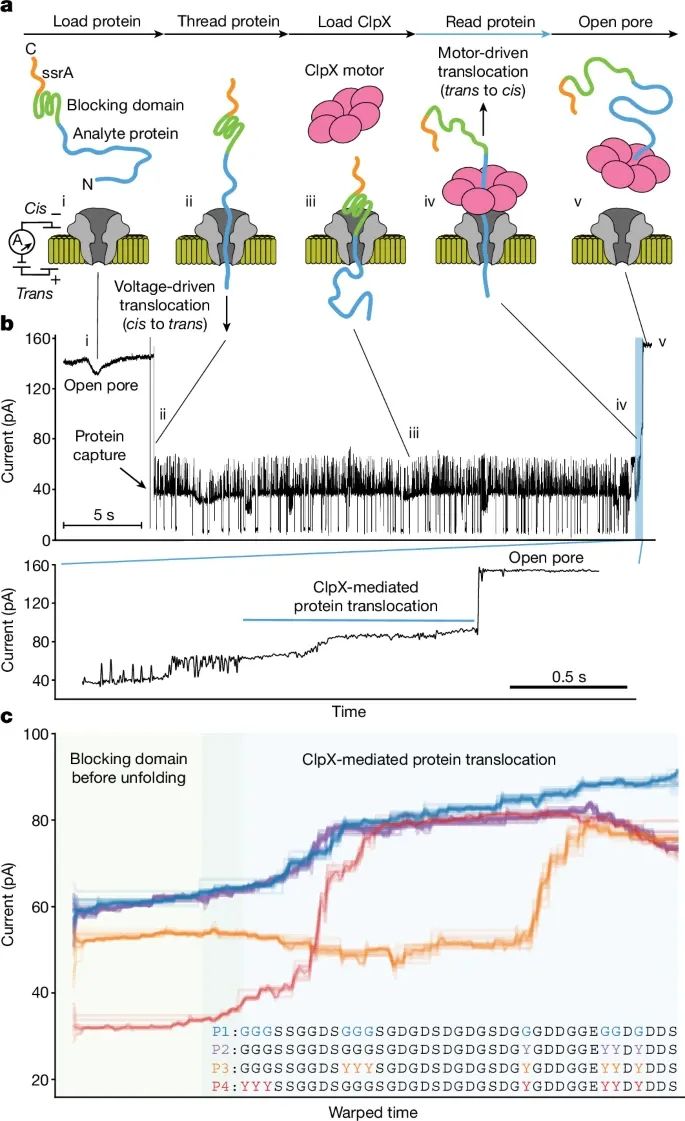
纳米孔技术结合解折酶ClpX读取蛋白质的过程(Credit: Nature)
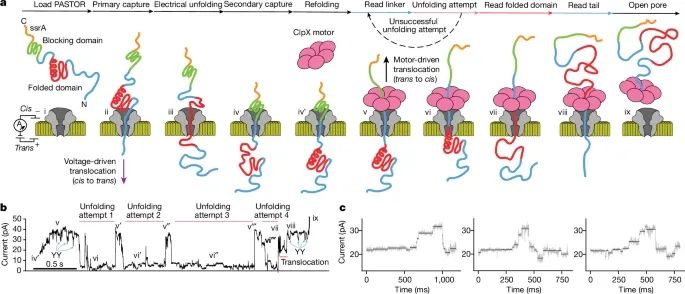
蛋白质通过纳米孔的过程:图a展示了在MinION平台上,解折酶介导的蛋白质转运方法。首先,蛋白质被电场驱动从顺电极(cis)穿入纳米孔,然后ClpX解折酶从反电极(trans)方向将其拉回,实现蛋白质的单分子转运和读取。
电流信号变化:图b显示了在蛋白质通过纳米孔时,电流信号的变化。每个状态的电流读数与蛋白质在纳米孔中的位置相关,特别是当蛋白质的捕获和ClpX开始作用时,电流读数出现显著的下降。这些电流信号可以被用来检测和解析蛋白质的结构信息。
蛋白质突变的检测:图c展示了不同突变蛋白的信号差异,通过对比正常蛋白(P1)和三个带有不同突变的蛋白质(P2, P3, P4),研究者发现每个蛋白的电流信号有明显的区别。这些差异与突变的位置密切相关,证明该方法可以区分单个氨基酸的突变。
研究采用了基于ClpX解折酶的蛋白质转运模型,通过在纳米孔平台上监测蛋白质通过纳米孔时的电流变化,来捕捉蛋白质的结构信息。具体步骤包括将蛋白质样品加载至纳米孔装置中,并通过电压驱动蛋白质链穿过纳米孔。ClpX解折酶的作用下,蛋白质被逐步解折,并以一定的速度通过孔道,产生特定的电流信号。这些信号被归类为“YY dips”和“variable regions (VRs)”,分别对应于双酪氨酸标记区域和可变区域。
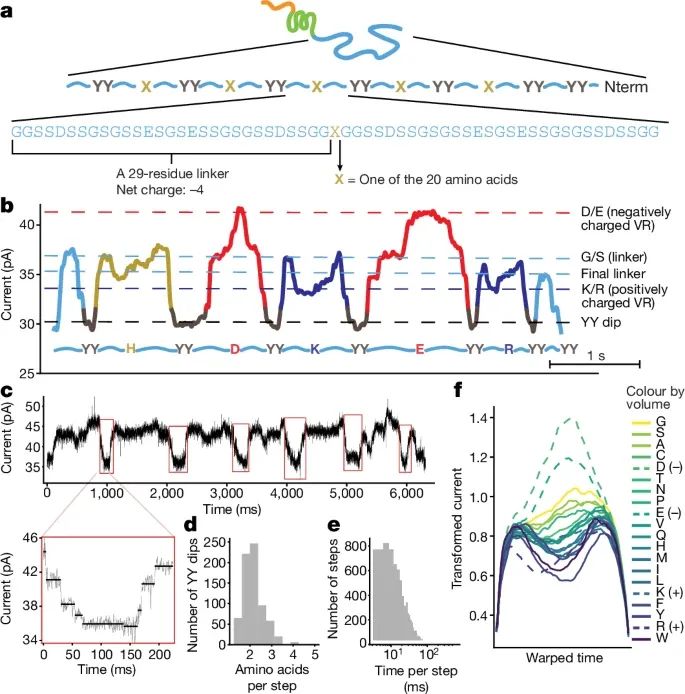
通过PASTOR技术检测单个氨基酸替换以及ClpX解折酶驱动蛋白质通过纳米孔的步进过程(Credit: Nature)
蛋白质构成与设计:图a展示了PASTOR蛋白的序列组成,包含重复的59个氨基酸块和在中间位置引入的单个氨基酸突变。通过设计这种特定的蛋白质序列,研究者能够在每个序列块中检测到特定的氨基酸突变。
电流信号的捕捉和分析:图b显示了PASTOR蛋白的纳米孔电流信号轨迹,每个重复的酪氨酸双突变(YY dips)产生了明显的电流下降,信号之间的可变区域则对应于氨基酸的不同突变。这些信号是通过对电流变化的仔细分析和分类获得的。
ClpX的步进行为:图c展示了ClpX解折酶在驱动蛋白质穿过纳米孔时的步进行为。研究表明,ClpX以大约两个氨基酸为步进单位来拉动蛋白质链,每一步的电流信号产生的变化显示了ClpX的精确步进行为。
单个氨基酸的识别:图d-f通过对电流信号的进一步分析,揭示了不同的氨基酸突变如何影响电流信号。研究者发现,电流信号的变化与氨基酸的体积和电荷密切相关,特别是带正电和带负电的氨基酸对电流的影响显著不同。图2f显示,体积较大的氨基酸会阻挡更多的电流,而带负电的残基会增加电流。
通过物理模型,研究者进一步将每个氨基酸的体积和电荷与电流信号关联起来。这个模型利用滑动窗口技术,对序列的电流信号进行逐段分析,并通过机器学习技术优化分类模型,最终实现对不同氨基酸突变的准确识别。
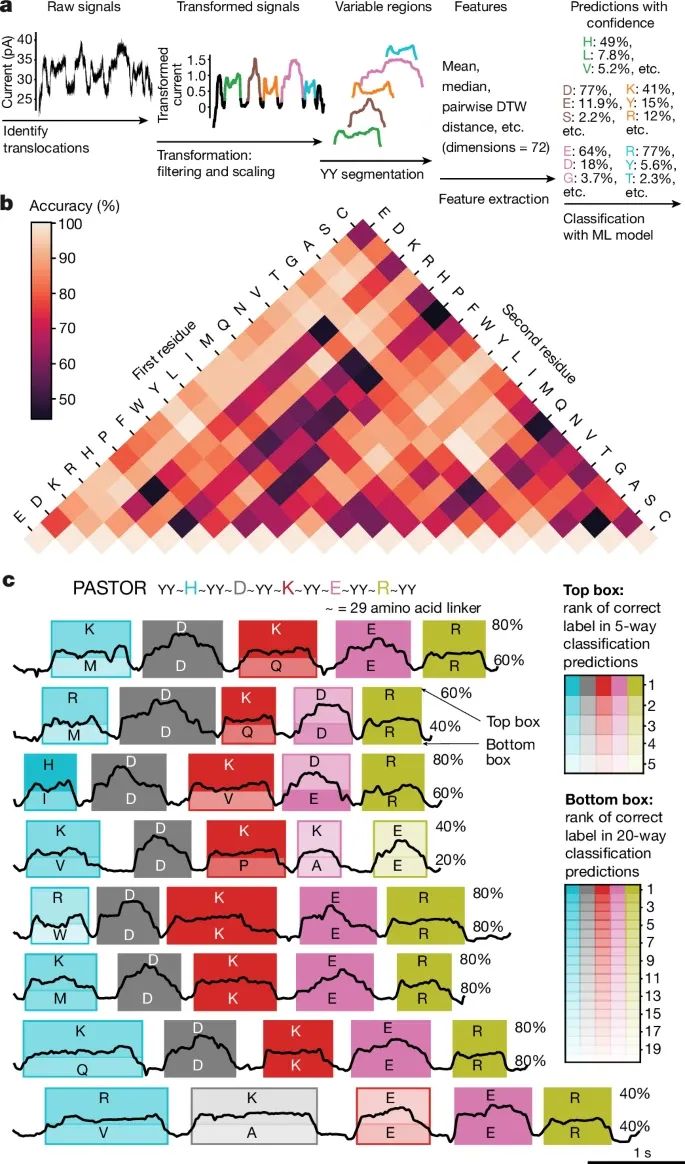
利用PASTOR技术进行单分子水平的纳米孔测序,来区分单个氨基酸突变的结果(Credit: Nature)
信号处理与分类:图a展示了PASTOR蛋白的信号处理流程。首先,对原始电流信号进行过滤和缩放,然后通过双酪氨酸标记(YY segmentation)对电流信号进行分段,并提取变量区域(variable regions, VRs)中的信号特征。这些特征被用于后续的机器学习分类器分析,以识别不同的氨基酸突变。
氨基酸对的区分准确性:图b是展示不同氨基酸对之间的分类准确性热图。研究者利用随机森林模型(random forest model)对不同的氨基酸进行分类分析,结果显示那些体积差异大或带负电的氨基酸对,能够以较高的准确性进行区分。例如,酪氨酸与天冬氨酸的区分达到了100%的准确性,而体积相似的氨基酸对(如亮氨酸和异亮氨酸)则更难以区分。
PASTOR蛋白的突变测序示例:图c展示了PASTOR蛋白(HDKER)的氨基酸突变的测序示例。通过对电流信号的变量区域进行预测,模型能够对PASTOR蛋白中的每个氨基酸残基进行准确预测。对于特定的氨基酸序列,研究者使用5种氨基酸分类(如G、V、W、R、D),模型的单次读取准确率为86%。在最具挑战的20种氨基酸分类任务中,模型的准确率为28%,远高于随机分类的5.5% 。
研究表明,基于ClpX解折酶的纳米孔测序技术能够实现单分子蛋白质链的高精度读取,尤其是对翻译后修饰的检测有较好的灵敏度。在检测过程中,研究团队通过对不同氨基酸残基进行信号分类,发现某些氨基酸(如带负电的磷酸化残基)会显著影响电流信号的幅度,表明该技术可以准确检测蛋白质中的翻译后修饰。此外,该研究还开发了一种“rereading”策略,通过引入“滑动序列”(slip sequence)来允许蛋白质多次通过纳米孔,从而提高测序的准确性和信噪比。
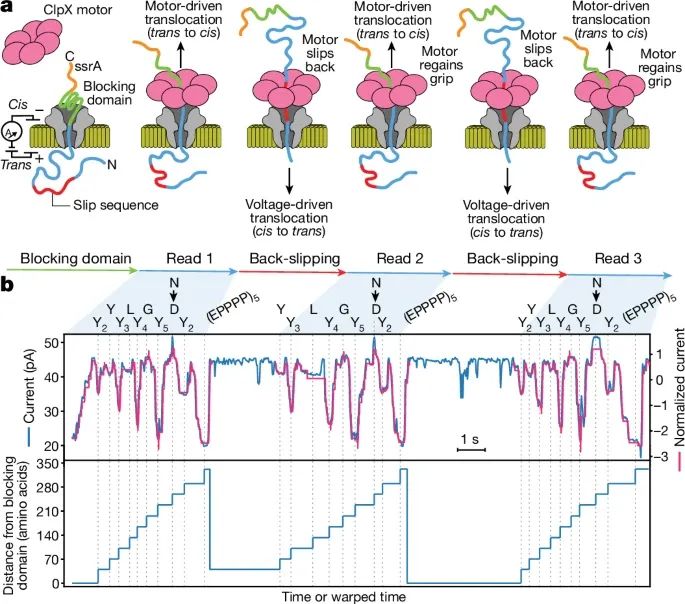
通过引入滑动序列(slippery sequence)实现蛋白质分子的多次重读(Credit: Nature)
滑动序列的工作模型:图a展示了滑动序列的工作原理。PASTOR蛋白中加入了富含脯氨酸的滑动序列((EPPPP)₅),这使得ClpX解折酶在拉动蛋白质时可能会失去抓握,导致蛋白质重新被电场驱动通过纳米孔。ClpX解折酶随后可以再次抓住蛋白质并继续拉动蛋白质从孔中出来,实现多次重读。
多次读取蛋白质的电流信号示例:图b展示了引入滑动序列的PASTOR蛋白(称为PASTOR-reread)的三次读取示例。在每次读取中,蛋白质的电流信号显示出一致的重复模式,尤其是每个变量区域(VR)的电流变化与模型预测信号高度一致。这表明蛋白质分子在经过多次重读后仍然保留了高度可重复的电流信号特征。
滑动距离的分布:图b还展示了不同读取事件中ClpX滑动的距离。研究发现,滑动的距离分布在短程(50-100个氨基酸)和长程(超过300个氨基酸)之间,大约40%的重读事件属于短距离滑动,30%的重读事件属于长距离滑动。通过这种策略,蛋白质分子可以被多次重读,大大提高了读取准确性。
多次重读提高测序准确性:研究表明,通过多次重读策略,单分子蛋白质测序的准确性得到了显著提升。在20种氨基酸的分类任务中,多次重读将准确率从单次读取的28%提升至61%。在7种氨基酸分类任务中,准确率从66%提升至99%。
实验数据表明,在优化的实验条件下,PASTOR蛋白的转运速率约为每秒63个氨基酸(即每个氨基酸的平均驻留时间为16毫秒),这与之前的ClpX转运速度估计值相符。通过重复读取和信号分类,研究团队成功地实现了对蛋白质条形码(barcode)的多次重读,显著提高了测序的准确性。
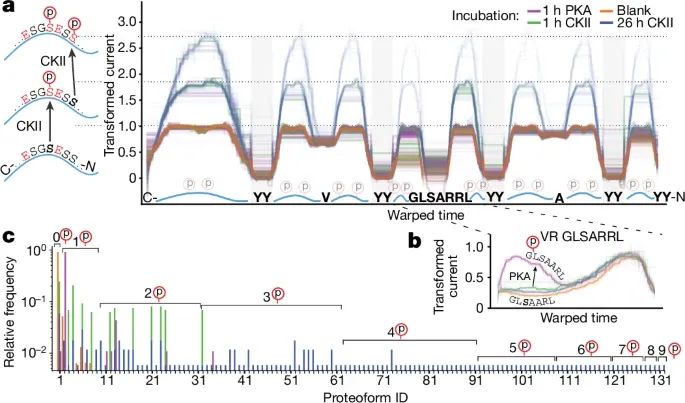
利用PASTOR技术对蛋白质分子中激酶磷酸化进行检测和图谱化的结果(Credit: Nature)
激酶磷酸化的检测模型:图a展示了一个新的PASTOR蛋白(PASTOR-phos),用于特异性检测激酶活性。蛋白质结构中设计了特定的磷酸化位点序列,例如针对蛋白激酶A(PKA)的LRRASLG序列(kemptide),以及含有CKII识别位点的序列。实验目的是通过纳米孔技术检测这些位点的磷酸化状态。
PKA的磷酸化检测:图b展示了PASTOR-phos蛋白在PKA激酶处理后产生的电流信号。检测到kemptide序列区域的电流显著增加,这与磷酸化后负电荷增加导致电流变化相吻合。98.9%的读取显示kemptide区域发生了磷酸化,而其余非kemptide区域的信号几乎没有变化,表明PASTOR技术能够特异性地检测PKA对LRRASLG序列的磷酸化作用。
CKII的磷酸化检测:当PASTOR-phos蛋白与CKII激酶孵育后,图a中的链接序列中,含有CKII识别位点的电流信号同样出现了显著变化。与PKA相比,CKII的磷酸化在八个不同的链接序列上均检测到了更高的电流峰值。更长时间的CKII处理(26小时)进一步增强了这些区域的磷酸化信号,这表明CKII在这些位点的磷酸化反应是特异性的并且可以累积。
蛋白质磷酸化图谱的绘制:图c展示了磷酸化修饰后的不同蛋白质形态的相对频率。研究者能够根据电流信号的变化,区分出单个分子中的不同磷酸化状态。特别是经过26小时CKII处理后,蛋白质分子中可能包含多达九个不同的磷酸化位点。通过PASTOR技术,研究者绘制出了不同条件下蛋白质的磷酸化图谱,表明该技术能够精准量化并检测蛋白质分子上的多种磷酸化状态。
PASTOR技术在读取折叠状态下的蛋白质结构域方面的能力(Credit: Nature)
折叠蛋白质的读取示例:图a展示了使用PASTOR技术对折叠蛋白质结构域进行连续读取的实验设计。研究者选用了绿色荧光蛋白(GFP)作为实验对象,GFP具有稳定的β折叠结构,其紧密的折叠状态对蛋白质解折和测序带来了挑战。通过引入ClpX解折酶,PASTOR技术能够逐步解折GFP的结构,并通过纳米孔测序检测其电流信号。
GFP的电流信号变化:图b展示了GFP蛋白质在解折和通过纳米孔时的电流信号变化。在GFP的折叠区域,通过ClpX逐步解折蛋白质链时,电流信号显示出较大的波动。这些波动反映了GFP的不同折叠区域在被解折过程中穿过纳米孔时的物理性质变化。实验结果显示,PASTOR技术可以实时跟踪并检测到蛋白质解折过程中不同折叠结构的动态变化。
多重结构域蛋白质的读取:图c展示了一个含有多个折叠结构域的蛋白质(包括三个GFP结构域)的读取结果。通过PASTOR技术,研究者能够依次读取每个GFP结构域的电流信号,并区分不同结构域的解折过程。每个结构域的解折步骤表现出类似的电流波动,证明PASTOR技术能够稳定且连续地读取多重折叠结构域蛋白质。
折叠蛋白质的读取准确性:图d展示了不同折叠结构域的电流信号统计数据。实验中,PASTOR技术在多次重读的情况下,能够精确检测和区分各个GFP结构域的电流特征,并且这种检测具有高度的可重复性。这表明该技术在面对复杂折叠蛋白质时,依然能够保持较高的读取准确性。
这项研究展示了PASTOR技术在单分子水平上识别完整蛋白质序列并表征翻译后修饰的潜力,为未来蛋白质测序和条形码技术的应用提供了有力的支持。研究结果表明,该技术有望在高通量蛋白质功能研究和精准医学领域发挥重要作用,尤其是在疾病标志物的检测和治疗靶点的发现方面。
参考文献
Motone K, Kontogiorgos-Heintz D, Wee J, Kurihara K, Yang S, Roote G, Fox OE, Fang Y, Queen M, Tolhurst M, Cardozo N, Jain M, Nivala J. Multi-pass, single-molecule nanopore reading of long protein strands. Nature. 2024 Sep 11. doi: 10.1038/s41586-024-07935-7. Epub ahead of print. PMID: 39261738.
https://www.nature.com/articles/s41586-024-07935-7


