“绘”解读真报告丨实体瘤全外显子组基因检测“小题大做”?真实案例多角度告诉你“并非如此”!
时间:2024-09-16 18:00:31 热度:37.1℃ 作者:网络
实体瘤全外显子组基因检测(WES),顾名思义,能够检测人类2万多个基因的外显子编码区域,相当于一些panel检测的扩容版。可以这么理解,如果把人类2万个基因作为一个基因库,panel检测只是从中挑出一部分与肿瘤发生/用药关系更为紧密的基因进行检测,而WES项目则是不挑不检,直接全库检尽!有点“宁愿多检费钱,不愿放弃一个基因位点”的意思。
于是,您可能产生质疑:使用WES项目,检测那么多基因有什么意义呢?是不是小题大做?
针对这类问题,您是否记得,我们半年前,曾以肺癌检测为例,做过一期解读介绍。
那么现在,我们再结合一些真实案例,探讨下肿瘤患者做WES基因检测为何并非小题大做?
案例一、WES检出了内含子上的融合变异,您先不要质疑
啥?!实体瘤WES不是针对基因外显子的检测么,咋位于内含子区域的融合变异也能检出呢?没搞错吧?
是的,可以理解,了解基因分子结构的您,一定会发出这样的质疑!而我通过报告实例来回答您,确实没搞错!
实际上,这涉及到了WES检测产品的设计。我司“实体瘤全外显子组基因检测”项目在覆盖了2万+个基因的全部外显子之外,还额外在56个基因的内含子区域,以及位于TERT基因间区的启动子位点,均设计了捕获探针。此外,众所周知,位于基因内含子-外显子剪接区域的位点(splice region site),通常也是功能区域所在,我司WES探针设计也精准覆盖,测序深度按照指南规范至少500×,如图1所示。
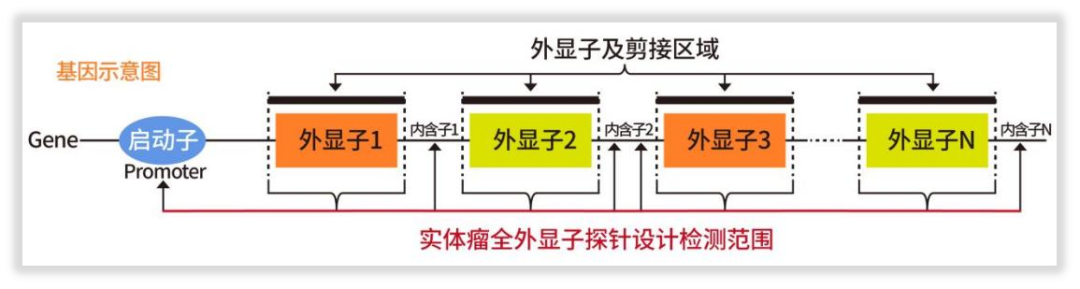
图1 实体瘤全外显子探针设计检测范围
正是这些探针的精心设计,那些肿瘤发生、预后及用药的关键融合变异和剪接区域等,也能一并覆盖。同时,检测覆盖的全面了,检测指标也会更加丰富,还能够精准检出HLA分型/杂合型缺失、肿瘤突变负荷(TMB,全外显子为金标准)、肿瘤新生抗原等多个重要指标!这对于患者的检测指导超出了预期,且大有裨益!
因此,我司“实体瘤全外显子组基因检测”实则是结合肿瘤的临床检测需求,进行了区域/位点检测的优化和增补,更多地为临床检测提供服务。那么,话说至此,我司WES项目既往检出过融合变异么?接下来,且看一则真实报道:
案例一解读
一位女性肺腺癌患者送检我司“实体瘤全外显子组基因检测”项目,经系统的质控评估和检测分析,报告中检出SDC4-ROS1融合变异,并按照规范依据证据级别给予了靶向用药提示。

图2 WES报告检出SDC4-ROS1融合变异用药指导
如果您质疑检测到底检测是否准确?我来从检测原始数据入手,调取下检测变异的IGV图,展示如图3。患者检出情况为SDC4基因(chr20)4号外显子和ROS1基因(chr6)32号外显子形成融合基因,我司报告中也细致地给出两个基因外显子的融合情况和解读,辅以知其然,知其所以然,还能够满足一些临床老师的科研写作需求呢。
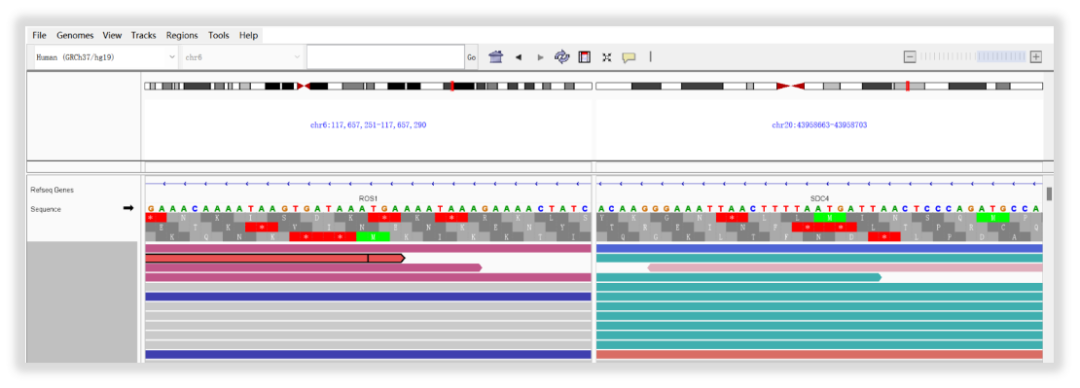
图3 SDC4-ROS1融合变异原始IGV图
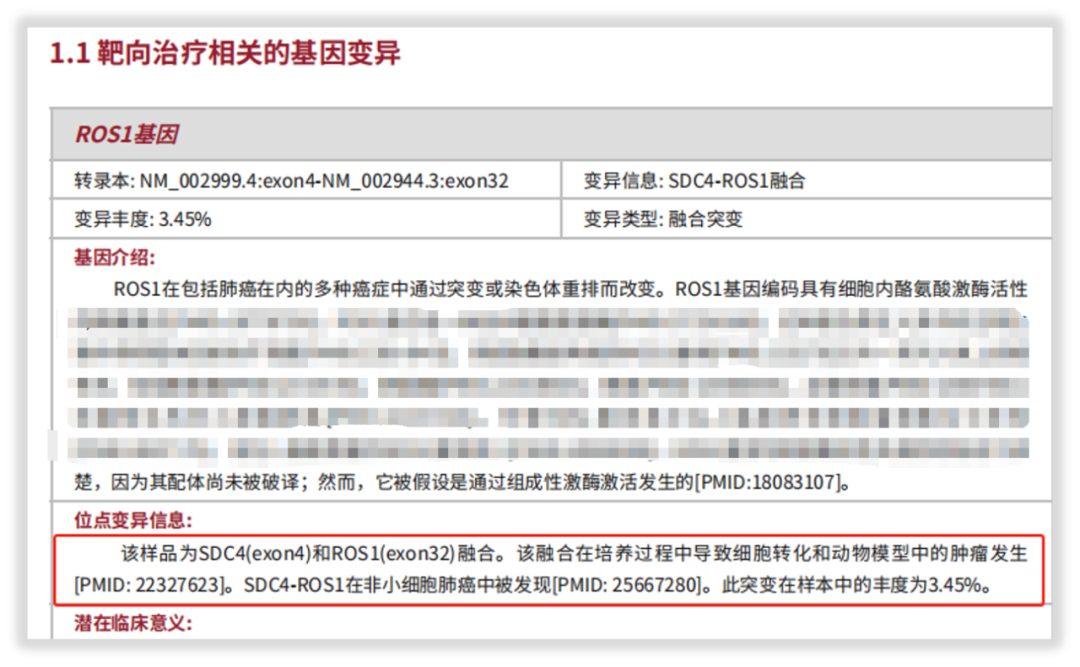
图4 SDC4-ROS1融合变异详细信息解读
通过这个案例,不仅证实了我司WES检测能够切实检出一些关键的融合基因,更体现出检测的准确性及报告解读的细致性!
案例二、别只因未匹配上用药而失望,更多的信息在指导您呢
当我们提到实体瘤全外显子检测项目时,你可能会发出灵魂拷问,检测那么多的内容,很多都匹配不上靶向治疗,是不是纯粹的浪费?
确实,基于目前的科学研究,相对人类的2万多个基因,目前获批药物治疗的基因靶点寥寥无几,就那么几个。当然,有药可用的基因检测毋庸置疑,但说实话,人类每个基因的功能都不应该被忽视,其他基因的检测并非“无用功”。
基于目前科学研究,很多基因已经被明确为肿瘤发生发展相关基因,虽然针对性的药物治疗,仍在科研探索阶段,但肿瘤患者的治疗过程不止是关注用药指导。举个例子,最知名的TP53基因突变。多年来,TP53靶向疗法“屡战屡败”,铸就了TP53的“不可成药”之说。是不是检测它就很没有必要呢?实则非也,除了靶向治疗之外,TP53基因突变更重要的可能在预后(差)、复发风险(高)、免疫治疗(疗效相对好)、靶向治疗(疗效差)、遗传(李法美尼综合征)等多个角度均能给出重要指导,方便临床做出更好的患者管理,依然能为患者甚至于其家人带来诸多获益,其实很多罕见基因也是这样,只不过TP53比较常见,很多panel都会纳入[1-2]。
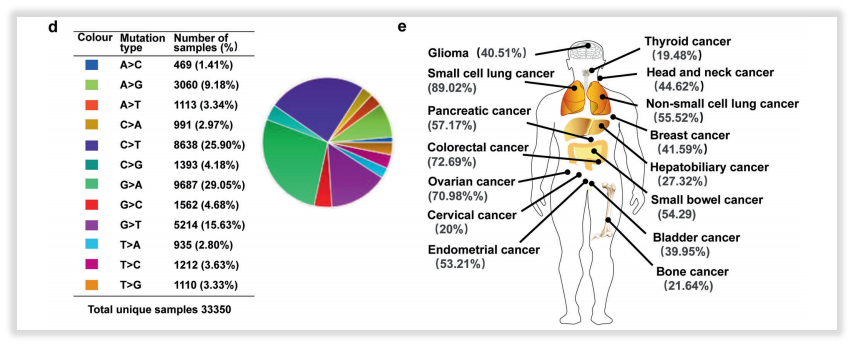
图5 TP53基因突变及不同肿瘤的发生情况
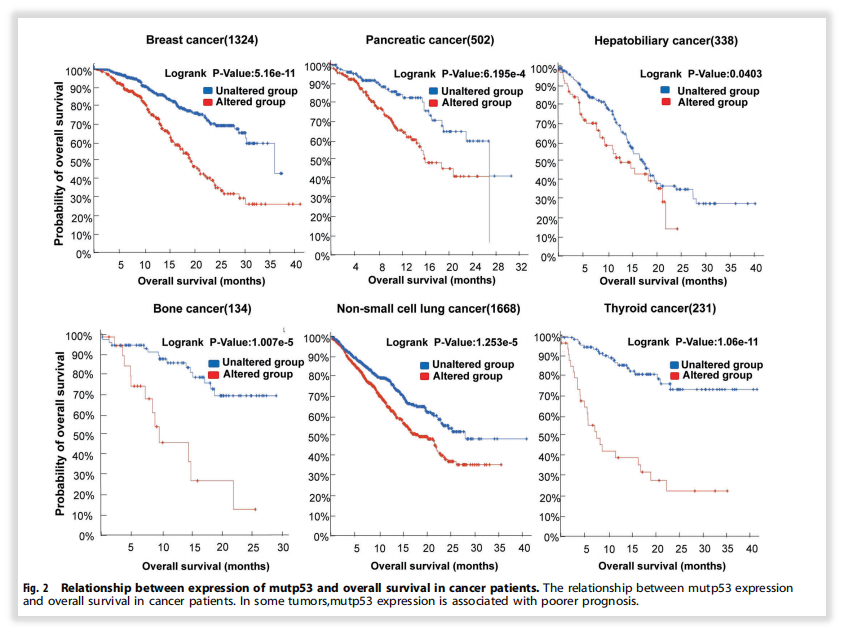
图6 TP53基因突变在不同肿瘤中的预后指导
再举一个陌生的,RBM10这个基因,检测他有个啥用?因为陌生,而被忽视,很多检测小panel都不会覆盖,但WES是必然覆盖的!我们以真实的WES报告来看下。
案例二解读
一例71岁男性肺腺癌患者,通过我司“实体瘤全外显子组基因检测”检出RBM10基因截短突变,并对其做出细致解读。该突变非常罕见,据报道在COSMIC数据库中,该突变在肺癌中仅被记录1次。
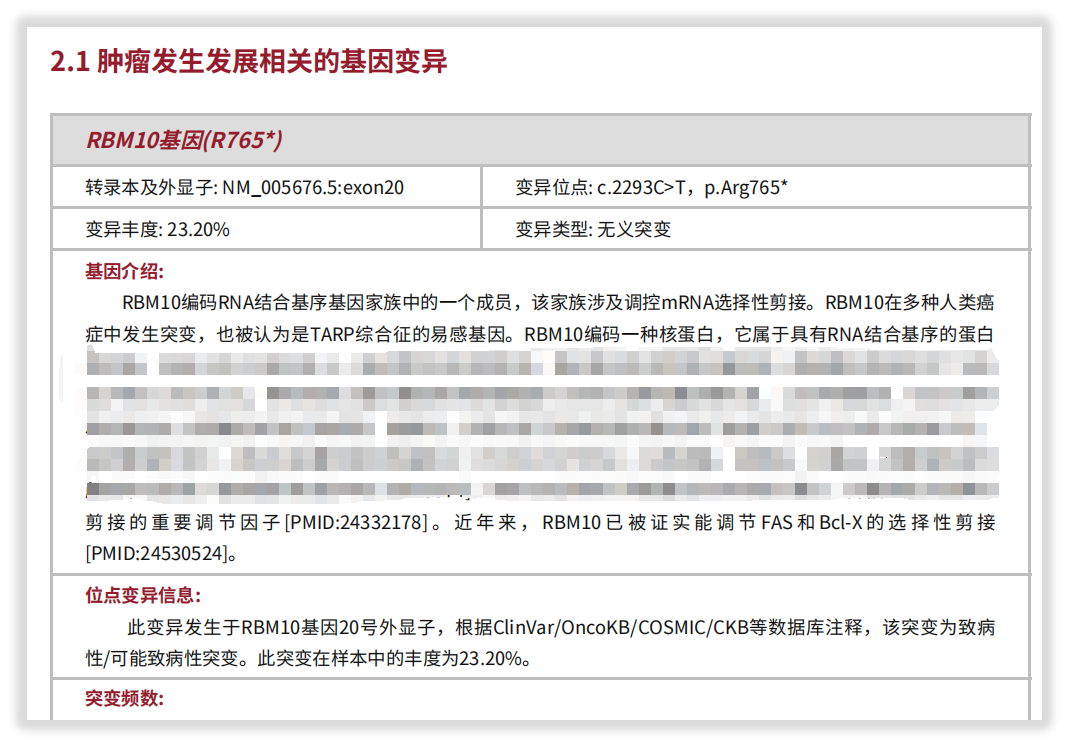
图7 患者检出RBM10基因截短突变(部分)
现阶段,RBM10基因尚无相关用药匹配,但一项最新研究,在50位患者中的研究证实,它是影响肺癌患者预后的重要因素[3]。RBM10基因突变常伴随多个癌症发展的基因突变,与更具侵袭性的疾病和更差的总体预后有关,大多数患者的疾病进展迅速。患者生存率上,RBM10突变队列的中位PFS为6.7个月,而非突变队列的中位PFS为13.9个月(图1A)然而,RBM10突变与EGFR突变共存时,EGFR-TKI治疗似乎更具保护性,可使病情稳定时间更长。这些为临床患者带来的指导信息,往往也对医生的治疗方案起着关键作用!而深知这些,我司除了在报告层面说明之外,还在积极撰写相关科普推文,提供更多的参考!
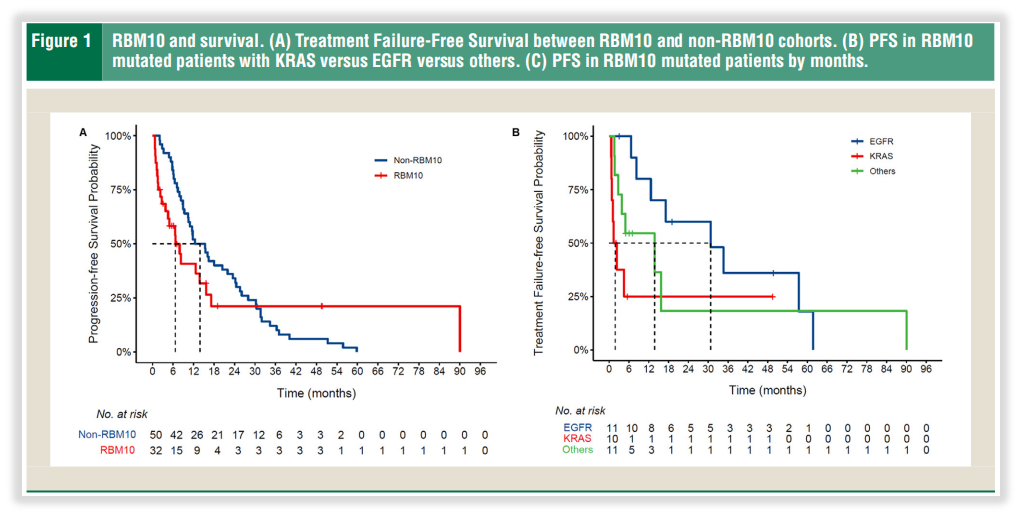
图8 RBM10基因突变影响肺癌患者预后
类似地拓展一下,我们都知道,临床意义未明突变(VUS),如共识里介绍:尚不足以判定该变异的功能影响或临床意义,但并非一成不变,不排除将来随着研究的深入和证据的积累,该变异的临床意义得以进一步划分[4]。而这些尚无用药的发生发展基因,亦是如此!您在获得指导的同时,请保持希望,随着药物研发的深入,期待不久的将来,必然有良好的针对性指导药物问世!
案例三、即便这个位点COSMIC未收录,也要如实提供意义解读
当你得知检测覆盖某个基因时,针对这个基因的全面性也是重要的一环。这里就不得不说WES检测的优势之一,它不分伯仲地检测2万多个基因的所有外显子。这对于一些抑癌基因的检测,是非常友好的!因为抑癌基因通常没有热点突变,截短突变可能发生于任意位点(有些基因可能高达上万位点),但数据库可能收录不了基因那么多的位点。因此,倘若您在报告中发现“在COSMIC数据库中,尚未收录该突变”,您也切莫惊讶!我来上案例,给您提供那些突变背后的故事。
案例三解读
一例来自国内某三甲医院的患者,经临床反馈为软组织肿瘤,考虑到患者的疾病表现进展快、高侵袭性、以及更全面的治疗方案指导等多重原因,送检我司“实体瘤全外显子组基因检测”。后来检出KDM6A L1171Sfs*32移码截短突变,报告提示“在COSMIC数据库中,尚未收录该突变”。
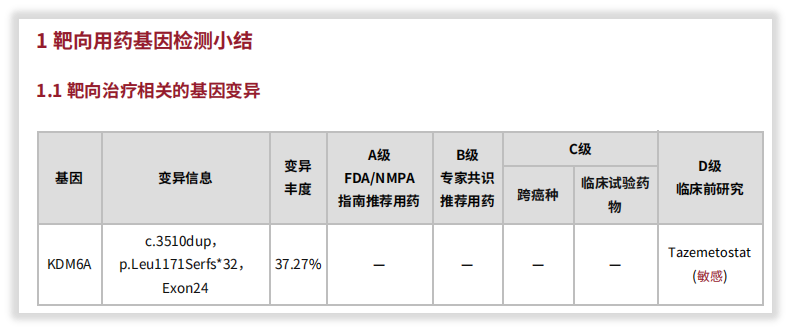
图9 患者检出KDM6A L1171Sfs*32突变
为确保检测的准确性,在出具报告时,就针对这个突变原始数据,调取了其位点IGV示意图(基因组变异:GRCh37(chrX):g.44942774dup)。图中显示在chrX:g.44942774位置插入了碱基T,很直观,很准确!
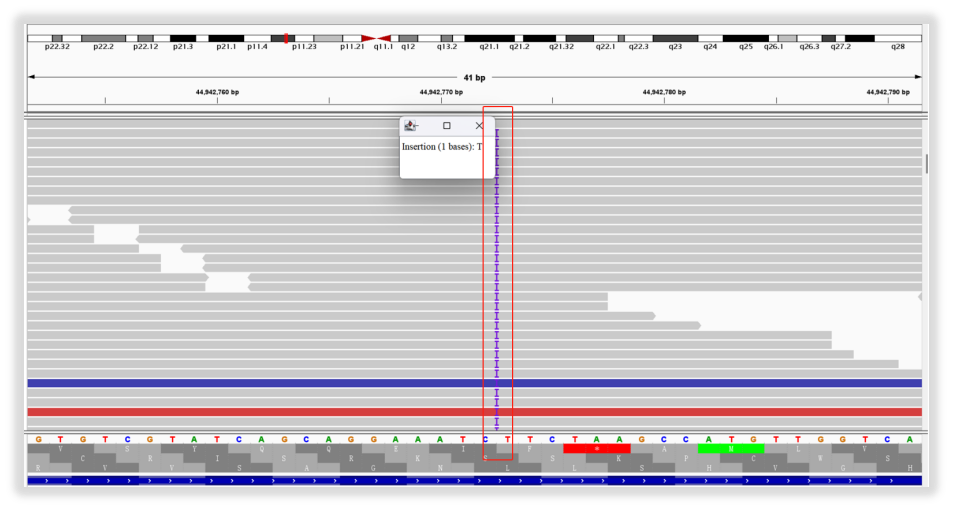
图10 KDM6A L1171Sfs*32突变IGV视图
进一步将该突变根据HGVS的转换,在氨基酸层面发生了p.(Leu1171Serfs*32),与我司报告中一致。检测结果完全属实!
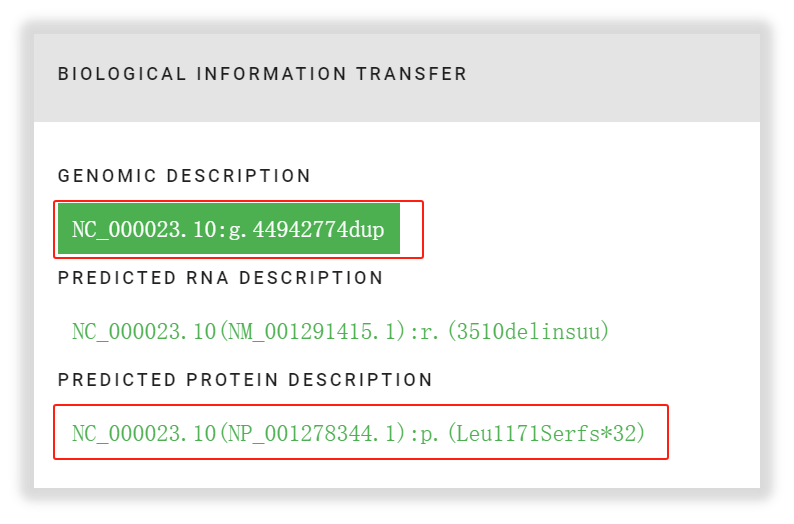
图11 KDM6A L1171Sfs*32突变的转换
了解了这些后,您可能还会惊讶,这个COSMIC都没报道的位点,我司报告中竟然给出了用药(Tazemetosta,D级别),并注明是根据OncoKB数据库。那这又是为什么呢?
为了知根知底,我们打开OncoKBTM数据库,输入KDM6A基因。哦,原来如此,KDM6A的用药指导,针对的是Truncating Mutations(截短突变)大类,而KDM6A L1171Sfs*32,正是截短突变。恍然大悟!试想我们常见的其他抑癌基因,诸如BRCA1/2、TP53,不也是这种情况么?
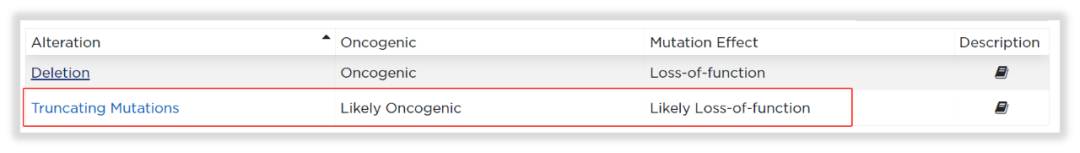
图12 KDM6A基因在OncoKB数据库的致癌突变收录
随后针对用药的进一步检索发现,OncoKBTM确有报道:携带KDM6A基因截短突变的细胞系对Tazemetostat敏感。由于证据来源是临床前的细胞系研究[5],所以,报告按规范将其推荐为D级别。同时WES报告检出的基因突变,也会纵览国内外临床试验资料库(如ClinicalTrials.gov、ChiCTR等),给出一些临床试验参考,再从临床试验角度提供更多信息!
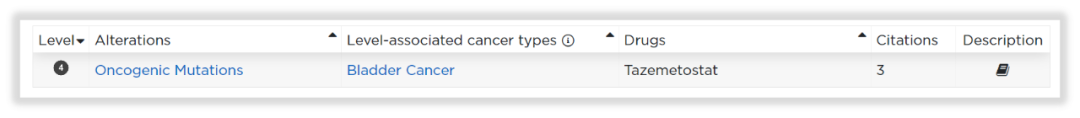
图13 KDM6A基因在OncoKB数据库的用药指导收录

图14 携带KDM6A基因截短突变的细胞系对Tazemetostat敏感[5]
总结来看,实体瘤全外显子组基因检测并非小题大做,请不要忽视它给患者提供的巨大力量。
因为它,更多的基因突变和关键指标不会遗漏,弥补热点PANEL基因检测的不足,更加全面;
因为它,更多的肿瘤发生发展、预后、遗传相关的基因突变被发现,且指导更多的获益;
因为它,晚期难治且治疗方案急缺的患者,可能迎来意想不到的转机,遗憾会少一些!
让我们一起了解它、接受它、使更多肿瘤患者获益!
参考文献:
[1]何建行梁文华.基于分子标志的非小细胞肺癌术后复发预测专家共识[J].中国肺癌杂志, 2022, 25(10):701-714.
[2]Chen X, Zhang T, Su W, Dou Z, Zhao D, Jin X, Lei H, Wang J, Xie X, Cheng B, Li Q, Zhang H, Di C. Mutant p53 in cancer: from molecular mechanism to therapeutic modulation. Cell Death Dis. 2022 Nov 18;13(11):974. doi: 10.1038/s41419-022-05408-1. PMID: 36400749; PMCID: PMC9674619.
[3]Reyes A, Afkhami M, Massarelli E, et al. RBM10 Mutation as a Potential Negative Prognostic/Predictive Biomarker to Therapy in Non-Small-Cell Lung Cancer. Clin Lung Cancer. Published online July 23, 2024. doi:10.1016/j.cllc.2024.07.010
[4]中国三级妇产科医院/妇幼保健院联盟妇科肿瘤遗传咨询协作组,中国抗癌协会中西整合子宫内膜癌专业委员会,徐丛剑,等.妇科肿瘤遗传咨询专家共识(2023年版)[J].中国癌症杂志, 2023, 33(11):1041-1064.
[5]Ler LD, Ghosh S, Chai X, Thike AA, Heng HL, Siew EY, Dey S, Koh LK, Lim JQ, Lim WK, Myint SS, Loh JL, Ong P, Sam XX, Huang D, Lim T, Tan PH, Nagarajan S, Cheng CW, Ho H, Ng LG, Yuen J, Lin PH, Chuang CK, Chang YH, Weng WH, Rozen SG, Tan P, Creasy CL, Pang ST, McCabe MT, Poon SL, Teh BT. Loss of tumor suppressor KDM6A amplifies PRC2-regulated transcriptional repression in bladder cancer and can be targeted through inhibition of EZH2. Sci Transl Med. 2017 Feb 22;9(378):eaai8312. doi: 10.1126/scitranslmed.aai8312. PMID: 28228601.



