【论著】| 肺腺癌中可变剪接调控因子KHSRP调控的差异剪接基因分析
时间:2023-08-21 11:39:16 热度:37.1℃ 作者:网络
[摘要] 背景与目的:可变剪接在细胞增殖、生长、凋亡及分化等复杂的蛋白质功能调控中发挥着至关重要的作用,能参与包括肿瘤在内的多种疾病的发生、发展过程,目前大多数可变剪接调控因子的功能及其作用机制尚不清楚。本研究旨在探讨KH型剪接调控蛋白(KH-type splicing regulatory protein,KHSRP)在肺腺癌中的差异剪接靶基因及靶基因的表达与患者预后及免疫细胞功能的相关性。方法:通过高通量可变剪接测序,筛选KHSRP下游差异剪接靶基因。通过肿瘤免疫评估资源(Tumor IMmune Estimation Resource,TIMER)和阿拉巴马大学伯明翰分校癌症数据分析门户(the University of Alabama at Birmingham cancer data analysis portal,ULCAN)分析KHSRP下游差异剪接靶基因在肺腺癌中的表达。通过Kaplan数据库分析KHSRP下游差异剪接靶基因的表达与患者预后的相关性。通过TIMER免疫模块分析KHSRP下游差异剪接靶基因与免疫细胞功能的相关性。结果:NUMB、ADD3及LIMCH1为KHSRP的下游关键可变剪接靶基因。NUMB、ADD3及LIMCH1在肺腺癌肿瘤组织中均呈低表达,其低表达者预后不良。NUMB、ADD3及LIMCH1与免疫细胞功能呈正相关。结论:在肺腺癌中,NUMB、ADD3及LIMCH1为KHSRP的差异剪接靶基因,且NUMB、ADD3及LIMCH1低表达会导致预后不良,并且与免疫细胞功能呈正相关。
[关键词] 肺腺癌;KH型剪接调控蛋白;可变剪接;差异剪接靶基因移
[Abstract] Background and purpose: Alternative splicing plays a crucial role in the functions and regulation of complex proteins such as cell proliferation, growth, apoptosis and differentiation, and can cause the occurrence and development of various diseases, including cancer. Currently, the functions and molecular mechanisms of most alternative splicing factors are not clear. The purpose of this study was to explore the differential splicing target genes of KH-type splicing regulatory protein (KHSRP) in lung adenocarcinoma and their correlation with prognosis and immune cell function. Methods: Screening downstream differential splicing target genes of KHSRP was carried out through high-throughput variable splicing sequencing. We analyzed the expressions of KHSRP downstream differential splicing target genes in lung adenocarcinoma through cancer databases Tumor IMmune Estimation Resource (TIMER) and the University of Alabama at Birmingham cancer data analysis portal (ULCAN). We also analyzed the correlation between the expression of differential splicing target genes downstream of KHSRP and prognosis through Kaplan database. The correlation between downstream differential splicing target genes of KHSRP and immune cell function was analyzed through the TIMER immune module. Results: NUMB endocytic adaptor protein (NUMB), adducin 3 (ADD3) and LIM and calponin homology domains 1 (LIMCH1) were key downstream variable splicing target genes of KHSRP. NUMB, ADD3 and LIMCH1 were all expressed at low level in lung adenocarcinoma tumor tissue. Low expressions of NUMB, ADD3 and LIMCH1 in lung cancer leaded to poor prognosis. NUMB, ADD3 and LIMCH1 were positively correlated with immune cell function. Conclusion: In lung adenocarcinoma, NUMB, ADD3 and LIMCH1 are differential splicing target genes of KHSRP, their low expression levels indicate poor prognosis, and they are positively correlated with immune cell function.
[Key words] Lung adenocarcinoma; KH-type splicing regulatory protein; Variable splicing; Differential splicing target genes
肺癌是全球范围内发病率和死亡率最高的恶性肿瘤。2018年,全球癌症统计报告估计有209万肺癌新发病例和176万肺癌死亡病例[1]。肺癌分为非小细胞肺癌和小细胞肺癌,其中非小细胞肺癌占全部肺癌的80%~90%,包括肺鳞癌和肺腺癌,其中肺腺癌为最常见的病理学类型。虽然目前关于肺腺癌的治疗已取得较大进步,但是由于肺腺癌患者的个体差异性、表皮生长因子受体(epidermal growth factor receptor,EGFR)突变及对化疗药物的耐药性,导致其死亡率仍较高,因此急需发掘肿瘤特异性新抗原,从而为肺腺癌治疗提供新靶点。
可变剪接也称为选择性剪接,可使前体mRNA产生不同的mRNA剪接异构体,导致不同的蛋白产物形成,从而形成新的细胞表位。作为基因调控较为普遍的机制之一,可变剪接在细胞增殖、生长、凋亡及分化中发挥着至关重要的作用,参与包括肿瘤在内的多种疾病的发生、发展过程[2]。在胰腺癌[3-4]、卵巢癌[5-6]、乳腺癌[7-8]、黑色素瘤[9-10]和胶质母细胞瘤[11-12]等多种肿瘤中均发现了大量高信度的可变剪接事件,剪接调控因子在过表达或敲减后,能够引起可变剪接的发生,从而导致细胞致瘤特性的改变。KH型剪接调控蛋白(KH-type splicing regulatory protein,KHSRP)在调节RNA剪切、RNA转运、RNA编辑、mRNA稳定及降解等过程中均具有重要作用,从而调控细胞增殖、分化、生长、凋亡及肿瘤形成等多种生物学过程[13]。目前,KHSRP与异质性核糖核蛋白C(heterogeneous nuclear ribonucleoprotein C,HNRNPC)相互作用后如何调控下游靶分子,从而影响Janus激酶-信号转导及转录激活因子(Janus kinase-signal transducers and activators of transcription,JAK-STAT)信号通路变化的具体机制尚未阐明。考虑到KHSRP对可变剪接的可能调控作用,本研究旨在研究KHSRP对下游差异可变剪接基因的调控,以确定KHSRP是否通过调控下游基因可变剪接促进肺腺癌进展。
1 资料和方法
1.1 细胞总RNA提取及反转录
研究中使用的所有细胞样本的总RNA均采用TRIzol试剂(美国Invitrogen公司)提取,然后使用PrimeScript RT试剂(日本TaKaRa公司)进行反转录获得cDNA。
1.2 聚合酶链反应(polymerase chain reaction,PCR)
以cDNA为模板,设计ADD3、LIMCH1及NUMB引物,采用PrimeScript RT试剂进行PCR,15 μL PCR反应体系如下:3 μL 5×PrimeSTAR、1.2 μL脱氧核苷三磷酸(deoxyribonucleoside triphosphate,dNTP)、10 μmol/L正向引物和反向引物各0.6 μL,使用稀释10倍的反转录产物 1.5 μL作为模板,将0.15 μL DNA聚合酶用ddH2O补至15 μL。按照以下程序进行PCR:98℃预变性2 min;98℃变性10 s,退火5~15 s(退火温 度≥55℃,5 s;退火温度<55℃,15 s),72℃延伸30 s,35个循环;72℃终延伸5 min。所使用的基因引物序列如下:ADD3正向引物序列为GCTCCTCCTAACCCATTT,反向引物序列为CATATCATCCTTGCCATTTA;LIMCH1正向引物序列为AACCAGTACCTCCCGAACA,反向引物序列为CCAACGAGCCAGGTCATC;NUMB正向引物序列为CTCCGATGACCAAACCAG,反向引物序列为GGACGCTCTTAGACACCTC。引物均由苏州金唯智生物科技有限公司合成。
1.3 DNA非变性聚丙烯酰胺凝胶电泳
配制8%非变性聚丙烯酰胺凝胶进行电泳,10 mL凝胶液配方如下:5.224 mL H2O,2.666 mL 30%丙烯酰胺(北京兰博利德生物技术有限公司),2 mL 5×TBE(上海碧云天生物技术有限公司),100 μL 10%APS(上海碧云天生物技术有限公司),10 μL TEMED(上海碧云天生物技术有限公司)。以1×TBE作为缓冲液,120 V电泳30 min左右,电泳完成后,将凝胶浸泡于3×核酸染料[天根生化科技(北京)有限公司]中,30 min后用凝胶成像仪拍照。
1.4 数据库分析
可变剪接差异靶基因在肺腺癌组织中的转录组表达水平采用肿瘤免疫评估资源(Tumor IMmune Estimation Resource,TIMER)数据库(https://cistrome.shinyapps.io/timer/)和阿拉巴马大学伯明翰分校癌症数据分析门户(the University of Alabama at Birmingham cancer data analysis portal,ULCAN)-癌症基因组图谱(The Cancer Genome Atlas,TCGA)数据库(https://ualcan.path.uab.edu/analysis.html)分析;采用肺腺癌Kaplan网站分析基因与肺腺癌患者预后的相关性;采用TIMER Gene(https://cistrome. shinyapps.io/timer/)分析基因与肺腺癌中免疫细胞功能的相关性。
1.5 Transwell迁移和侵袭实验
用于迁移和侵袭实验的小室购自美国Corning公司。迁移实验将空穿小室插入24孔板中,下室加入800 μL含有10%胎牛血清(fetal bovine serum,FBS)的细胞培养基作为化学引诱剂,上室加入0.5×105个重悬于200 μL无血清细胞培养基中的肺癌细胞。侵袭实验将胶穿小室插入24孔板中,下室加入800 μL含有10%FBS的细胞培养基作为化学引诱剂,上室加入1×105个重悬于200 μL无血清细胞培养基中的肺癌细胞。迁移实验的肺腺癌细胞培养16 h,侵袭实验的肺腺癌细胞培养18 h,用棉签擦去小室上室的细胞,用甲醇溶液固定从小室上室通过膜迁移到小室下室的细胞30 min,并使用结晶紫染色液染色细胞20 min,晾干后,对染色细胞进行拍照计数。
1.6 统计学处理
本研究中两组数据之间的比较均采用GraphPad Prism 9软件中的双侧Student t检验,通过Kaplan-Meier Plotter数据库(http://kmplot.com/analysis/)分析基因与肺腺癌患者的预后相关性。P<0.05为差异有统计学意义。
2 结 果
2.1 KHSRP调控的可变剪接事件及信号通路
分别提取A549细胞的KHSRP-siRNA(2条干扰片段)及对照载体的总RNA,采用RNA-Seq高通量测序和生物信息学分析,鉴定KHSRP敲除后发生的可变剪接事件。结果共鉴定出356个KHSRP调控的可变剪接事件[以伪发现率(false discovery rate,FDR)<0.05,P<0.05作为判断标准],样品中有外显子跳跃、5’末端选择性剪接、3’末端选择性剪接等7种类型的可变剪接,其中,外显子跳跃出现221个,占总数的62.08%。生物信息学分析发现主要集中在肿瘤坏死因子(tumor necrosis factor,TNF)、紧密连接、肌动蛋白细胞骨架调节等信号通路(图1)。

图1 KHSRP调控的下游可变剪接事件
Fig. 1 Downstream alternative splicing events regulated by KHSRP
A: Identification of alternative splicing events and types after KHSRP knockdown by RNA sequencing; B: Analysis of signaling pathways regulated by KHSRP.
2.2 KHSRP调控的差异剪接靶基因
为进一步验证RNA-Seq测序结果的可靠性,因外显子跳跃是最常见的可变剪接类型,本研究挑选了测序结果中KHSRP敲低后的前20个可变剪接靶基因,按照筛选条件(FDR<0.05,P <0.05)初步筛选到14个外显子跳跃类型的可变剪接靶基因,进一步通过IGV软件查询外显子的表达水平,排除肉眼可见的表达水平非常低的基因,从而缩小感兴趣基因的范围,通过以上方法,我们最终找到3个基因作为感兴趣的KHSRP差异剪接靶基因。经反转录(reverse transcription,RT)-PCR电泳证实在A549细胞中敲除KHSRP后,ADD3的17号外显子和NUMB的13号外显子表达显著增加,并且Uniprot查询外显子序列发现,ADD3的17号外显子和NUMB的13号外显子均存在于基因的经典转录本上,提示包含17号外显子的经典转录本ADD3 L-异构体和包含13号外显子的经典转录本NUMB L-异构体可能发挥抑癌作用;而LIMCH1的20号外显子表达显著下降,并且Uniprot查询外显子序列发现,LIMCH1的20号外显子不在基因的经典转录本,提示包含20号外显子的LIMCH1 L-异构体可能发挥促癌作用,而其不包含20号外显子的经典转录本LIMCH1 S-异构体可能发挥抑癌作用。由此推测ADD3、NUMB及LIMCH1可能为KHSRP调控的差异剪接靶基因,并且在肺腺癌中可能均发挥抑制肺腺癌进展的作用(图2)。
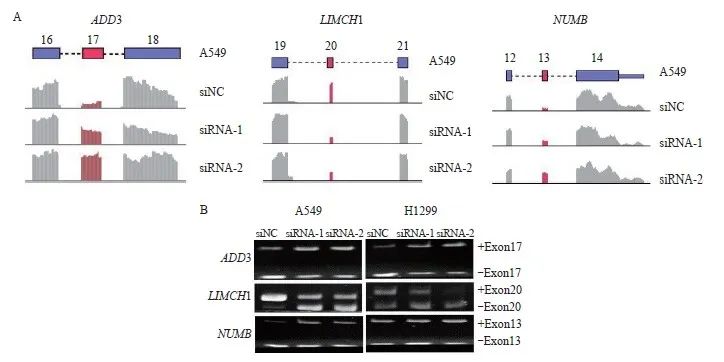
图2 KHSRP调控的下游可变剪接事件验证
Fig. 2 Verification of downstream alternative splicing events regulated by KHSRP
A: Diagram of KHSRP target gene exon skip by RNA-Seq analysis; B: Alternative splicing events of KHSRP target genes by RT-PCR analysis.
2.3 差异剪接靶基因在肺腺癌肿瘤组织和细胞系中的表达水平
为进一步验证这3个基因在肺腺癌中的表达情况,本研究利用TIMER和ULCAN两个癌症数据库进行转录组表达分析,发现ADD3、LIMCH1和NUMB基因在肺腺癌肿瘤组织中均低表达(图3A~3C)。
通过查询美国国家生物技术信息中心(National Center for Biotechnology Information, NCBI)网站(http://www.ncbi.nlm.nih.gov/)发现,ADD3编码2个可变剪接异构体,LIMCH1编码20个可变剪接异构体,NUMB编码4个可变剪接异构体,本研究挑选编码剪接异构体最少的ADD3进行进一步的实验验证。首先,在20对肺癌临床样本cDNA中检测了ADD3可变剪接异构体的转录本表达情况,发现在20对肺癌样本中,ADD3 L/S-异构体在65%肺癌肿瘤组织中的表达高于配对癌旁组织,在10%肺癌肿瘤组织中的表达低于配对癌旁组织,在25%肺癌肿瘤组织中的表达与配对癌旁组织接近(图3D)。另外,在8个肺腺癌细胞系中检测ADD3可变剪接异构体的表达,发现ADD3 L-异构体在H1975、PC9、H292和H358细胞中的表达比S-异构体高,而ADD3 L-异构体在A549、H1299、H838和H1650细胞中的表达比S-异构体少(图3E),ADD3可变剪接异构体在多种肺腺癌细胞系中的表达模式差异显著。
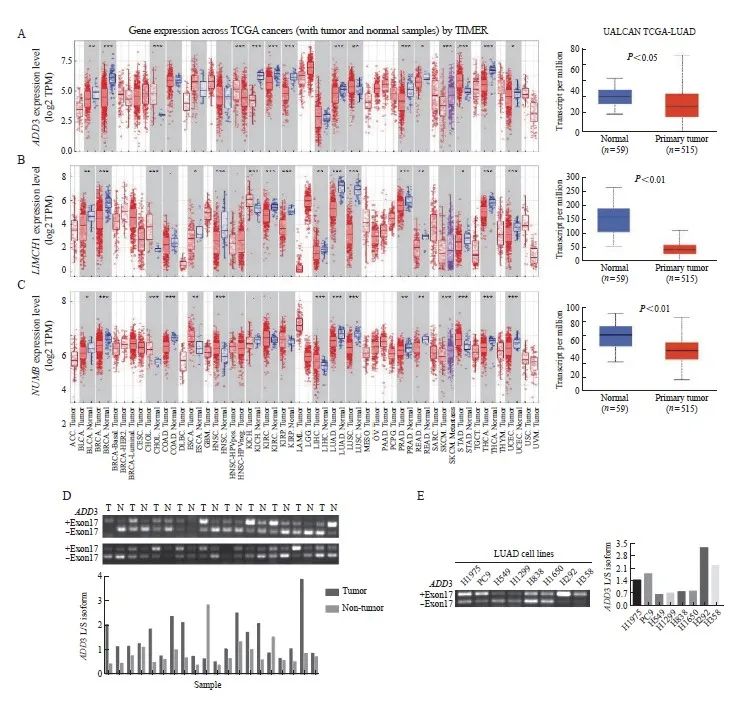
图3 差异剪接基因在肺腺癌中的表达
Fig. 3 Gene expression of different splicing transcript in lung adenocarcinoma
Analysis of ADD3 (A), LIMCH1 (B) and NUMB (C) expression in lung adenocarcinoma tumor tissues and normal tissues using online TIMER and ULCAN database; D: Expression of ADD3 variable splicing isomers in 20 pairs of lung cancer tumor tissues and paired adjacent tissues; E: Expression of ADD3 variable splicing isomers in lung adenocarcinoma cell lines. *: P<0.05; **: P<0.01; ***: P<0.001.
2.4 差异剪接靶基因在肺腺癌肿瘤组织中低表达与患者预后不良有关
为进一步分析ADD3、LIMCH1及NUMB基因与肺腺癌患者的预后相关性,本研究利用肺腺癌生存分析数据库Kaplan进行分析,发现ADD3、LIMCH1和NUMB基因在肺腺癌中低表达与肺腺癌患者预后不良有关,提示ADD3、LIMCH1及NUMB基因与肺腺癌恶性表型相关(图4)。
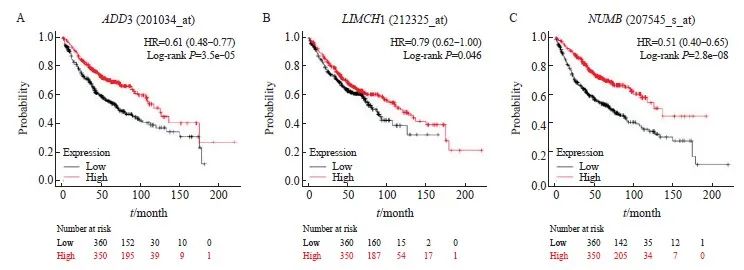
图4 差异剪接基因与肺腺癌预后相关性分析
Fig. 4 Analysis of the correlation between differential alternative genes and overall survival of lung cancer
Kaplan-Meier survival analysis of the correlation between ADD3 (A), LIMCH1 (B), NUMB (C) expression levels and overall survival of lung adenocarcinoma patients was performed.
2.5 差异剪接靶基因与肺腺癌中免疫细胞功能呈正相关
可变剪接和免疫细胞功能在癌症中均发挥重要作用,因此,本研究将KHSRP可能的差异剪接靶基因与肺腺癌中免疫细胞的功能进行分析,发现ADD3、NUMB与B细胞、T细胞、巨噬细胞、中性粒细胞及树突状细胞功能呈正相关,而LIMCH1与巨噬细胞功能呈正相关(图5),提示ADD3、NUMB及LIMCH1可能作为肺腺癌特异性新抗原,在肺腺癌中可能通过调节免疫细胞功能,使免疫系统功能总体呈激活状态,从而起到抑制肺腺癌进展的作用。
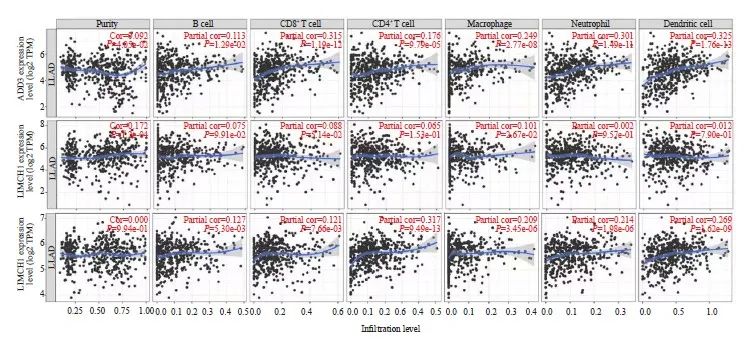
图5 差异剪接基因与免疫细胞功能相关性分析
Fig. 5 Correlation analysis between differential splicing genes and immune cell function
Correlation analysis between ADD3 (A), LIMCH1 (B), NUMB (C) and lung adenocarcinoma immune cell function using the online TIMER database.
2.6 ADD3 L-异构体抑制肺腺癌细胞系A549细胞迁移和侵袭潜能
为进一步验证ADD3可变剪接异构体在肺腺癌中的功能,针对ADD3 L-异构体和S-异构体分别设计了2条siRNA,在A549细胞中敲低L-异构体和S-异构体(图6A),transwell实验发现,敲低ADD3 L-异构体可以促进肺腺癌细胞的体外迁移和侵袭潜能,而敲低ADD3 S-异构体后A549细胞的体外侵袭和迁移潜能无显著变化(图6B),提示KHSRP可能通过下调ADD3基因L-异构体剪接体的表达促进肺腺癌进展。

图6 ADD3可变剪接异构体在A549细胞中对迁移和侵袭潜能的影响
Fig. 6 The effect of ADD3 variable splicing isomers on migration and invasive potential in A549 cells
A: Knockdown efficiency of ADD3 variable splicing isomer in A549 cells; B: Analysis of transwell migration and invasion experiment of knockdown ADD3 variable splicing isomer in A549 cells.*: P>0.05, compared with A549 siNC; **: P<0.01, compared with A549 siNC; ***: P<0.001, compared with A549 siNC.
3 讨 论
选择性剪接的异常变化可能会影响肿瘤进展[14],还可能破坏肿瘤进展中的蛋白质相互作用途径[15-16]。前期研究[17]发现,可变剪接调控蛋白KHSRP在肺癌中显著促进肺癌细胞增殖、侵袭和迁移功能。KHSRP基因编码一种多功能RNA结合蛋白,参与多种细胞生物学过程,包括转录、选择性前信使核糖核酸剪接和信使核糖核酸定位[18]。我们推测KHSRP可能通过调控下游蛋白的可变剪接促进肺腺癌细胞增殖、侵袭及迁移潜能。因此,在肺腺癌细胞A549中敲低KHSRP后,按照筛选条件(FDR<0.05,P<0.05)进行初步筛选,通过IGV软件查询外显子的表达水平,排除肉眼可见的表达水平非常低的基因,从而缩小感兴趣基因的范围,最终找到3种基因作为感兴趣的KHSRP差异剪接靶基因。通过PCR电泳对测序结果进行实验验证,发现ADD3的17号外显子及NUMB的13号外显子表达显著增加,LIMCH1的20号外显子表达显著下降,3个基因均发生显著的外显子跳跃现象,提示KHSRP可能通过调控ADD3、NUMB及LIMCH1的外显子跳跃这一最常见的可变剪接类型进而影响肺癌进展。2013年Bechara等[19]发现NUMB在肺癌细胞中发生可变剪接,并被RNA结合蛋白RBM5/6/10调控;2021年Wang等[20]发现细胞骨架蛋白ADD3被RNA结合蛋白QKI-5调控从而影响肺癌进展,这两项研究与本研究相呼应,进一步说明本研究的可变剪接测序结果是可靠的。本研究发现并证实ADD3、NUMB及LIMCH1在肺腺癌中作为KHSRP的可变剪接靶基因影响肺腺癌进展。
通过Uniprot网站查询发生外显子跳跃的序列,判断包含跳跃外显子的L-异构体是否位于基因的经典转录本上,发现ADD3的17号外显子和NUMB的13号外显子均存在于基因的经典转录本上,提示包含17号外显子的经典转录本ADD3 L-异构体和包含13号外显子的经典转录本NUMB L-异构体可能发挥抑癌作用;而LIMCH1的20号外显子则不在基因的经典转录本上,提示不包含20号外LIMCH1 S-异构体为经典转录本,可能发挥抑癌作用。由此推测ADD3、NUMB及LIMCH1为KHSRP调控的差异剪接靶基因,并且在肺腺癌中可能均发挥抑制肺腺癌进展的作用。为验证这一猜想,本研究分别通过TIMER-Diff EXP基因表达模块和ULCAN-TCGA模块分析了ADD3、NUMB及LIMCH1在肺腺癌中的表达情况,发现这3种基因在肺腺癌中均呈低表达状态。另外,本研究进一步检测了本实验室收集的20对肺癌临床样本中ADD3的可变剪接异构体的表达情况,ADD3 L/S-异构体在65%肺癌肿瘤组织中的表达高于配对癌旁组织,在10%肺癌肿瘤组织中的表达低于配对癌旁组织,在25%肺癌肿瘤组织中的表达与配对癌旁组织接近,此结果与上述数据库表达分析结果不一致,原因可能是本研究的临床样本量较少,需要扩大样本量以进一步验证。2019年Lechuga等[21]发现在肺癌细胞H1573中敲低ADD3可以促进肺癌细胞的迁移潜能;而2021年Wang等[20]发现ADD3可变剪接异构体在肺癌细胞系A549和H520中促进细胞迁移和生长,两项研究结果相反,原因可能是ADD3在不同细胞系中发挥的功能不同。因此本研究继续检测了ADD3可变剪接异构体在不同肺腺癌细胞系中的表达情况,发现ADD3可变剪接异构体在不同肺腺癌细胞中差异较大,这也许可以解释ADD3在不同细胞系中对细胞迁移能力具有双重影响的现象。本研究又通过肺腺癌Kaplan网站分析了这3种基因与肺腺癌预后的相关性,发现3种基因在肺腺癌中呈低表达,且低表达者预后不良,以上分析结果进一步证实了ADD3、NUMB及LIMCH1可能抑制肺腺癌进展的推测。
肿瘤细胞可以修饰其表面抗原,从而改变肿瘤组织的微环境,逃避免疫系统的监视、识别和攻击,并继续生长和增殖。肿瘤突变负荷与免疫检查点阻断反应之间存在正相关。来自体细胞突变的肿瘤新抗原激活适应性免疫反应,杀死癌细胞。同样,具有新抗原生成能力的肿瘤相关剪接事件被认为是免疫治疗反应的重要预测因子[22-23]。越来越多的临床研究[24-26]表明,针对癌症种系抗原的T细胞受体工程化T(T-cell receptor engineered T,TCR-T)细胞治疗和基于新抗原的疫苗都具有显著效果。肿瘤特异性新抗原可以由编码突变衍生,也可以由其他过程产生,如剪接事件的改变。一些分析高通量测序数据的研究[27-29]显示,肿瘤特异性选择性剪接非常丰富,并可能产生有助于表位库的新表位。研究[30]表明,剪接障碍直接影响在免疫途径中发挥关键作用的基因,从而影响癌症免疫治疗效果。识别不同剪接异构体在特定癌症中的作用和剪接因子的调节作用,对于揭示肿瘤逃避免疫反应机制具有重要意义。因此,本研究进一步利用TIMER网站分析了ADD3、LIMCH1及NUMB与肺腺癌中免疫细胞功能的相关性,结果显示,ADD3、NUMB与B细胞、T细胞、巨噬细胞、中性粒细胞及树突状细胞功能均呈正相关,而LIMCH1与巨噬细胞功能呈正相关,提示ADD3、NUMB及LIMCH1可能作为肺腺癌特异性新抗原,在肺腺癌中可能通过调节免疫细胞功能,使免疫系统功能总体呈激活状态,从而起到抑制肺腺癌进展的作用。
综上所述,本研究发现,在肺腺癌中ADD3、LIMCH1及NUMB为可变剪接调控因子KHSRP的下游关键差异剪接靶基因,且其在肺腺癌中低表达与患者预后不良有关;免疫相关性分析发现ADD3、LIMCH1及NUMB可能与免疫细胞功能增强相关。虽然以上结果还需进一步验证,但本研究有望为寻找有效的肺腺癌治疗靶点提供新的线索。
利益冲突声明:所有作者均声明不存在利益冲突。
[参考文献]
[1]BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424.
[2]TIAN C H, WANG Y, YU H P, et al. A gene expression map of shoot domains reveals regulatory mechanisms[J]. Nat Commun, 2019, 10(1): 141.
[3]LI M M, WU P D, YANG Z C, et al. miR-193a-5p promotes pancreatic cancer cell metastasis through SRSF6-mediated alternative splicing of OGDHL and ECM1[J]. Am J Cancer Res, 2020, 10(1): 38-59.
[4]JBARA A, LIN K T, STOSSEL C, et al. RBFOX2 modulates a metastatic signature of alternative splicing in pancreatic cancer[J]. Nature, 2023, 617(7959): 147-153.
[5] PELLARIN I, DALL’ACQUA A, GAMBELLI A, et al. Splicing factor proline- and glutamine-rich (SFPQ) protein regulates platinum response in ovarian cancer-modulating SRSF2 activity[J]. Oncogene, 2020, 39(22): 4390-4403.
[6] HE Y T, OUYANG Z B, LIU W W, et al. TARDBP promotes ovarian cancer progression by altering vascular endothelial growth factor splicing[J]. Oncogene, 2023, 42(1): 49-61.
[7] HU X H, HARVEY S E, ZHENG R, et al. The RNA-binding protein AKAP8 suppresses tumor metastasis by antagonizing EMT-associated alternative splicing[J]. Nat Commun, 2020, 11(1): 486.
[8] YADAV P, PANDEY A, KAKANI P, et al. Hypoxia-induced loss of SRSF2-dependent DNA methylation promotes CTCFmediated alternative splicing of VEGFA in breast cancer[J]. iScience, 2023, 26(6): 106804.
[9] JIA R B, CHAI P W, WANG S Z, et al. m6A modification suppresses ocular melanoma through modulating HINT2 mRNA translation[J]. Mol Cancer, 2019, 18(1): 161.
[10] LOBAS A A, SOLOVYEVA E M, LEVITSKY L I, et al. Identification of alternative splicing in proteomes of human melanoma cell lines without RNA sequencing data[J]. Int J Mol Sci, 2023, 24(3): 2466.
[11] SONG X, WAN X C, HUANG T Z, et al. SRSF3-regulated RNA alternative splicing promotes glioblastoma tumorigenicity by affecting multiple cellular processes[J]. Cancer Res, 2019, 79(20): 5288-5301.
[12] LARIONOVA T D, BASTOLA S, AKSININA T E, et al. Alternative RNA splicing modulates ribosomal composition and determines the spatial phenotype of glioblastoma cells[J]. Nat Cell Biol, 2022, 24(10): 1541-1557.
[13] KING P H, CHEN C Y. Role of KSRP in control of type Ⅰ interferon and cytokine expression[J]. J Interferon Cytokine Res, 2014, 34(4): 267-274.
[14] BRADLEY R K, ANCZUKÓW O. RNA splicing dysregulation and the hallmarks of cancer[J]. Nat Rev Cancer, 2023, 23(3): 135-155.
[15] AGRAWAL A A, YU L, SMITH P G, et al. Targeting splicing abnormalities in cancer[J]. Curr Opin Genet Dev, 2018, 48: 67-74.
[16] CLIMENTE-GONZÁLEZ H, PORTA-PARDO E, GODZIK A, et al. The functional impact of alternative splicing in cancer[J]. Cell Rep, 2017, 20(9): 2215-2226.
[17] YAN M X, SUN L, LI J, et al. RNA-binding protein KHSRP promotes tumor growth and metastasis in non-small cell lung cancer[J]. J Exp Clin Cancer Res, 2019, 38(1): 478.
[18] MIN H, TURCK C W, NIKOLIC J M, et al. A new regulatory protein, KSRP, mediates exon inclusion through an intronic splicing enhancer[J]. Genes Dev, 1997, 11(8): 1023-1036.
[19] BECHARA E G, SEBESTYÉN E, BERNARDIS I, et al. RBM5, 6, and 10 differentially regulate NUMB alternative splicing to control cancer cell proliferation[J]. Mol Cell, 2013, 52(5): 720-733.
[20] WANG J Z, FU X, FANG Z Y, et al. QKI-5 regulates the alternative splicing of cytoskeletal gene ADD3 in lung cancer[J]. J Mol Cell Biol, 2021, 13(5): 347-360.
[21] LECHUGA S, AMIN P H, WOLEN A R, et al. Adducins inhibit lung cancer cell migration through mechanisms involving regulation of cell-matrix adhesion and cadherin-11 expression
[J]. Biochim Biophys Acta Mol Cell Res, 2019, 1866(3): 395-408.
[22] WANG Y B, BAO Y F, ZHANG S R, et al. Splicing dysregulation in cancer: from mechanistic understanding to a new class of therapeutic targets[J]. Sci China Life Sci, 2020, 63(4): 469-484.
[23] REN P P, LU L Y, CAI S S, et al. Alternative splicing: a new cause and potential therapeutic target in autoimmune disease[J]. Front Immunol, 2021, 12: 713540.
[24] PAUCEK R D, BALTIMORE D, LI G D. The cellular immunotherapy revolution: arming the immune system for precision therapy[J]. Trends Immunol, 2019, 40(4): 292-309.
[25] LIU Y T, YAN X, ZHANG F, et al. TCR-T immunotherapy: the challenges and solutions[J]. Front Oncol, 2021, 11: 794183.
[26] SELLARS M C, WU C J, FRITSCH E F. Cancer vaccines: building a bridge over troubled waters[J]. Cell, 2022, 185(15): 2770-2788.
[27] JAYASINGHE R G, CAO S, GAO Q S, et al. Systematic analysis of splice-site-creating mutations in cancer[J]. Cell Rep, 2018, 23(1): 270-281.e3.
[28] KAHLES A, LEHMANN K V, TOUSSAINT N C, et al. Comprehensive analysis of alternative splicing across tumors from 8, 705 patients[J]. Cancer Cell, 2018, 34(2): 211-224. e6.
[29] BURBAGE M, ROCAÑÍN-ARJÓ A, BAUDON B, et al. Epigenetically controlled tumor antigens derived from splice junctions between exons and transposable elements[J]. Sci Immunol, 2023, 8(80): eabm6360.
[30] FRANKIW L, BALTIMORE D, LI G D. Alternative mRNA splicing in cancer immunotherapy[J]. Nat Rev Immunol, 2019, 19(11): 675-687.


