Genome Med:大规模长读长WGS队列分析揭示WES无法检测到的分子诊断结果
时间:2024-01-26 19:26:31 热度:37.1℃ 作者:网络
孟德尔遗传病是由单基因中的高影响变异所导致的,因此鉴定这些变异是一个重要的诊断步骤。NGS可以在缺乏正确临床诊断(即仅基于表型)的情况下,对候选变异进行外显子组或全基因组范围的检测。目前,短读长测序(WES、WGS)主导着临床诊断领域的研究与应用,但平均诊断率通常<50%。
相比于短读长WGS,长读长全基因组测序(lrWGS)有可能解决WES的技术限制。目前,市场上主要有两种lrWGS技术:Pacific Biosciences(PacBio)的单分子实时(SMRT)测序和Oxford Nanopore Technologies(ONT)的纳米孔测序。已有多项研究展示了lrWGS相对于短读长WGS和WES在诊断上具有附加价值,尤其是在具有挑战性的病例中。但评估lrWGS在对疑似孟德尔遗传病患者进行诊断时的附加价值方面仍需深入研究。
近日,来自沙特阿拉伯的研究团队对疑似常染色体隐性遗传病的患者进行了迄今为止规模最大的低深度lrWGS应用的探讨,分析导致WES未能进行基因诊断的各种因素,以及lrWGS作为一种反射性检测的应用。该研究表明,虽然lrWGS清楚地揭示了被WES遗漏的因果变异,但注释挑战仍然是WES无法诊断的一个重要原因。该研究结果发表Genome Medicine上,文章题为“Beyond the exome: utility of long-read whole genome sequencing in exome-negative autosomal recessive diseases”的研究论文。

研究团队共招募了34个中东血统家庭,其中可能包括无法通过临床WES(基于短读长测序)进行分子诊断的常染色体隐性疾病患者。通过直接的临床评估和详细的病历回顾获得了详细的表型信息。根据表型或家族史,只有表型可能符合常染色体隐性病因的家族被纳入研究(图1)。为了确保高分子量基因组DNA,研究人员从至少一名患者建立了淋巴母细胞株(LCL)。LCL也被用作RNA的来源,通过RT-PCR和RT-qPCR适当地确认了非编码变异的影响。部分家庭还同意进行皮肤活检,以建立初级成纤维细胞株,也被用作RNA的来源。此外,研究人员还获得了患者参与者的同意,以发布可识别的临床图像。
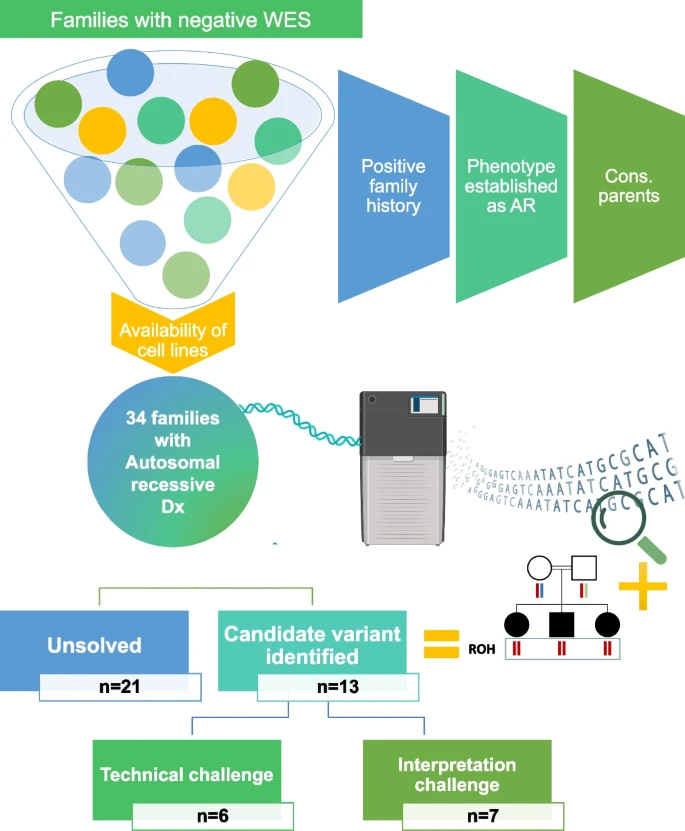
图1.研究设计示意图。
研究团队对每个家族的索引个体进行再分析,每个索引患者都接受了平均10倍深度的lrWGS检测。使用自体合子引导分析lrWGS数据,在34个家族中的13个(38%)中鉴定出候选变异。
lrWGS揭示WES无法检测到的分子诊断
1.TYMS作为致死性新生儿乳酸酸中毒的新候选基因
14DG1582是来自同一家庭的三名新生儿之一,他们在新生儿期死亡且经过生化确认严重乳酸中毒和疑似线粒体功能障碍。自合子引导的lrWGS分析揭示了TYMS基因中的一个同源结构变异(SV)。TYMS(MIM 188350)编码胸苷酸合成酶,是参与拯救dTTP途径的线粒体蛋白质,其缺乏可导致尿嘧啶插入错误和线粒体功能障碍。与对照组相比,RT-qPCR显示患者细胞中TYMS表达显著降低(图2)。经验证,该变异在一个受影响者中被证实为纯合子,在未显示任何先天性角化不良证据的父母中为杂合,该状态最近已与涉及TYMS的双基因遗传联系起来。与对照组相比,患者来源的成纤维细胞对已知的TYMS特异性抑制剂5-氟尿嘧啶(5-FU)表现出高度敏感。
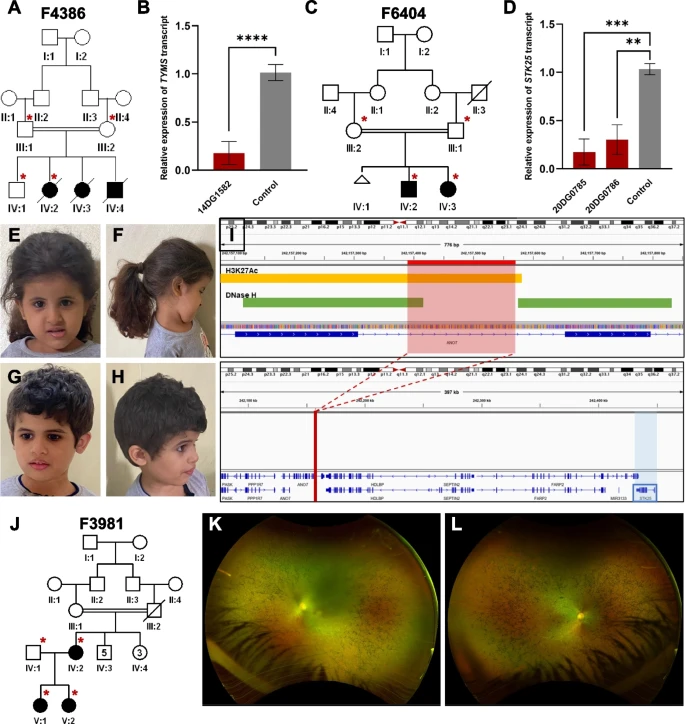
图2.WES未检测到的SV。
2.家族性带状角膜病的一种新调节元件变异
20DG0235和20DG0239是两个患有非综合征带状角膜病的兄妹(图3)。自合子引导的lrWGS分析揭示了SLC4A4基因的5'UTR上游有一个新型同源单核苷酸变异(SNV)。已知SLC4A4(MIM 603345)隐性变异可导致带状角膜病,作为多系统综合症的一部分,而这两个兄妹却没有。研究团队假设这可能是由特定的转录失调引起的可变表达。RT-qPCR数据显示,与对照相比,两兄妹的SLC4A4表达显著降低(图3C)。为了确认该变异的调控性质,研究人员设计了使用野生型和突变序列的分析。与对照相比,突变序列显示了45%-50%的转录活性降低。
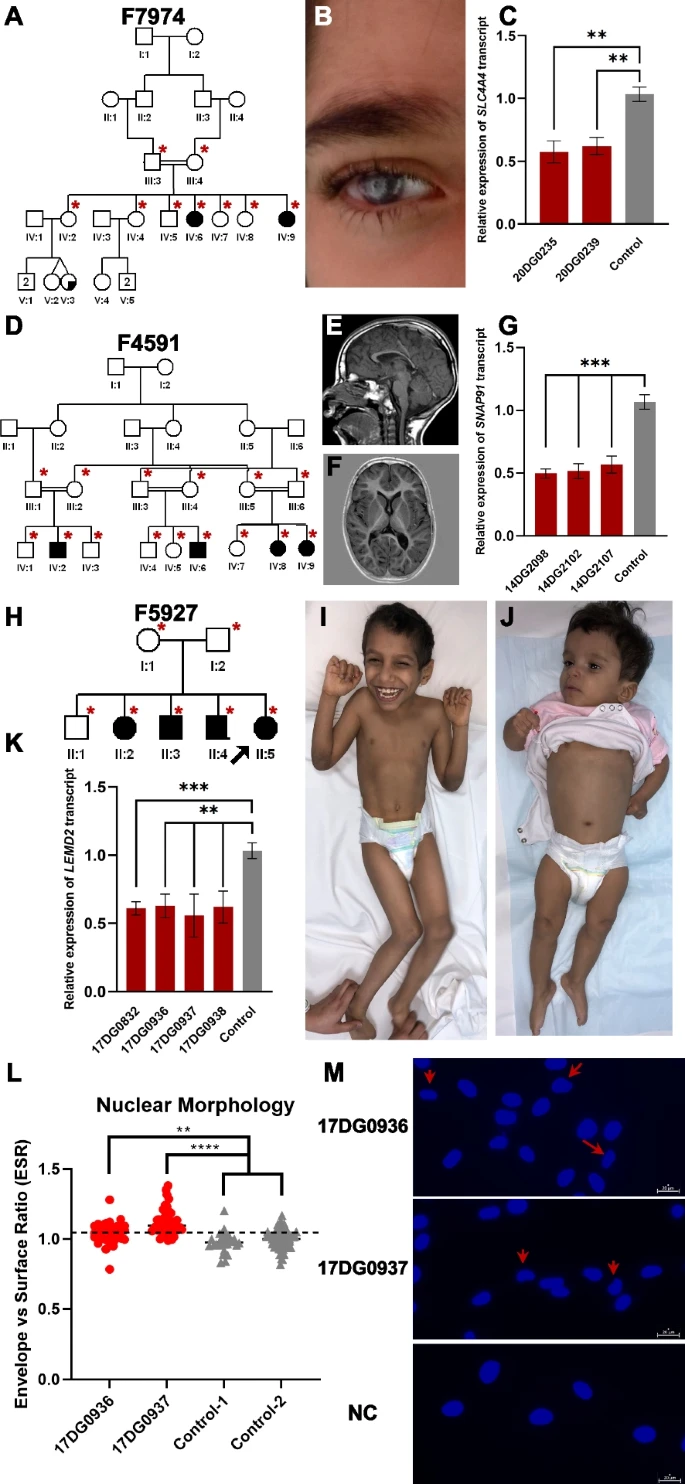
图3.WES未检测到的变异。
lrWGS突出WES的注释挑战
13DG1395和16DG0856是三个死产婴儿中的两个,表现为小头畸形、宫内生长迟缓、严重的颅面畸形伴有裂隙以及与Diamond-Blackfan综合症高度一致的严重骨骼畸形(图4)。自合子引导的lrWGS分析揭示了FLVCR1基因中的一个4 bp的纯合子缺失。这种经典的剪切变异也在WES中被发现,但被认为是一个偶然发现,因为它曾在一个患有成年起病共济失调和视网膜色素变性的家庭中报告过,这是OMIM中唯一列出的表型。RT-PCR实验表明该变异导致了一个剪切缺陷和蛋白质的早期截短。
图4.解决了具有注释挑战性变异的家庭。
上述结果显示,虽然为了节约成本而降低了lrWGS的深度,但仍然能够在38%的研究家庭中利用位置映射识别候选变异。虽然如此,研究团队对低深度策略在62%阴性家庭中的贡献感兴趣。研究团队选择F6440作为最有说服力的家庭进行进一步分析,因为表型映射到一个单一的位点,但lrWGS未能识别到任何候选变异。家庭F6440包括多个受影响的个体,共享的表型包括深刻的脑发育迟缓、外周痉挛、癫痫等。研究团队利用光学基因组映射来调查lrWGS未能检测到的可能存在的SV,发现了一个新的插入,破坏了CHMP5的阅读框架。研究人员认为CHMP5是这种新型神经发育障碍的有力候选基因。(图5)
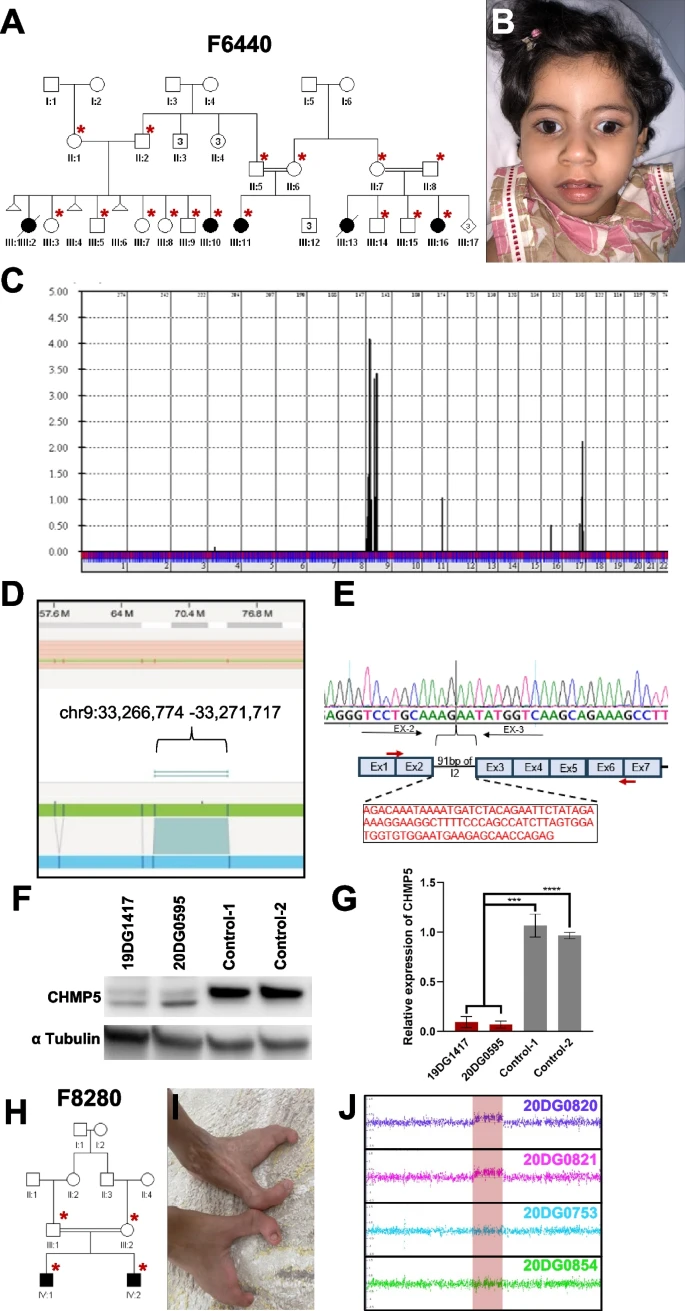
图5.lrWGS未能发现的遗传变异。
结 语
当WES无法进行基因诊断时,下一步应该做什么是重要的问题。该研究的目标是验证:当WES中的注释挑战得到充分解决并被排除时,应该使用lrWGS进行诊断检测。具体来说,这些病例富集了短读长WES不容易发现的变异类别,如SV、重复序列和非编码变异。
该研究展示了许多新的基因-疾病关系(新的候选基因和已确定基因的新表型),有待未来队列的证实。在34个家族的队列中,有13例(38%)发现了可能的致病变异。令人惊讶的是,研究团队在7个家族中发现了短读长WES可能已经识别的致病变异,后者突显了在注释层面尤其具有挑战性的情况。总的来说,lrWGS在检测SV方面展现出明显的优势,同时研究团队提出了在常染色体隐性疾病背景下实施lrWGS的方法,即利用位置映射使其效用最大化。
论文原文:
AlAbdi, L., Shamseldin, H.E., Khouj, E. et al. Beyond the exome: utility of long-read whole genome sequencing in exome-negative autosomal recessive diseases. Genome Med 15, 114 (2023). https://doi.org/10.1186/s13073-023-01270-8


