IVD技术丨RCA技术在蛋白原位检测及超灵敏免疫中的应用
时间:2024-05-31 23:02:01 热度:37.1℃ 作者:网络
RCA是以一小段环状寡核苷酸为模板,以dNTPs为原料,在DNA/RNA聚合酶作用下扩增产生一条长重复单链DNA/RNA,如下图所示。滚环扩增方法目前已经广泛应用于基因组学、蛋白组学、分子诊断、生物传感、药物研发等领域。线性扩增的扩增效率可以达到10^5倍,指数扩增的扩增效率可以达到10^9倍。
2006年Ulf Landegren在《Nature Methods》期刊发表的文章:Direct observation of individual endogenous protein complexes in situ by proximity ligation[1],主要介绍了邻近寡核苷酸探针两个相互作用靶蛋白抗体分别偶联,当抗体与靶蛋白结合时,可引导环状DNA链的形成,DNA环又作为滚环扩增(RCA)的模板,使相互作用的蛋白质分子对可以在人类细胞系和临床标本中可视化和计数。如果两个邻近探针彼此紧密结合,例如通过结合同一复合物中的两种蛋白,随后添加的线性连接寡核苷酸被引导形成通过酶促DNA连接共价连接的环状结构。连接后,使用其中一个邻近探针作为引物启动RCA。RCA产物通过与RCA产物中的标签序列互补的荧光标记寡核苷酸杂交检测。填充的实心圆圈代表非引物近端探针封闭的2`-O-methyl RNA 3` 末端。形成邻近连接反应的圆圈中的绿线在RCA产物(蓝色)中产生了多个互补序列拷贝。这种基序通过杂交荧光标记的检测寡核苷酸(绿色)来检测。探针设计原理图及相关的探针序列如下,寡核苷酸通过巯基修饰偶联到特定抗体上。
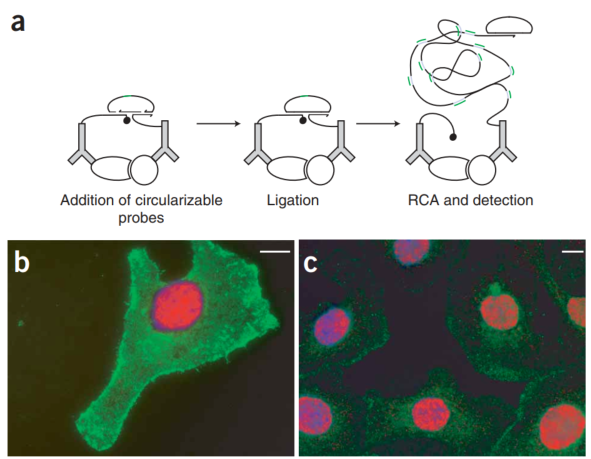
P-LISA在蛋白质原位检测中可以直接揭示正常细胞和组织中蛋白质之间的相互作用,而不会受到过表达或异位表达的影响。该方法有助于在基础研究和临床诊断中建立和使用全面的蛋白相互作用组图谱。
如果将两个相互作用的寡核苷酸结合到二抗上面:如来源于兔子的二抗偶联nonpriming proximity probe oligonucleotide,来源于小鼠的二抗偶联RCA primer proximity probe oligonucleotide,那么可形成通用性试剂盒检测两个抗体的相互作用。
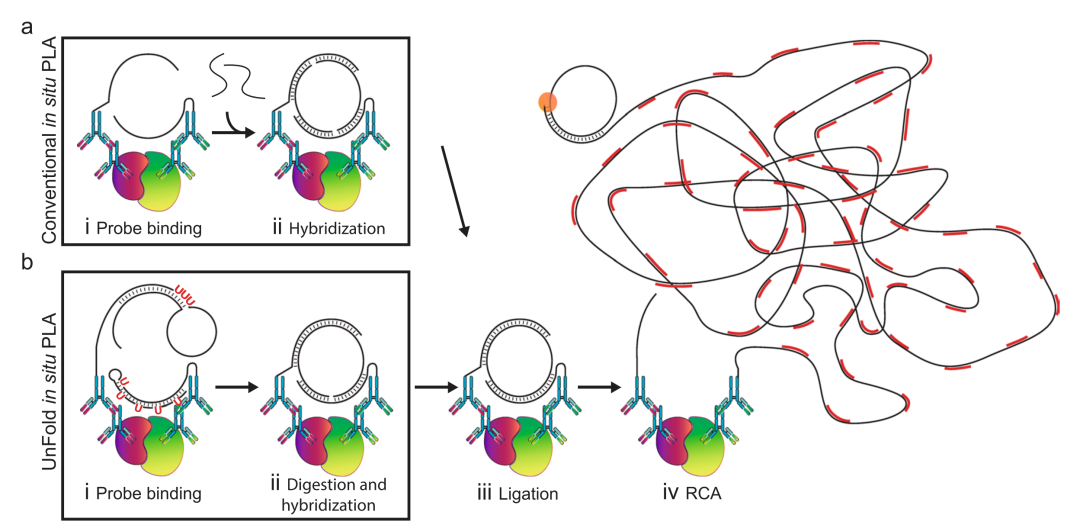
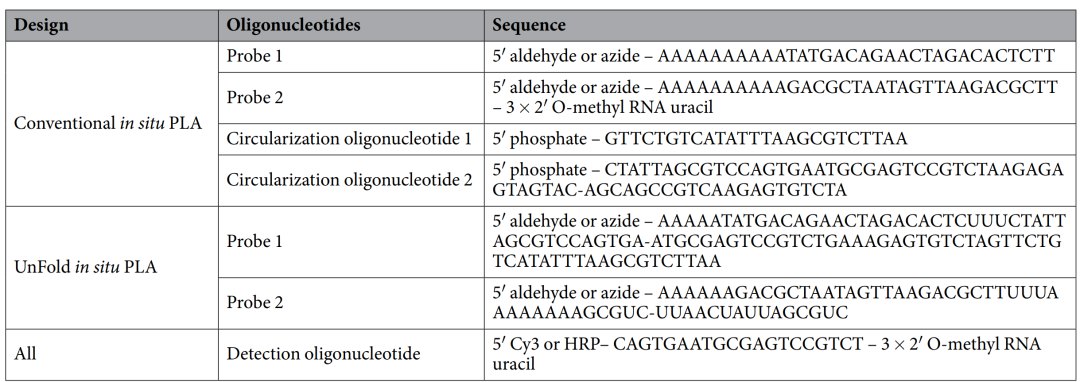
2018年Ulf Landegren在《Scientific Report》上发表的文章:Improved efficiency of in situ protein analysis by proximity ligation using UnFold probes[2]对上述文章的研究方案重新设计了用于原位邻近连接试验(PLA)的探针,使目标蛋白的定位检测更加高效。原位PLA依赖于一对抗体-寡核苷酸偶联物(PLA探针)对目标蛋白的识别,它们共同产生DNA环,模板定位的滚环扩增反应。对靶蛋白的双重识别需要通过忽略抗体之外的任何交叉反应来提高选择性,并且允许检测蛋白-蛋白相互作用和翻译后修饰。这篇文章中作者描述了PLA探针的一种改进设计——UnFold probe,其中形成环状DNA链所需的所有元素都被纳入探针中。通过在检测反应中包括酶促“去折叠”步骤来防止展开探针之间的过早相互作用。这使得只有在去除多余的试剂后,一对对试剂才能形成DNA环。探针设计原理图及相关的探针序列如下,寡核苷酸通过叠氮化修饰偶联到特定抗体上。
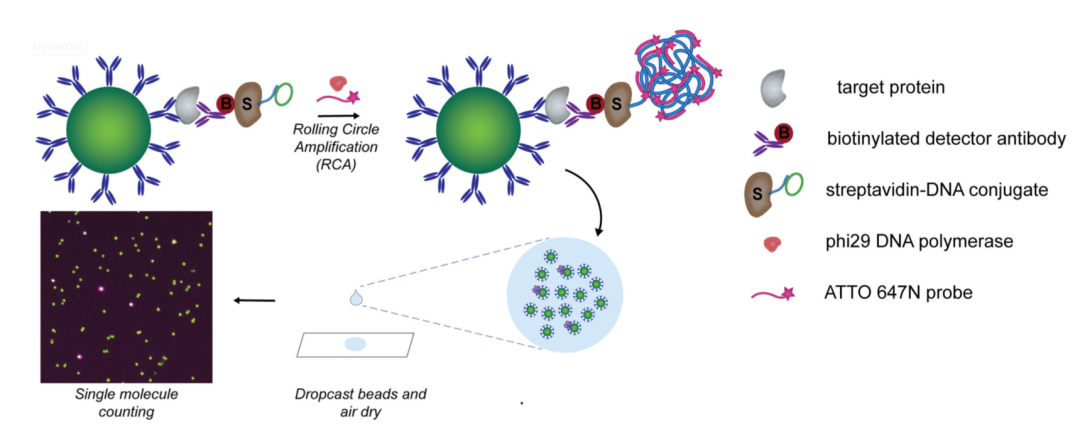
2020年,David R Walt在《J Am Chem Soc》期刊上文章讲述了研究团队开发的一种简单的单分子计数策略[3],dropcast单分子测定(dSimoa)。通过提高采样效率和更简单的工作流程,可以计数更多的目标分子。在这种方法中,珠子简单地滴投到显微镜载玻片上,干燥成单层膜,用于数字信号读出。将免疫检测与RCA(滚环扩增)技术相结合。在抗体包被的顺磁性微珠上形成单个免疫复合物三明治并用链霉亲和素-DNA 偶联物标记后,对微珠进行滚环扩增 (RCA) 以产生连接到每个免疫复合物的长连接子。在 RCA 期间,荧光标记的DNA探针与连接子杂交,以在携带完整免疫复合物三明治的珠子上产生局部荧光信号。RCA后,将珠子浓缩,滴投到显微镜载玻片上,并使其干燥以形成单层薄膜。通过液滴膜的荧光成像和计数“开”和“关”磁珠来计数单个靶分子。
2022年David R Walt在《ACS Nano》上发表的文章,讲述了一种新的单分子免疫检测方法MOSAIC,一种更灵敏、更精简的数字ELISA平台,即分子珠上信号扩增个体计数(Molecular On-bead Signal Amplification for Individual Counting, MOSAIC)[4],该平台的检测原子摩尔下限较低,灵敏度比其他方法提高了一个数量级。MOSAIC为稀有目标分子提供了高采样效率,分析珠数可以很容易地调整,以提高高测量精度的信号到背景。此外,与基于微孔或液滴的数字方法相比,基于溶液的MOSAIC信号读出扩展了可同时测量的分析物数量,以飞摩尔或更低的灵敏度进行多重检测。
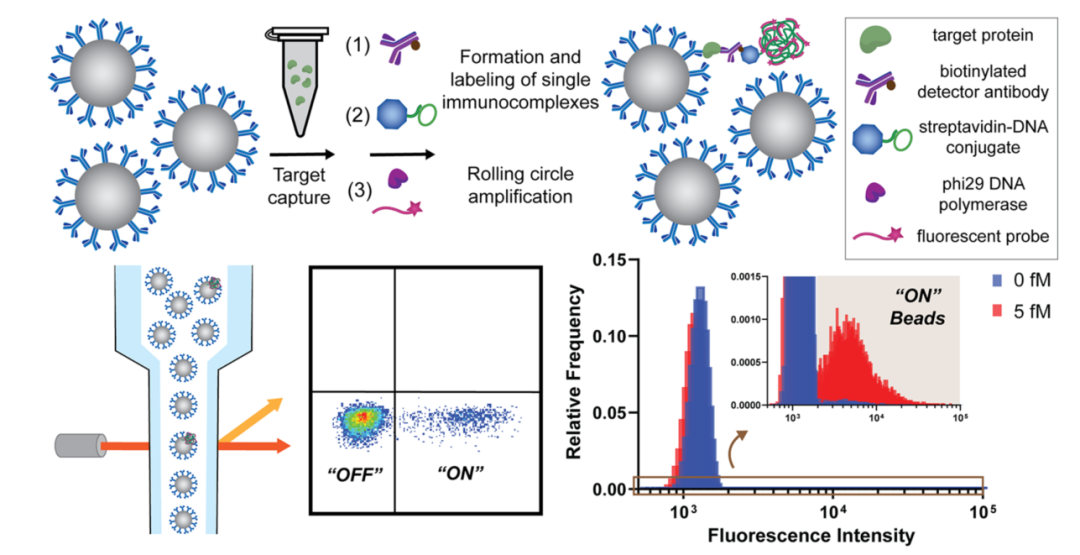
在2020和2022年的两篇文章中Dvid均采用RCA扩增的方式放大检测信号。所用序列信息如下,通过叠氮修饰将寡核苷酸酸与抗体偶联。

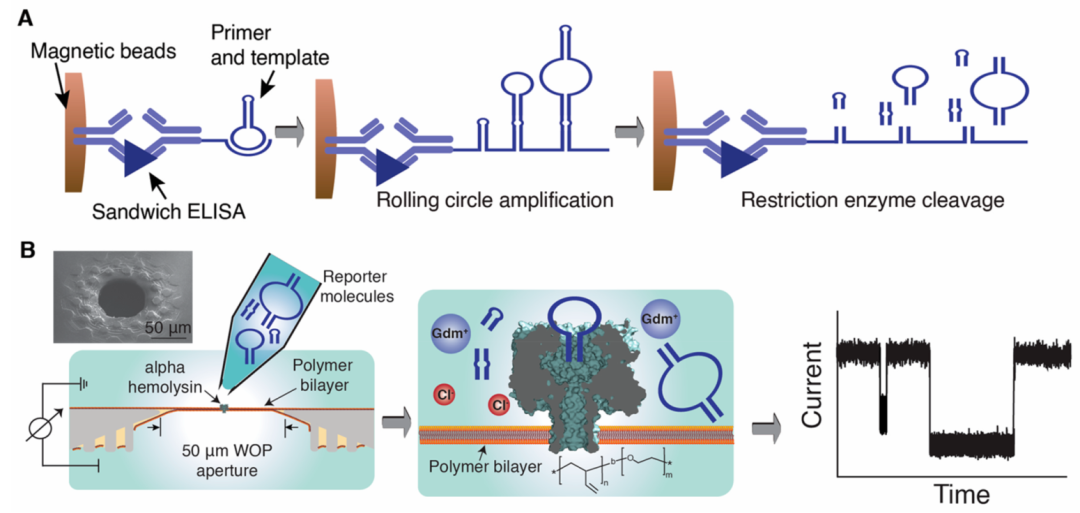
2023年David R Walt在《ACS Nano》期刊上发表的文章中介绍了一种新的纳米孔应用策略[5],该策略利用免疫捕获、等温滚圈扩增和产品的序列特异性片段来释放多个DNA报告分子用于纳米孔检测。这些DNA片段报告器产生一组纳米孔信号,形成独特的指纹或簇。因此,这种指纹签名允许生物标记物的鉴定和定量分析。
免疫PCR技术需要将寡核苷酸序列与检测抗体或者是二抗偶联,在偶联实验中会发现偶联剂的选择、功能基团的选择、缓冲体系条件及使用浓度等对偶联结果产生巨大影响,严重阻碍免疫分子的检测。未解决该技术问题,客服技术难点,杭州乐为科技有限公司在尝试过多种偶联剂、多种功能基团的等等方面,最终确定了高效、简便的偶联方案,利于纯化及下游免疫结合及PCR分析。
参考文献:
[1] Söderberg O, Gullberg M, Jarvius M, Ridderstråle K, Leuchowius KJ, Jarvius J, Wester K, Hydbring P, Bahram F, Larsson LG, Landegren U. Direct observation of individual endogenous protein complexes in situ by proximity ligation. Nat Methods. 2006 Dec;3(12):995-1000. doi: 10.1038/nmeth947. Epub 2006 Oct 29. PMID: 17072308.
[2] Klaesson A, Grannas K, Ebai T, Heldin J, Koos B, Leino M, Raykova D, Oelrich J, Arngården L, Söderberg O, Landegren U. Improved efficiency of in situ protein analysis by proximity ligation using UnFold probes. Sci Rep. 2018 Mar 29;8(1):5400. doi: 10.1038/s41598-018-23582-1. PMID: 29599435; PMCID: PMC5876389.
[3] Wu C, Garden PM, Walt DR. Ultrasensitive Detection of Attomolar Protein Concentrations by Dropcast Single Molecule Assays. J Am Chem Soc. 2020 Jul 15;142(28):12314-12323. doi: 10.1021/jacs.0c04331. Epub 2020 Jun 30. PMID: 32602703; PMCID: PMC7368998.
[4] Wu C, Dougan TJ, Walt DR. High-Throughput, High-Multiplex Digital Protein Detection with Attomolar Sensitivity. ACS Nano. 2022 Jan 25;16(1):1025-1035. doi: 10.1021/acsnano.1c08675. Epub 2022 Jan 14. PMID: 35029381; PMCID: PMC9499451.
[5] Kang X, Wu C, Alibakhshi MA, Liu X, Yu L, Walt DR, Wanunu M. Nanopore-Based Fingerprint Immunoassay Based on Rolling Circle Amplification and DNA Fragmentation. ACS Nano. 2023 Mar 28;17(6):5412-5420. doi: 10.1021/acsnano.2c09889. Epub 2023 Mar 6. PMID: 36877993; PMCID: PMC10629239.


