Int J Mol Sci:基于ML方法和分子对接的HCV早期诊断标志物稳健检测混合方法
时间:2023-05-08 15:27:20 热度:37.1℃ 作者:网络

背景:丙型肝炎是一种多因素疾病,据报道,截至2020年1月1日,全球丙型肝炎病毒(HCV)感染人数为5680万(95%不确定区间(UI)为55.2–67.8)。尽管这一数字低于2015年,但基于235个国家和地区研究的预测表明,由于新冠肺炎,我们无法如期实现2030年的全球消除目标。最近开发的靶向病毒NS3蛋白酶和NS5A和NS5B聚合酶的直接作用抗病毒剂(DAAs)在治疗HCV患者方面非常有效。然而,由于缺乏HCV疫苗、耐药突变的可能性、DAA治愈的个体中严重肝病的进展以及其他新出现的问题,全球根除HCV仍然很复杂。在HCV感染中维持肝脏损伤的致病机制甚至可能是其他肝脏疾病的基础,因此潜在地成为新的有效早期诊断标记和/或治疗靶点,甚至在未感染的患者中。值得注意的是,基因组表达改变发生在肝病进展的组织学证据之前,这表明在感染的急性期发生的事件影响患者的预后。此外,组织学评估可能存在偏差,病理学家对肝脏结构异常的解释也不尽相同。因此,确定能够早期和精确检测肝损伤的准确生物标志物至关重要。尽管宿主基因表达失调,但越来越多的证据表明,微小核糖核酸(miRNAs)在丙型肝炎病毒和宿主细胞之间的生物相互作用中起着至关重要的作用,这可能被宿主细胞用来控制病毒感染。所有这些研究揭示了miRNAs在病毒感染和疾病进展中不可或缺的作用。微小RNA是小的非编码RNA,它们非精确地互补,并与靶mRNA的30个非翻译区结合,导致mRNA失调。越来越多的证据表明,miRNAs是病毒和宿主相互作用网络中的核心因素之一。然而,用于疾病治疗的miRNA的表达迄今为止一直是一项困难的任务,因为它们在不同情况下具有可变的性质,但是在不同条件下差异表达的相同性质允许miRNA被用作疾病的生物标志物。一些特殊的miRNAs与肝脏特异性病理的进展高度相关,并且miRNAs水平的改变甚至比常规蛋白质更敏感和特异。因此,它们中的一些可以作为HCV感染的肝病患者的新的、侵入性较小的诊断和预后生物标志物。此外,它们是开发新的抗丙型肝炎病毒药物的有希望的治疗靶点。
目的:本研究的第一个目的是评估miRNAs的表达是否与丙型肝炎病毒的不同功能状态相关,以确定一个非侵入性的诊断生物标志物。因此,采用RT-qPCR方法检测了42例不同功能状态的丙型肝炎病毒肝组织和23例正常肝组织中188miRNAs的表达。这些数据被进一步应用于筛选在丙型肝炎病毒和正常肝组织中差异表达的miRNAs(DEmiRNAs)。其次,为了找出可能作为药物靶点的关键基因,利用miRNet预测了DEmiRNA的潜在靶基因。为了进一步验证靶基因,我们利用GEO数据库下载了GSE34798-丙型肝炎病毒mRNA表达数据集。然后,在数据集上进行了五种集成机器学习算法(随机森林算法、Adboost算法、Bging算法、Boosting算法和XGBoost算法)。在对模型进行评估后,根据最优模型选择重要的特征,并利用V-Enny图筛选有效的目的基因。为了寻找HUB靶基因,在获得PPI网络后,使用MCODE(Cytoscape Plug-in分子复合体检测)和cell Hubba Plug-in筛选HUB靶基因。为了探索可能击中HUB靶基因的化合物,下载了18个具有抗肝纤维化作用的小分子的结构,并使用AutoDock工具1.5.6进行分子自动对接。
方法:应用RT-qPCR方法检测42例不同功能状态的丙型肝炎病毒肝组织和23例正常肝组织中188miRNAs的表达。筛选出差异表达的miRNA(DEmiRNAs),对目的基因进行预测。为了验证目标基因,对一个丙型肝炎病毒基因芯片数据集进行了五种机器学习算法(随机森林算法、Adboost算法、Bging算法、Boosting算法、XGBoost算法),然后根据最优模型选择重要特征。在鉴定了HUB靶基因后,为了评估可能击中关键HUB靶基因的化合物的效力,进行了分子对接。
结果:根据我们的数据,8个DEmiRNAs与早期阶段有关,8个DEmiRNAs与肝功能恶化和丙型肝炎病毒严重程度增加有关。在目标基因的验证阶段,模型评估显示XGBoost(AuC=0.978)优于其他机器学习算法。最大团中心性算法的结果确定CDK1是一个HUB靶基因,hsa-miR-335、hsa-miR-140、hsa-miR-152和hsa-miR-195可以提示CDK1是一个HUB靶基因。由于病毒蛋白促进CDK1的激活以促进细胞有丝分裂,药物抑制可能具有抗丙型肝炎病毒的治疗前景。分子对接实验结果表明,芍药苷(−6.32kcal/m ol)和山药苷(−6.01kcal/m ol)与CDK1有很强的亲和力,这可能是一种有吸引力的抗丙型肝炎病毒的化合物。
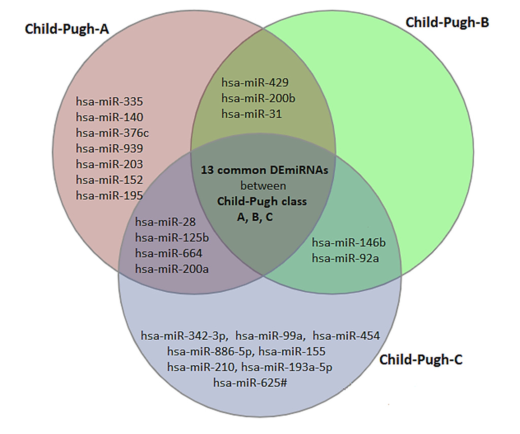
图1.V-ENN图显示,13个差异表达的miRNAs(DEmiRNAs)由Child-Pugh A、B和C类共享,可用作丙型肝炎诊断的生物标记物,但不是疾病分期。然而,该图还描绘了8个用于丙型肝炎早期诊断的特异性生物标志物,包括hsa-miR-335、hsa-miR-140、hsa-miR-376c、hsa-miR-939和8个DEmiRNAs,它们都可以作为C期功能的潜在生物标志物。
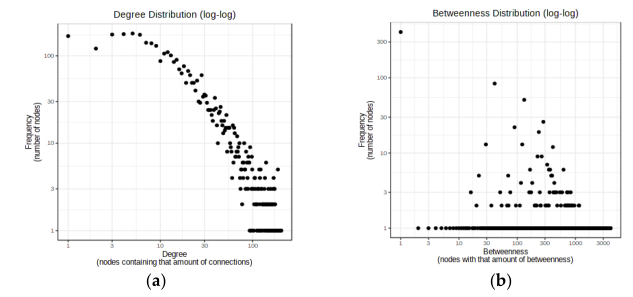
图2 (A)Child-Pugh A中下调的特定DEmiRNAs和预测的靶基因的程度分布图。该网络的直径为4,平均路径长度为2.26,其中hsa-mir-152(158)和hsa-mir-195(119)节点的连接数最多;(B)Child-Pugh C下调的特定DEmiRNAs与预测的靶基因的介数分布图。网络的直径为5,平均路径长度为2.01,HSA-MIR-155(0.907471357)和HSA-MIR-99A(0.6527755)具有较高的介数中心度和最短路径数,这表明了这些节点在网络中的关键作用
表1 不同机器学习模型预测结果的比较。
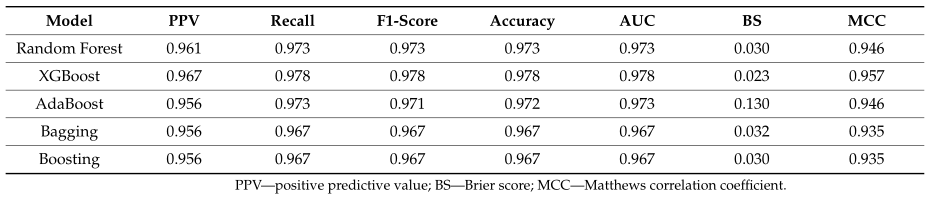
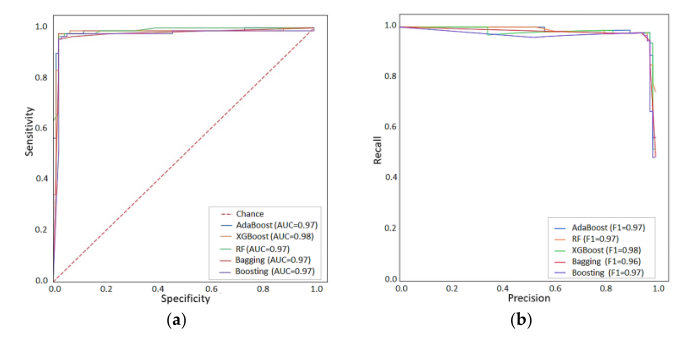
图3。XGBoost、AdaBoost、Boosting、Bagging和Random Forest的模型性能显示为ROC-AUC (a)和Precision-Recall曲线(b)。
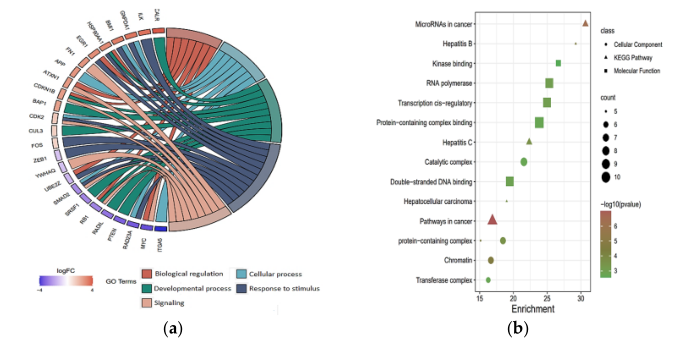
图4 关键目标基因的基因本体分析;(a)弦图说明了重要GO项和目标基因之间的关系,以及大多数目标基因参与对刺激的反应的生物过程;(b)散点图显示细胞成分、分子功能和KEGG富集分析
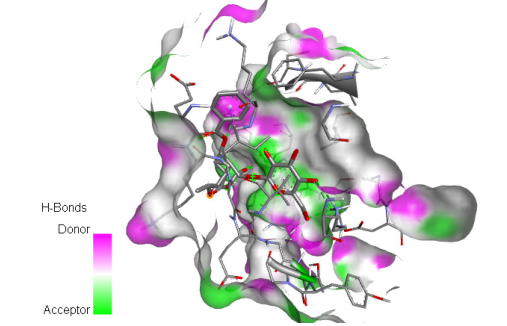
图5 芍药苷分子与CDK1蛋白上的六个氢键受体、两个疏水键(π-适马和π-烷基)对接,结合位点通过其表面可见。
结论:这项研究的发现可能在miRNA生物标记物的背景下为早期丙型肝炎诊断提供重要证据。此外,识别的HUB靶基因和具有高结合亲和力的小分子可能构成一组新的丙型肝炎病毒治疗靶点。
原文出处:Gholizadeh M, Łapczuk-Romańska J, Post M, et al.A Mixture Method for Robust Detection HCV Early Diagnosis Biomarker with ML Approach and Molecular Docking.Int J Mol Sci 2023 Apr 13;24(8)


