Nature:快速纳米孔测序+机器学习,手术期间实现中枢神经系统肿瘤的准确分类
时间:2023-11-03 17:43:14 热度:37.1℃ 作者:网络
中枢神经系统(CNS)肿瘤是最致命的癌症类型之一。CNS肿瘤的主要治疗手段包括对肿瘤进行神经外科切除,其中必须考虑的是在最大化切除范围与最小化神经损伤风险之间做出最佳选择。对不同种类CNS肿瘤的处理方式有很大不同,具有特定组蛋白H3(H3K27)突变的弥漫性中线胶质瘤被认为是无法治愈的,对这一类肿瘤的治疗方法是优先保障患者的生活质量,而不是完全切除;对于A型后颅窝室管膜瘤和非典型畸胎瘤样横纹肌样瘤,应遵循以大体全切除为目标的策略,这样获得较好的预后。
然而,外科医生在手术前对确切的肿瘤类型了解有限。目前的标准实践依赖于术前成像和术中组织学分析,但这些并不总是准确的,有时甚至是错误的。全基因组DNA甲基化模式的改变是肿瘤的显著特征,对DNA甲基化的评估可以揭示肿瘤起源和预后的信息。纳米孔测序可以直接检测甲基化谱并大幅减少样本制备时间。因此,可以在手术早期阶段对组织样本进行快速纳米孔测序,以便及时获得分子诊断,从而影响和制定神经外科手术策略。
近日,来自荷兰的研究团队发现基于低成本术中测序的机器学习诊断有助于神经外科决策。研究团队开发了一种与患者无关的转移学习神经网络Sturgeon,可用于CNS肿瘤的分子亚分类。将快速纳米孔测序与Sturgeon模型结合,在45个回顾性CNS肿瘤样本开始测序后40分钟内,Sturgeon给出了准确诊断。研究团队在25次手术过程中展示了该方法的实时适用性,实现了不到90分钟的诊断周转时间。相关研究结果发表在Nature上,文章题为“Ultra-fast deep-learned CNS tumour classification during surgery”。

文章发表在Nature上
01 通过模拟进行神经网络训练
鉴于CNS肿瘤甲基化谱的现有参考数据库大多建立在基于微阵列测序基础上,研究人员将已发表文献中包含来自CNS肿瘤和对照组织样本的2,801个参考标记甲基化谱作为参照模拟纳米孔测序。
“这项研究的创新之处之一是我们使用模拟来弥补这一差距,这样我们仍然可以从参考微阵列数据中学习,然后将其有效地应用于最新的纳米孔数据,”文章通讯作者、荷兰Oncode研究所J. de Ridder指出。
总体而言,在现有纳米孔测序读长和通量基础上,Sturgeon进行了3680万次模拟纳米孔测序运行的训练,并在另外420万次模拟纳米孔测序运行中进行了验证(图1)。模拟的纳米孔测序数据用于训练四个神经网络,这些神经网络均经过独立训练、验证和校准(图1b)。研究人员评估了Sturgeon子模型在保留测试集上的性能,在相当于测序40分钟时,Sturgeon子模型在所有类别中的F1得分为0.935(图1c)。此外,Sturgeon的性能与测序深度直接相关,在模拟测序的前50分钟内,性能提高最明显,覆盖了450K CpG位点的0.6%至4%。

图1. 模拟、交叉验证方法和模拟数据结果的示意图。
Sturgeon的训练数据集由不同年龄的患者组成(平均年龄29岁,36%年龄小于13岁)。为进一步验证Sturgeon在儿科环境中的表现,研究团队在接受CNS肿瘤切除手术的患者中获得了94个全基因组甲基化谱,进行模拟纳米孔测序实验后应用了Sturgeon模型。结果显示,对于明确诊断的病例,在短短25分钟的模拟测序中,Sturgeon以95.3%的正确率分类。在保守阈值为0.95时,86.2%的模拟样本被正确分类。Sturgeon与已有的分类模型(Heidelberg v11b4)表现相当。(图2)
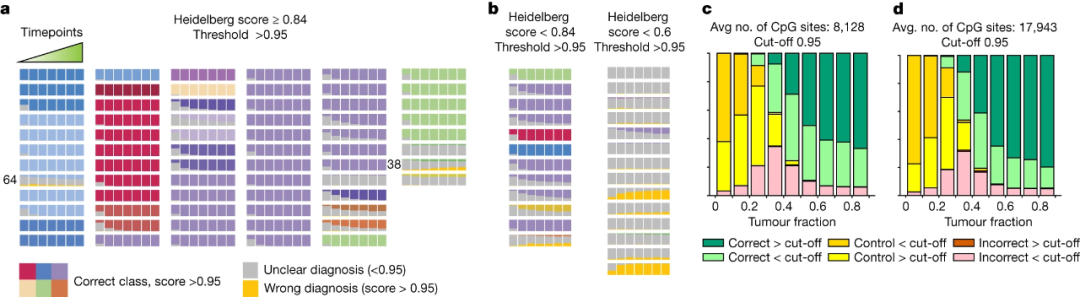
图2.儿科CNS肿瘤甲基化阵列模拟纳米孔运行的分类性能随时间的变化。
02 Sturgeon对纳米孔测序样本的分类
接下来,研究人员评估了Sturgeon在真实纳米孔测序数据上的性能。研究人员从荷兰马克西玛公主中心(PMC)生物库的27个儿童CNS肿瘤 DNA样本进行了回顾性测序和分类。应用Sturgeon来增加reads数量,模拟minION测序实验。分类结果表明,对于27个样本中的24个,在相当于25分钟的测序后,Sturgeon正确分类的分数高于0.95,平均而言,该阈值在测序的15-20分钟内可达到(图3a)。
为了进一步评估Sturgeon的稳健性及其对批次和操作偏差的敏感性,研究人员在公开可用的数据集(GSE209865)上对其进行了验证,该数据集由415次CNS肿瘤的纳米孔测序数据组成(图3c)。Sturgeon对383个(92.2%)样本进行了正确分类,32个(7.7%)被错误分类。与nanoDx相比,Sturgeon表现更好,并且对CpG位点覆盖与术中测序兼容更有信心。
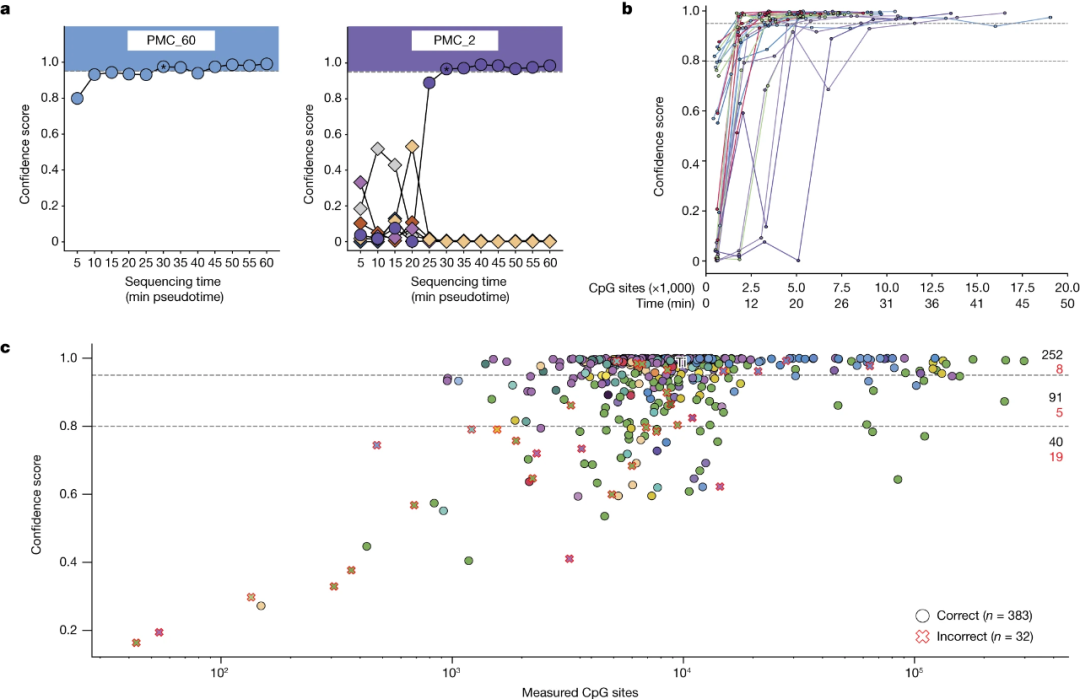
图3. 将Sturgeon应用于纳米孔测序样本。
03 将Sturgeon应用于术中测序
为证明Sturgeon在术中测序环境中的临床可行性,研究人员在荷兰两家不同医院的25次手术中执行了该方案,包括来自PMC手术的20个儿童样本和5个成人样本。手术中获得的组织样本一部分用于术中测序,另一部分用于组织学评估。为了快速获得高浓度的输入DNA,我们优化了DNA提取方案,在17-20分钟内从新鲜组织样本中提取DNA。结果显示,Sturgeon能够在最多45分钟的测序时间内对72%的肿瘤(18/25)进行亚类水平的正确分类。(图4)

图4. 术中测序周转时间。
结 语
该研究展示了术中甲基化纳米孔测序用于儿童和成人CNS肿瘤分类的实际可行性,并可用于改善手术决策。研究团队从现成的甲基化阵列数据中生成的模拟纳米孔测序数据上进行训练,开发了深度学习方法Sturgeon。Sturgeon与快速纳米孔测序结合,可以根据术中生成的序列数据快速准确地对肿瘤类型进行分类。
研究团队表示可以通过GitHub获得Sturgeon工具,行业合作伙伴至少有机会接受这一技术,并真正采取下一步措施,以确保它真正进入临床。
“这是一个范式转变,”华盛顿大学儿科遗传学家Danny Miller表示。“你可以想象在手术室中有这样一个设备,外科医生将样本交给技术人员,然后由技术人员加载设备,30到40分钟后,他们会得到某种肿瘤类型的答案。”
总之,该研究表明1.5小时的周转时间对于大多数样本来说是可行的。这与手术时间表完全兼容,以指导外科医生如何进行手术。
参考资料:
Vermeulen, C., Pagès-Gallego, M., Kester, L., Kranendonk, M. E. G., Wesseling, P., Verburg, N., ... & de Ridder, J. (2023). Ultra-fast deep-learned CNS tumour classification during surgery. Nature, 1-8.
https://www.nature.com/articles/s41586-023-06615-2


